数据集介绍
GEO数据库
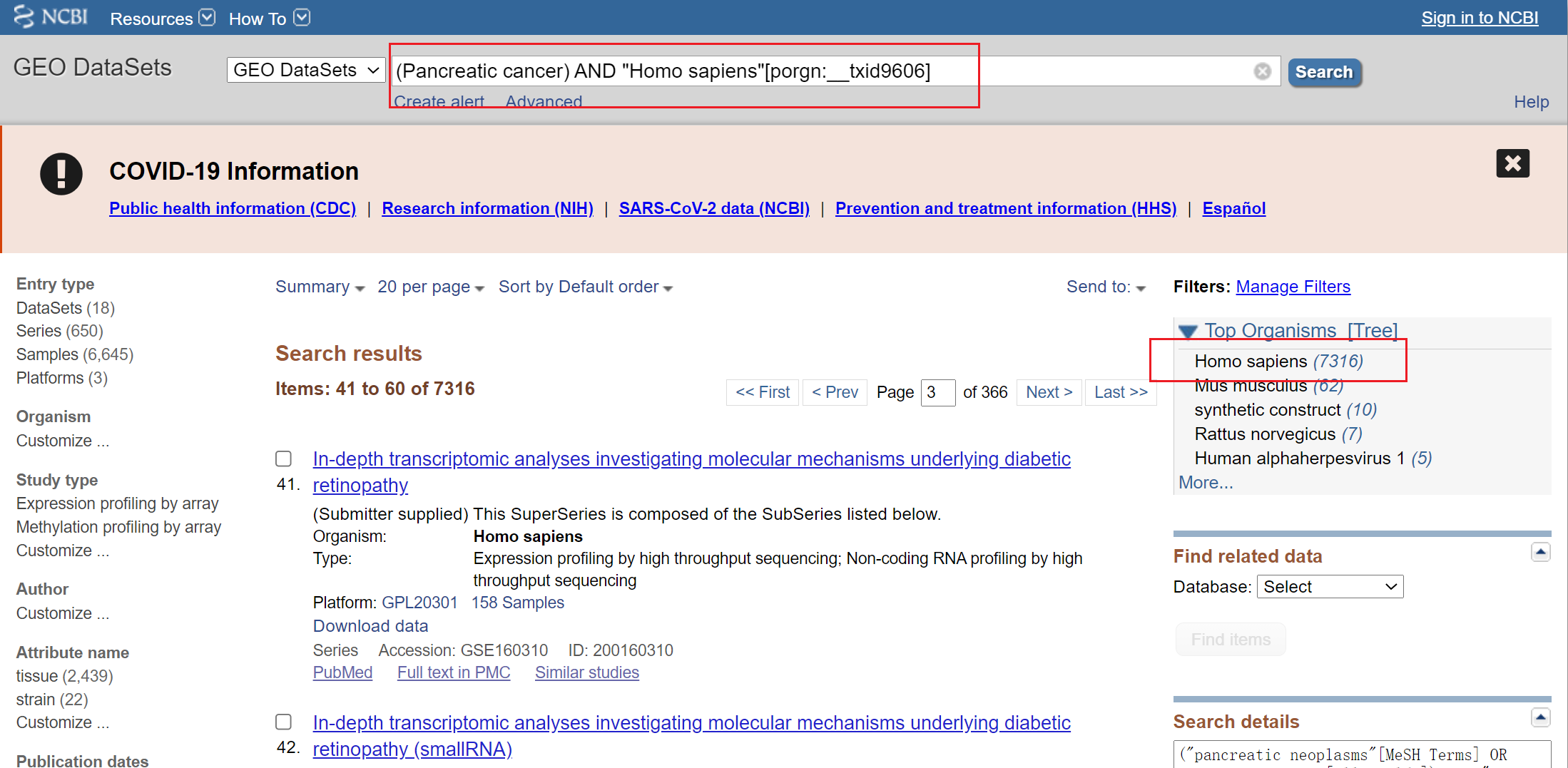
1、选择GEO DataSets:输入自己感兴趣的关键词。
2、选择Homo sapiens(人类研究)。
3、点击词条查看详细信息。
注:对于感兴趣的GSE,也可从文章中直接获取,然后直接用GSE号进行搜索。
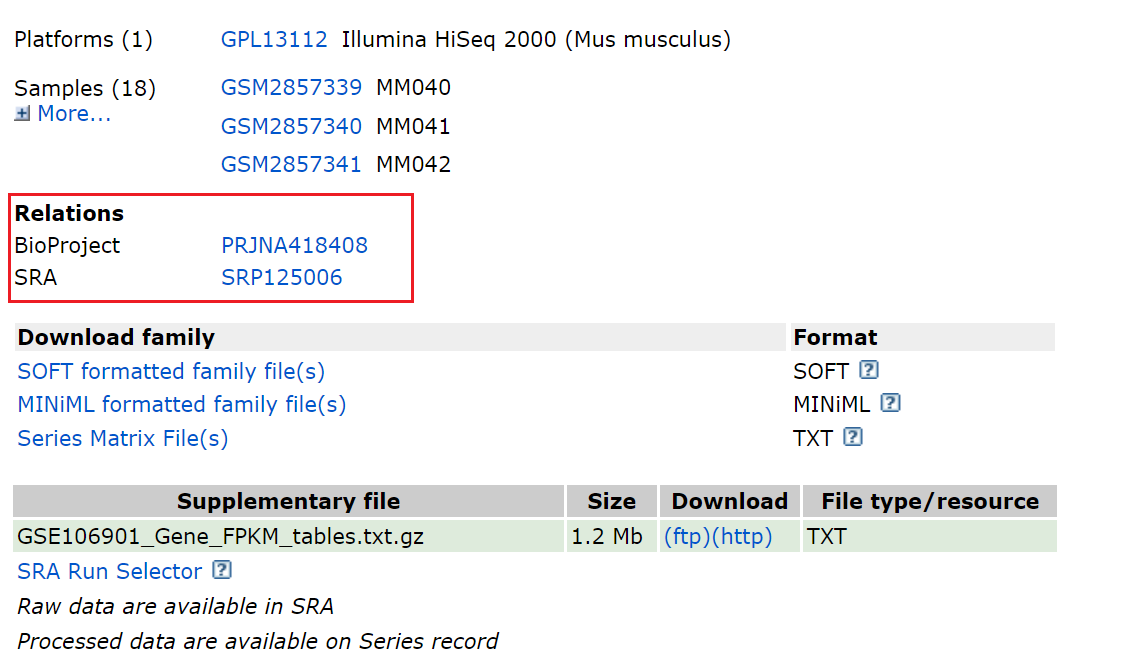
数据集信息
下载数据相关信息
ENA数据库
使用BioProject编号在ENA数据库中下载数据。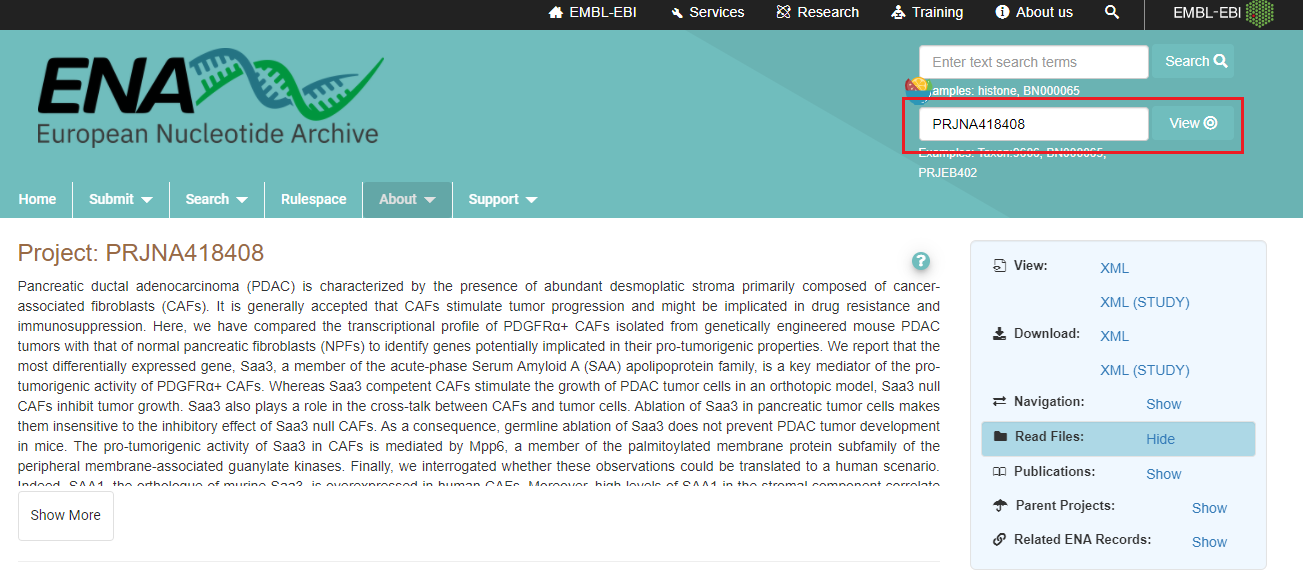
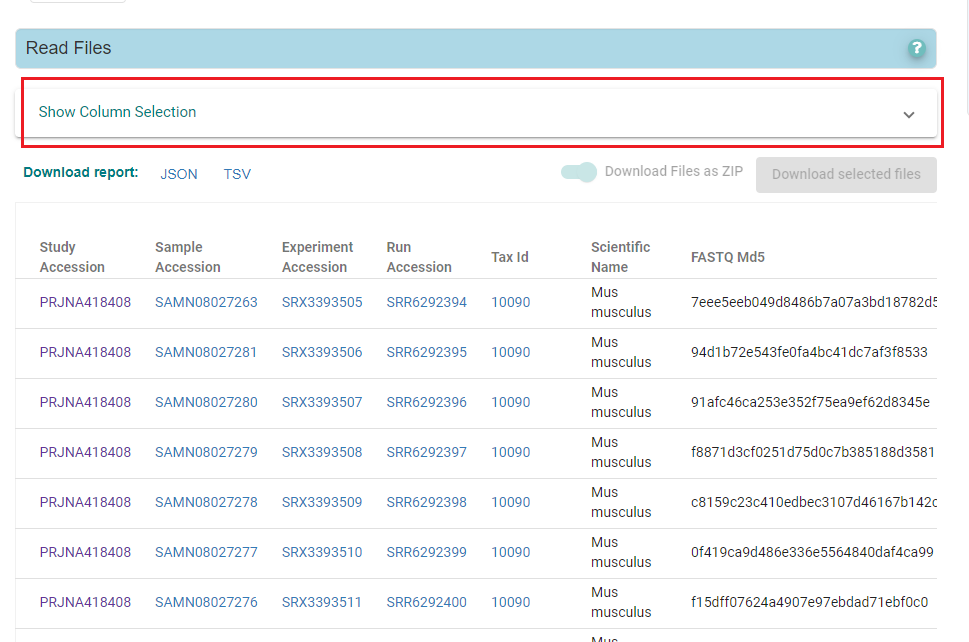
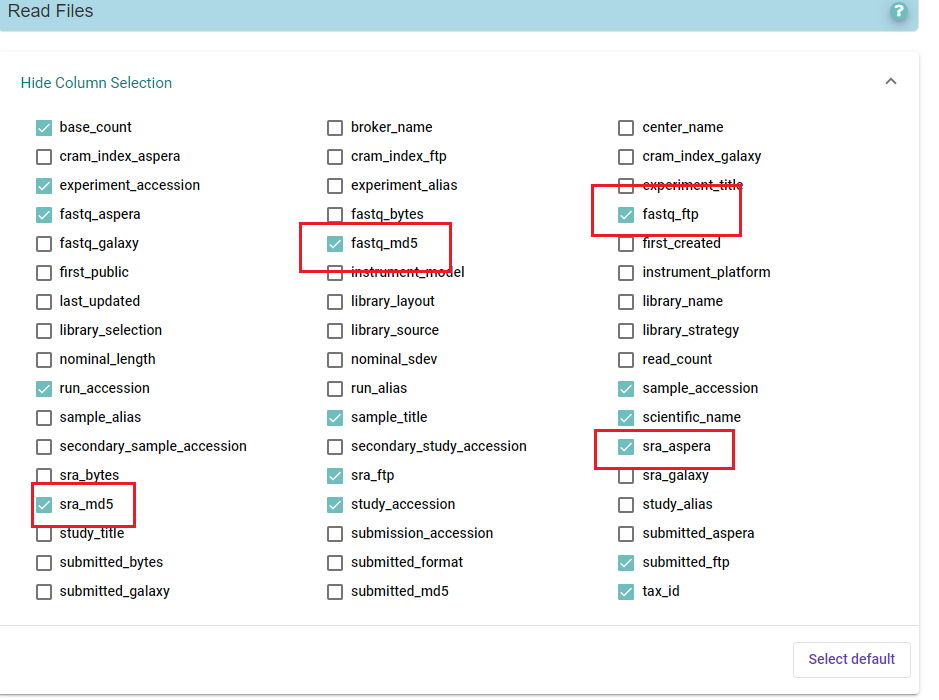
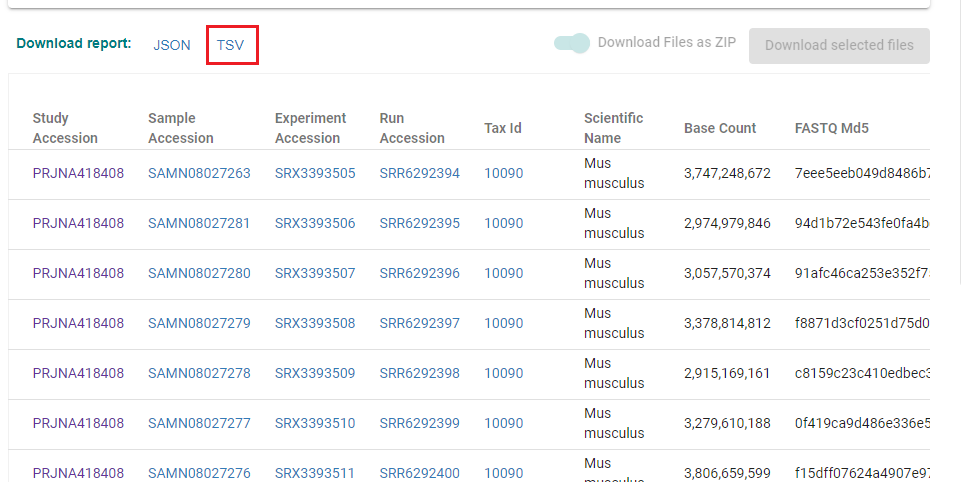
文件下载好之后通过ftp传到服务器中。
数据下载内容
使用aspera软件下载数据,需要获取ftp地址、aspera下载地址、md5值等。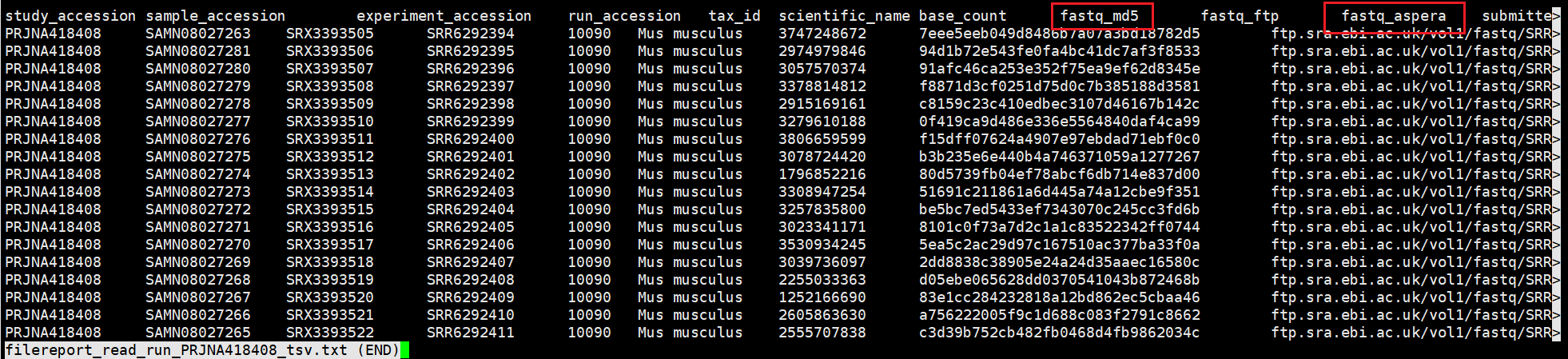
使用aspera软件下载
aspera软件常用参数
-Q ## 启用-Q或-QQ传输策略,使用-l和-m来设定目标和最低传输速度。-T ## 取消加密传输。若不添加此参数,可能会下载不了。-i ## 输入私钥,安装后有在目录 ~/.aspera/connect/etc/下有几个私钥,使用服务器用asperaweb_id_dsa.openssh文件作为私钥。-l ## 设置最大传输速度,比如设置为 200M 则表示最大传输速度为 200m/s。若不设置该参数,则一般可达到10m/s的速度-k ## 表示断点续传,通常设置为1即可--host ## ftp的host名,NCBI的为ftp-private.ncbi.nlm.nih.gov;EBI的为fasp.sra.ebi.ac.uk。--user ## 用户名,NCBI的为anonftp,EBI的为era-fasp。--mode ## 选择模式,上传为 send,下载为 recv。
单个样本下载
不常用。
## 激活小环境conda activate rna## 下载单个文件# fastq格式 注意下面参数链接的文件要换成自己目录的# 首先确定自己的密钥在哪里find ~/ -name '*.openssh'# 定义变量,注意等号两边不能有空格key_ssh=/home/t_rna/miniconda3/envs/rna/etc/asperaweb_id_dsa.opensshfq_url=fasp.sra.ebi.ac.uk:/vol1/fastq/SRR103/008/SRR1039508/SRR1039508_1.fastq.gz# 下载ascp -k 1 -QT -l 300m -P33001 -i $key_ssh era-fasp@$fq_url ./
批量样本下载
常用。
## 批量下载# 先查看自己需要的信息在哪一列head -n 1 filereport_read_run_PRJNA* |tr '\t' '\n' |cat -n# 得到fq.url文件,如果行尾存在特殊字符,运行 sed -i "s/\s*$//g" fq.url 去掉行尾特殊字符cat filereport_read_run_PRJNA229998_tsv.txt |awk -F '\t' 'NR>1 {print $20}' |tr ';' '\n' |grep '_' >fq.url# 下载三个样本测试head -n 4 fq.url >temp.url# 命令key_ssh=/home/t_rna/miniconda3/envs/rna/etc/asperaweb_id_dsa.opensshcat temp.url | while read iddoascp -k 1 -QT -l 300m -P33001 -i $key_ssh era-fasp@${id} ./done
数据完整性检验
MD5值检验
MD5,即”Message-Digest Algorithm 5(信息-摘要算法)”,其主要通过采集文件的信息摘要,以此进行计算并加密。
通过MD5算法进行加密,文件就可以获得一个唯一的MD5值,这个值是独一无二的,就像我们的指纹一样,因此我们就可以通过文件的MD5值来确定文件是否正确。
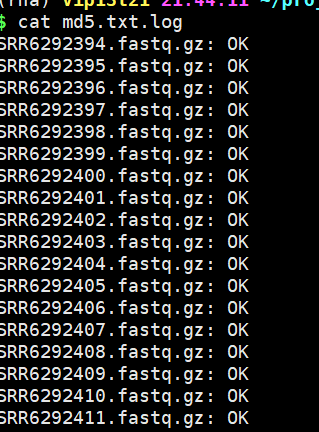
## 数据完整性检验(非常重要!!!)# 得到md5值,md5值与文件名之间为两个空格,检验格式要求cat filereport_read_run_PRJNA229998_tsv.txt|awk -F'\t' 'NR>1{print$8}' |tr ';' '\n' >md51cat filereport_read_run_PRJNA229998_tsv.txt|awk -F'\t' 'NR>1{print$8}' |tr ';' '\n' |awk -F'/' '{print$NF}' >md52# 将md51和md52合并在一起,中间为两个空格,md5格式要求paste -d' ' md51 md52 |grep '_' >md5.txt# md5值检验nohup md5sum -c md5.txt >check &
md5值检验OK。