galaxy 介绍
Galaxy 是由美国宾夕法尼亚州立大学(Penn State University)和约翰霍普金 斯大学(Jonns Hopkins University )联合开发的基于Web 的开源生物信息分析平台,目前在整个北美乃至全世界都有广泛的应用。 Galaxy 是一个开放性的平台,功能强大并支持二次开发,其集成了大量的生物信息分析工具,为用户提供了一个简单易用的生物信息分析界面。 通过Galaxy 提供的多种数据上传方式,用户可方便快速地上传数据,并通过浏览器选择所需的分析工具,设置分析参数之后即可提交数据分析请求。利用 Galaxy 中已安装的分析工具,用户还可创建和调用可重复使用的数据分析流程, 并对这些流程进行修改和导入导出。Galaxy 还具有历史记录功能,用户可查看自己所上传的所有数据以及执行过的分析工具和分析流程,并可直接从历史记录中创建数据分析流程。Galaxy 支持数据的可视化,内置多种图表功能,可绘制直方图,饼图,折线图等。对于已上传的数据,可视化结果和工作流,用户都可以设 置成共享状态分享给其他用户使用。除此之外Galaxy 还支持自定义工具的添加, 可按照需求扩展分析工具集。
视频学习
https://www.coursera.org/learn/galaxy-project
免费的公开课,但是缺少字幕,有爱心的小伙伴可以为视频提供翻译。我翻译了RNA-seq Analysis: Mapping 这一节。
安装
官方网址 https://galaxyproject.org/admin/get-galaxy/
环境要求
- UNIX / Linux或Mac OSX
- Python 2.7
克隆分支
因为 galaxy 是开源的的,所以直接 clone 分支就行
- 新分支
$ git clone -b release_19.01 https://github.com/galaxyproject/galaxy.git
- 如已经有本地克隆,更新分支
$ git fetch origin && git checkout release_19.01 && git pull --ff-only origin release_19.01
启动
只需要在文件下找到 run.sh 执行就行。第一次启动会下载安装很多依赖文件,需要很长时间,耐心等待。第二次启动可以使用nohup命令让其后台一直运行。
$ sh run.sh
or
$ nohup ./run.sh &
启动完成后,就可以在本机的浏览器上输入 http://localhost:8080/ 来使用。终止使用 Ctrl-C 或者 kill -9。如果没有启动的话,可能你在使用 conda 环境,请退出。
配置
先复制一份配置文件,然后在里边修改。
cp config/galaxy.yml.sample config/galaxy.ymlvim config/galaxy.yml
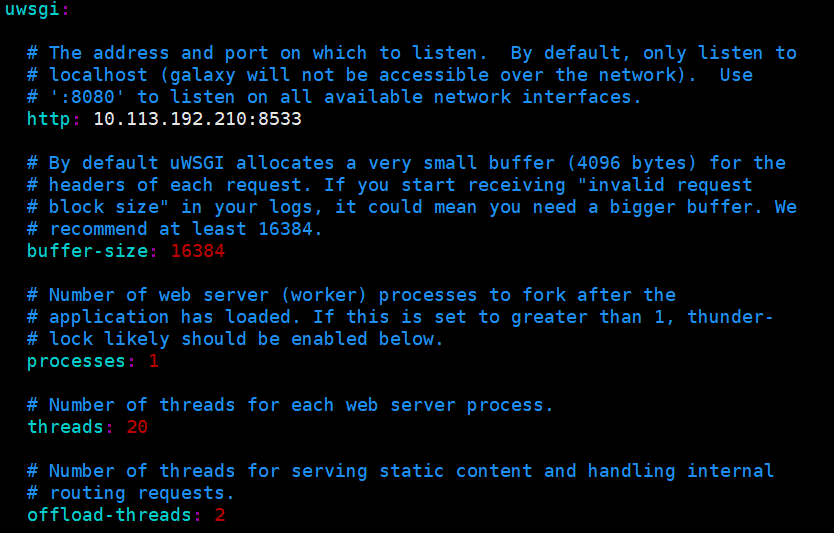
http: 即为作为服务器的主机的IP和端口,端口可以根据自己情况设,然后将端口放入防火墙白名单。就可以在其他电脑浏览器中输入ip+端口使用了 。其他配置参数酌情修改。
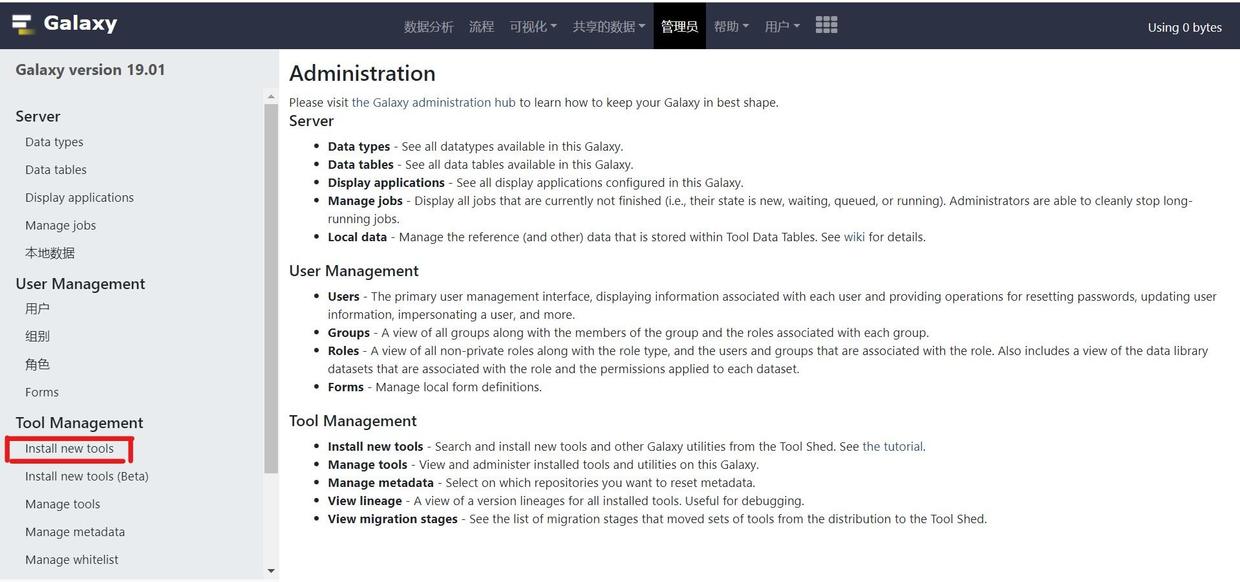
成为管理员
要通过 web 安装工具、管理用户等需要先成为管理员。在配置文件中加入作为管理员的用户名。然后通过web注册登录后就可以有管理员权限。
# this should be a comma-separated list of valid Galaxy usersadmin_users: user1@example.com,user2@example.com