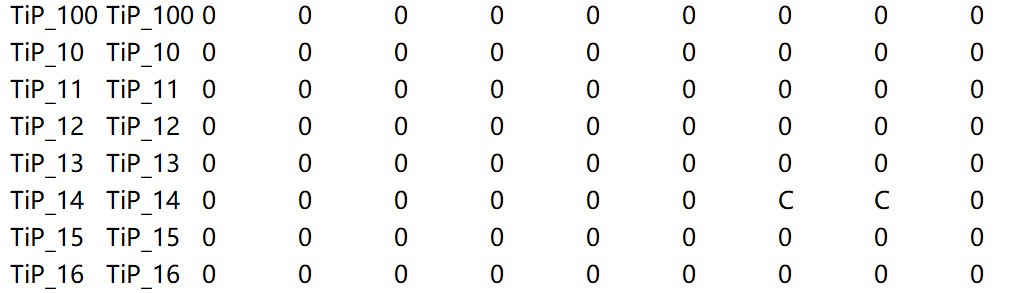使用工具 vcftools
vcftools --vcf SNP_merge.vcf --plink --out SNP
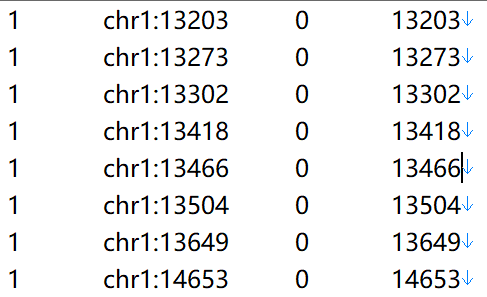
map文件 有四列,分别为
- 第一列:染色体编号为数字, 未知为0
- 第二列:SNP名称为字符或数字, 如果不重要, 可以从1编号, 注意要和ped文件SNP列一一对应
- 第三列:染色体的摩尔根距离(未知用0表示)
- 第四列:SNP物理坐标
ped 文件 数据没有行头, 空格或者tab隔开的文件。必须要有六列, 包括系谱信息, 表型信息。
Family ID (‘FID’)
Within-family ID (‘IID’; cannot be ‘0’)
Within-family ID of father (‘0’ if father isn’t in dataset)
Within-family ID of mother (‘0’ if mother isn’t in dataset)
Sex code (‘1’ = male, ‘2’ = female, ‘0’ = unknown)
Phenotype value (‘1’ = control, ‘2’ = case, ‘-9’/‘0’/non-numeric = missing data if case/control)
- 第一列: Family ID # 如果没有, 可以用个体ID代替
- 第二列: Individual ID # 个体ID编号
- 第三列: Paternal ID # 父本编号
- 第四列: Maternal ID # 母本编号
- 第五列: Sex (1=male; 2=female; other=unknown) # 性别, 如果未知, 用0表示
- 第六列: Phenotype # 表型数据, 如果未知, 用0表示
- 第七列以后: 为SNP分型数据, 可以是AT CG或11 12, 或者A T C G或1 1 2 2