利用conda 安装KOBAS
conda 的出现为软件安装提供很多便利,用户无需自己在编译安装软件,同时安装过程也会解决依赖。由于源码安装KOBAS比较麻烦,所以强烈推荐用conda安装:
# 创建环境conda create -n kobas# 激活环境conda activate kobas# 安装kobas,conda install -c bioconda kobas# 如果安装了mamba,可以改用mamba 安装, 安装速度有较大的提升。mamba install -c bioconda kobas# 安装完成之后,试运行,查看是否安装成功kobas-annotate -h
运行报错解决
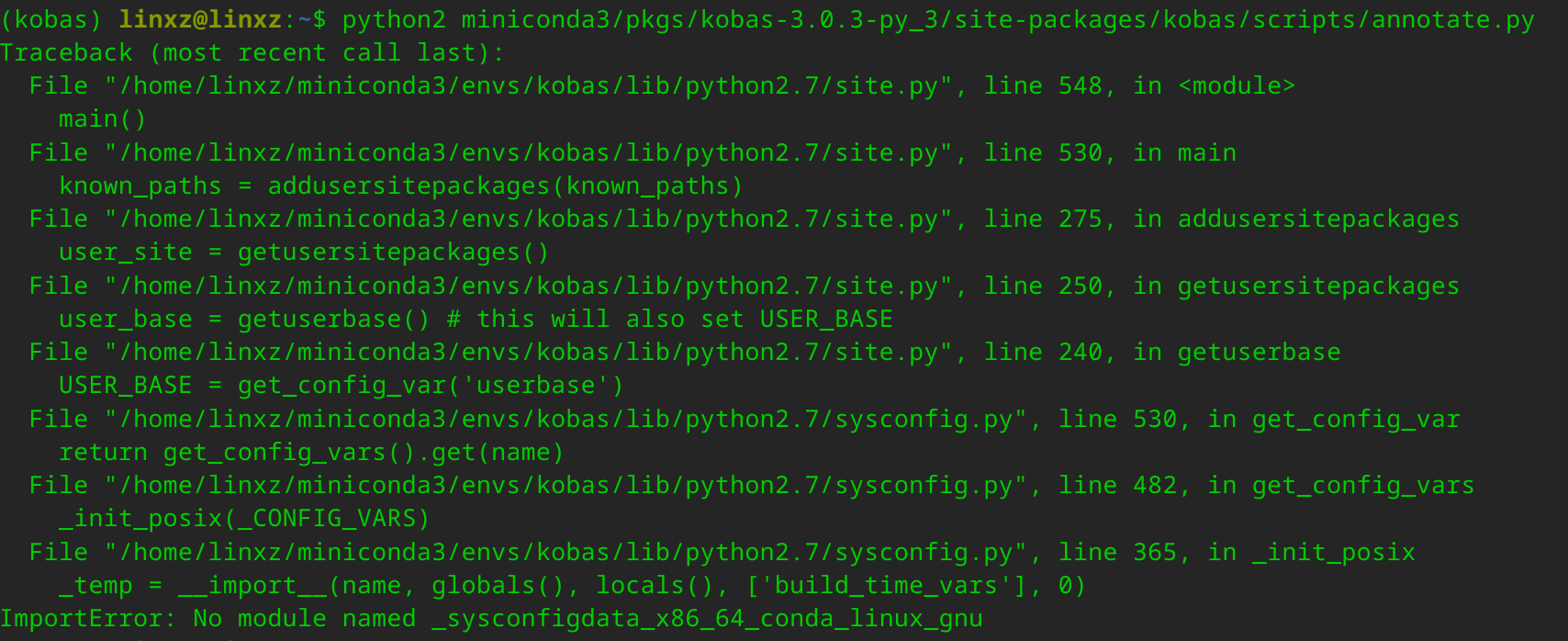
在UOS 20.1 系统利用conda 安转kobas后,退出环境再重新激活kobas环境,会报ImportError: No module named _sysconfigdata_x86_64_conda_linux_gnu 错误,百度了一下,原来因为这个模块的名字以下这个_sysconfigdata_x86_64_conda_cos6_linux_gnu,有的软件是依赖_sysconfigdata_x86_64_conda_cos6_linux_gnu,有的软件是依赖_sysconfigdata_x86_64_conda_linux_gnu ,解决办法也很简单找出来复制一下就可以解决这个报错,以下报错解决方法:
# 找出相关的包sudo find ~ -name _sysconfigdata_x86_64* | grep python2.7# 复制cp /home/linxz/miniconda3/envs/kobas/lib/python2.7/_sysconfigdata_x86_64_conda_cos6_linux_gnu.py /home/linxz/miniconda3/envs/kobas/lib/python2.7/_sysconfigdata_x86_64_conda_linux_gnu.py# 报错搞定
数据库下载
建议利用FileZilla等FTP下载软件,通过FTP进行数据下载。
KEGG注释
由于blast+ 本身比较慢,又有diamond 这个替代blast+的神器,所以注释的时候,建议利用diamond进行blast,然后将diamond 的结果给KOBAS的进行注释。如果是无参转录组(或者是有参转录组,但KOBAS不支持的物种或ID匹配不上),可以先用KOBAS进行注释然后进行KEGG富集分析(支持物种可以直接进行富集分析)。以下是注释代码:
# 物种所有基因进行注释# blastdiamond blastp -q TAIR/TAIR8_pep_20080412 -d diamond/ko.dmnd -o test/TAIR_pep.m8# 注释kobas-annotate -i test/TAIR_pep.m8 -t blastout:tab -s ko -o kegg.annotate.tmp.all -k KOBAS/# 关注基因进行注释或者从总的注释结果提取注释信息diamond blastp -q TAIR/test.fa -d diamond/ko.dmnd -o test/test.m8kobas-annotate -i test/test.m8 -t blastout:tab -s ko -o kegg.annotate.tmp.all -k KOBAS/
KEGG富集分析
# 富集分析kobas-identify -f kegg.annotate.tmp -o kegg.identify.tmp -p K -m h -b kegg.annotate.tmp.all -k KOBAS/
作图
自行百度相关代码。

