in Other Tools 21 小时前 346 浏览
随着单细胞 RNA 测序技术的迅速发展,有可能以高分辨率分解细胞类型的成分。为了识别稀有细胞类型,已经开发了许多方法。但是,现有方法仍然无法扩展到大型数据集,从而限制了其实用性。为了克服这一限制,达纳 - 法伯癌症研究所的研究人员开发了一种名为 GiniClust3 的新软件包,该软件包是 GiniClust2 的扩展,并且比以前的版本明显更快且内存效率更高。
使用 GiniClust3,只需大约 7 小时即可从包含一百万个细胞的数据集中识别常见和稀有细胞簇。细胞类型映射和扰动分析表明,GiniClust3 可以可靠地识别细胞簇。
分析拥有超过一百万个细胞的小鼠大脑数据集
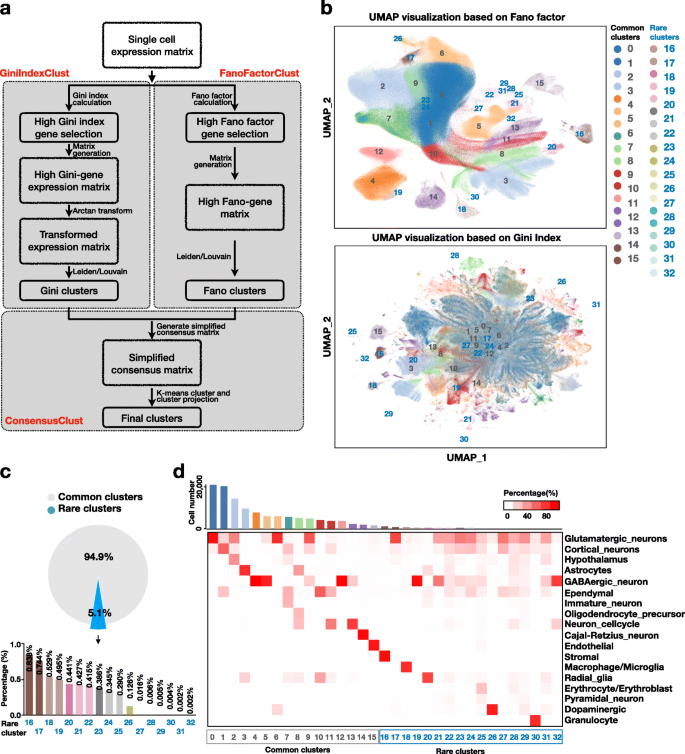
a GiniClust3 管道概述。输入单细胞表达矩阵基于分别由 Gini 索引(GiniIndexClust)和 Fano 因子(FanoFactorClust)选择的特征进行聚类。然后,使用可感知群集的加权共识群集算法(ConsensusClust)集成结果。b 分别 基于 Fano 因子(顶部)和 Gini 指数(底部)选定特征的基因表达模式的 UMAP 可视化。共识聚类结果用不同的颜色表示。c 整个群体中稀有细胞簇的比例。d 通过 scMCA 分析对常见和罕见簇的细胞类型作图的热图。顶部的条形图指示每个群集的单元号
综上所述,这些结果表明,GiniClust3 是识别常见和稀有细胞群体的强大工具,并且可以处理大型数据集。
可用性 – GiniCluster3 是在开源 python 包中实现的,可从https://github.com/rdong08/GiniClust3 获得。
董瑞,袁国强。(2020)GiniClust3:一种用于稀有细胞类型识别的快速且高效存储的工具。BMC 生物信息学 21(1):158。[ 文章 ]
标签:基尼系数 稀有细胞鉴定 可扩展性 单细胞 rna-seq
https://www.rna-seqblog.com/giniclust3-a-fast-and-memory-efficient-tool-for-rare-cell-type-identification/
https://www.rna-seqblog.com/giniclust3-a-fast-and-memory-efficient-tool-for-rare-cell-type-identification/