目前在使用深度学习进行医学图像处理,主要进行肺部结节检测,在预处理时经常会使用到SimpleITK软件包,它是一个很好用的读取医学图像信息软件包。
内容列表
SimpleITK
- SimpleITK.ReadImage(path)
- SimpleITK.GetArrayFromImage(itk_img)
- itk_img.GetOrigin()
- itk_img.GetSpacing()
- itk_img.GetDirection()
- 具体分析一下Origin与Spacing:
SimpleITK
Insight Segmentation and Registration Toolkit (ITK)是一个开源、跨平台的框架,可以提供给开发者增强功能的图像分析和处理套件。其中最为著名的就是SimpleITK,是一个简化版的、构建于ITK最顶层的模块。SimpleITK旨在易化图像处理流程和方法。
目前常用的代码有如下几种:
itk_img = SimpleITK.ReadImage(path)img_array = SimpleITK.GetArrayFromImage(itk_img)print("Img array: ", img_array.shape) #读取图像大小origin = numpy.array(itk_img.GetOrigin()) #读取图像原点坐标print("Origin (x,y,z): ", origin)direction = numpy.array(itk_img.GetDirection()) #读取图像方向print("Direction: ", direction)spacing = numpy.array(itk_img.GetSpacing()) # 读取图像尺度信息print("Spacing (x,y,z): ", spacing)//读取dicom文件file = sitk.ReadImage(filepath)获取基本信息,大小,像素间距,坐标原点,方向file.GetSize()file.GetOrigin()file.GetSpacing()file.GetDirection()读取dicom序列reader = sitk.ImageSeriesReader()reader.MetaDataDictionaryArrayUpdateOn()#这一步是加载公开的元信息reader.LoadPrivateTagsOn()#这一步是加载私有的元信息#根据文件夹获取序列ID,一个文件夹里面通常是一个病人的所有切片,会分为好几个序列series_IDs = sitk.ImageSeriesReader.GetGDCMSeriesIDs(directorypath)#选取其中一个序列ID,获得该序列的若干文件名dicom_names = reader.GetGDCMSeriesFileNames( directorypath,series_ID)reader.SetFileNames(dicom_names)#设置文件名image3D = reader.Execute()#读取dicom序列
import SimpleITK as sitk# Dicom序列所在文件夹路径(在我们的实验中,该文件夹下有多个dcm序列,混合在一起)file_path = "/data/jianjunming/BEOT/BEOT_1st/B/B13-5219998/"# 获取该文件下的所有序列ID,每个序列对应一个ID, 返回的series_IDs为一个列表series_IDs = sitk.ImageSeriesReader.GetGDCMSeriesIDs(file_path)# 查看该文件夹下的序列数量nb_series = len(series_IDs)print(nb_series)# 通过ID获取该ID对应的序列所有切片的完整路径, series_IDs[0]代表的是第一个序列的ID# 如果不添加series_IDs[0]这个参数,则默认获取第一个序列的所有切片路径series_file_names = sitk.ImageSeriesReader.GetGDCMSeriesFileNames(file_path, series_IDs[0])# 新建一个ImageSeriesReader对象series_reader = sitk.ImageSeriesReader()# 通过之前获取到的序列的切片路径来读取该序列series_reader.SetFileNames(series_file_names)# 获取该序列对应的3D图像image3D = series_reader.Execute()# 查看该3D图像的尺寸print(image3D.GetSize())# 将序列保存为单个的DCM或者NRRD文件sitk.WriteImage(image3D, 'img3D.dcm')# sitk.WriteImage(image3D, 'img3D.nrrd')
下面分别分析一下上面用到的几个信息:
1,SimpleITK.ReadImage(path)
最简单的在path中读取图像信息,一般读取格式为mhd文件与dicom文件
2,SimpleITK.GetArrayFromImage(itk_img)
将读取出来的图像信息用像素值表示出来 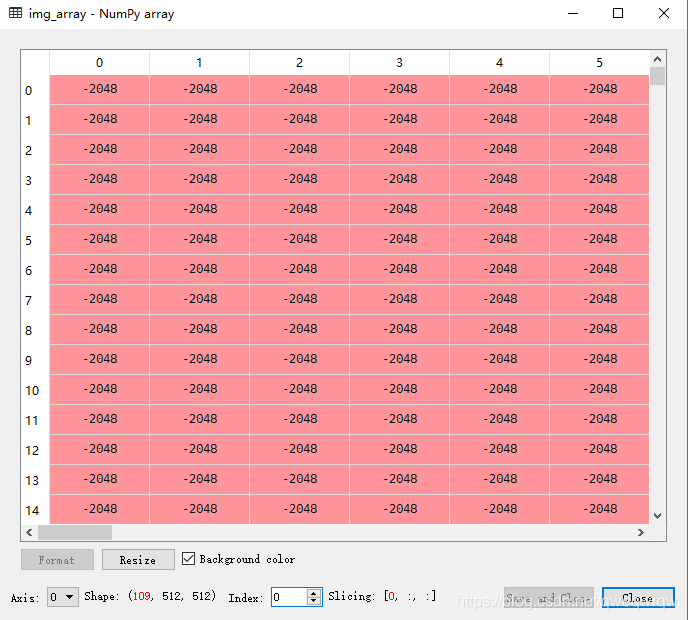
如上为读取出来的数组,底下的shape就是整个读取出来的图像大小,如此图为长宽分别为512,深度为109的图像。
3,itk_img.GetOrigin()
此操作为读取图像的原点信息,因为每张图像的不同,它的位置也与原点的距离不同,如此图: 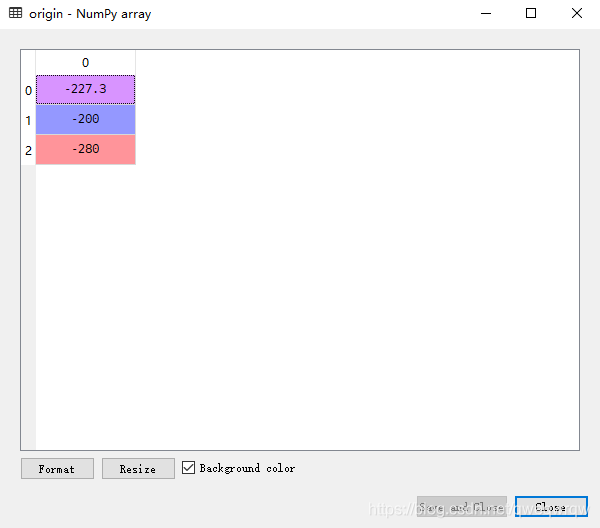
4, itk_img.GetSpacing()
此操作为读取图像的体素大小,我觉得这个数值更像尺度一些,每张图都不同 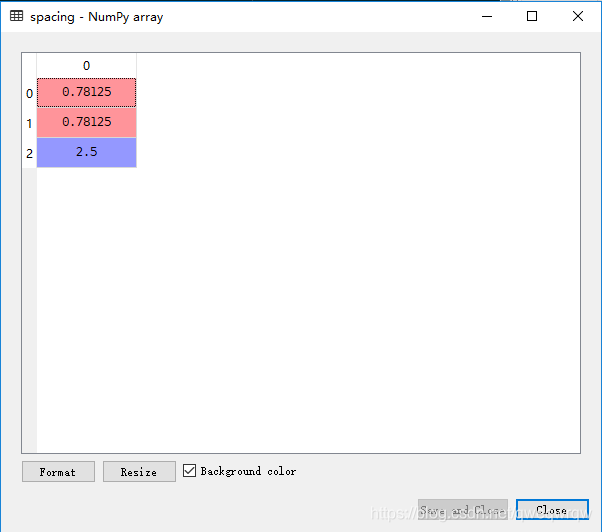
本图z的尺度为2.5,意思就是每1mm显示为2.5个深度。
5,itk_img.GetDirection()
此操作为读取图像方向,一般一系列图像都是同样的:
[1., 0., 0.,
0., 1., 0.,
0., 0., 1.]
6,具体分析一下Origin与Spacing:
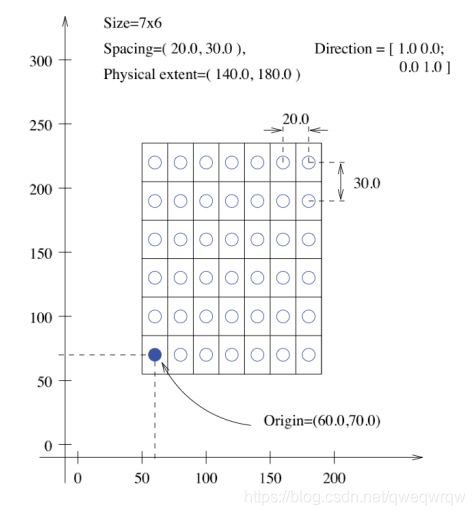
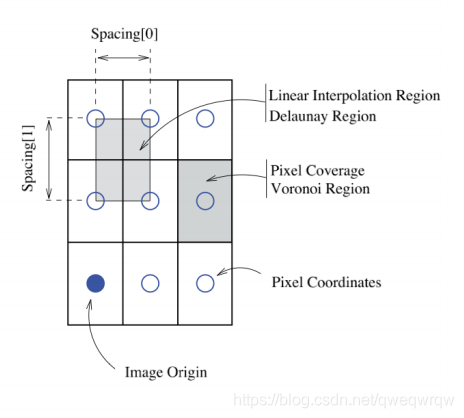
上面两图很好的诠释了Origin与Spacing与图像的关系
原文链接:https://blog.csdn.net/qweqwrqw/article/details/88427971

