蛋白质序列数据库

三大数据库共同构建蛋白质序列数据库——uniprot 数据库
Uniprot 数据库
uniprot 数据库三个层次数据库:
1)UniPrac:收录所有UniProt 数据库子库中的蛋白质序列,量大,粗糙。
2)UniRef:归纳UniProt 几个主要数据库并将重复序列去除后的数据库。
3)UniProtKB:有详细注释并与其他数据库有链接的数据库(Swiss-Prot / TrEMBL)
uniprot 数据信息内容
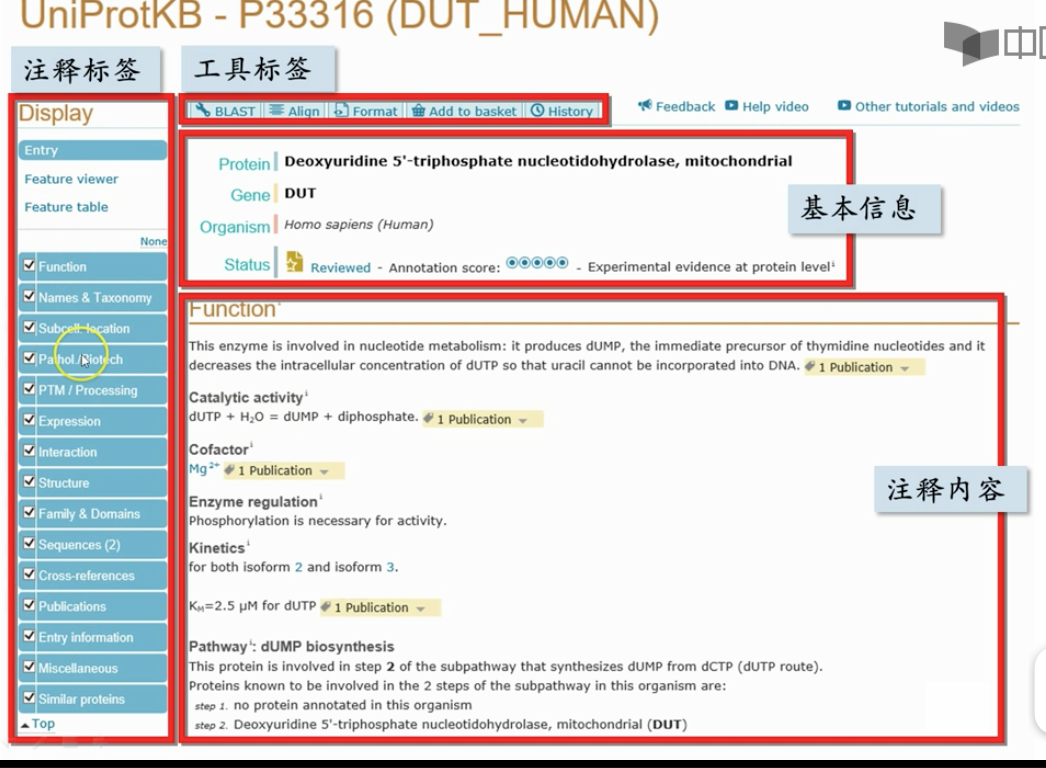
亚细胞定位
成熟的蛋白质必须在特定的部位才能发挥生物学功能,蛋白质在不同组分中的定位即为蛋白质的亚细胞定位。
亚细胞定位,对蛋白质的生理功能有着直接的影响。
处于合适的位置才能发挥其正常功能。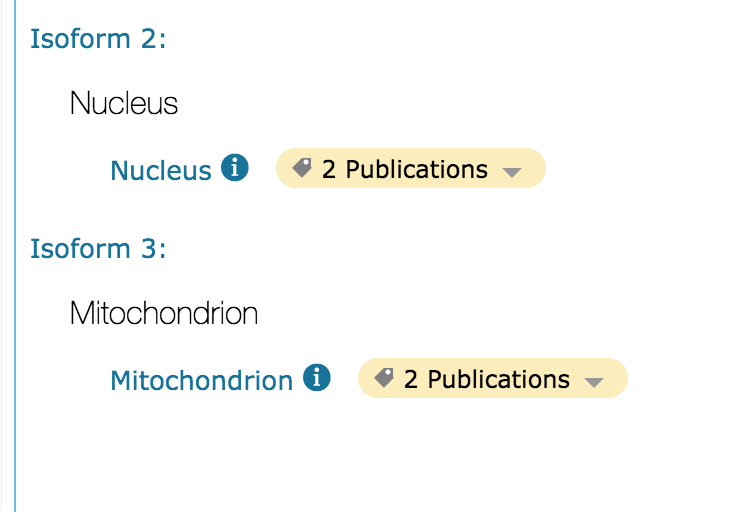
可以看到两种异构体信息,分别位于细胞核与线粒体中。
这与先前在genebank 中看到的两端mRNA 信息一致。(多出一段序列的是信号肽,对应线粒体定位)
pathol/biotech
提供蛋白质突变或缺失导致的疾病及表型信息。
PTM/processing
提供蛋白质翻译后修饰或翻译后加工的信息。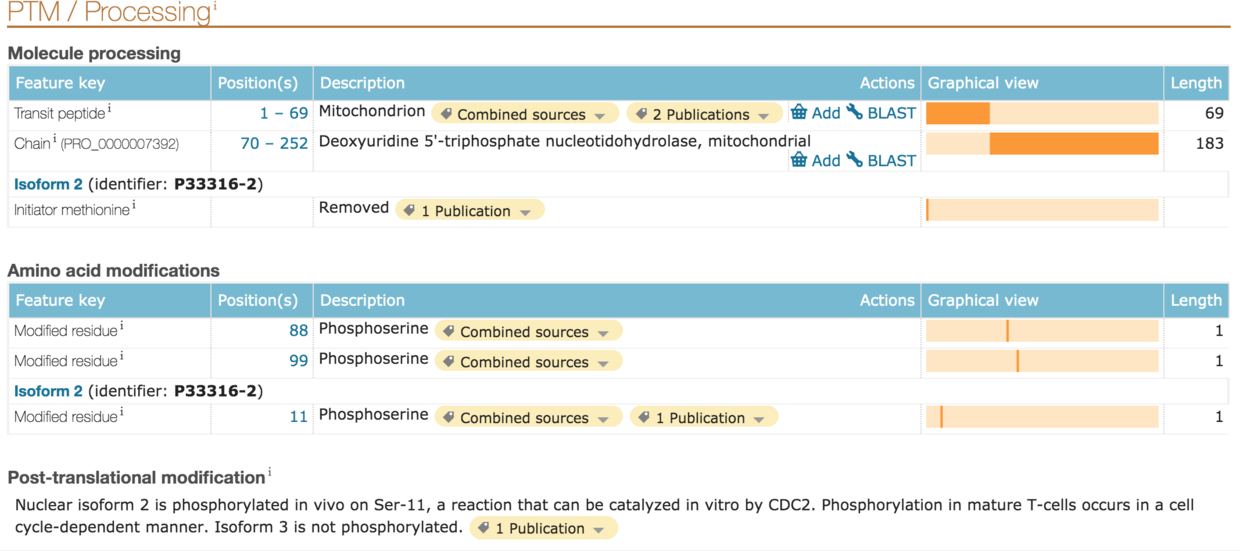
Expression
提供了基因在mRNA水平或蛋白质水平的表达信息,或者在不同器官的表达信息。
interaction
蛋白质相互作用信息。
1)uniproKB 中有直接记录的两两相互作用蛋白质信息。
2)其他蛋白互作数据库记录。如string 网络数据库的链接。
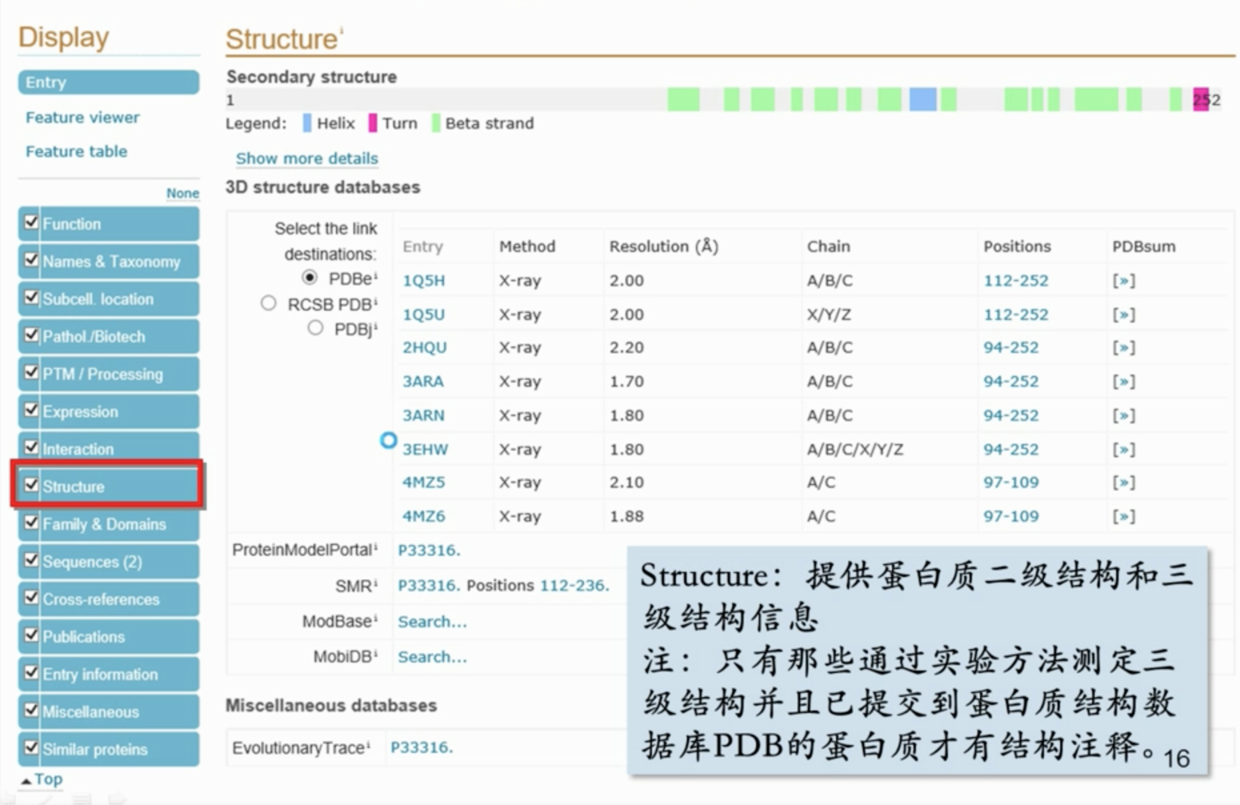
Structure
family&domains
提供蛋白质家族及结构域信息。
有三个重要的区域与其他蛋白结合。
还有与系统发生学的一些内容
sequence
提供蛋白质氨基酸序列信息
多个isoforms 会显示多个序列信息
选择FASTA 获得该格式序列信息
cross-references
列出了所有其他含有该蛋白信息的数据库链接
similiar protein
在UniRef 中找到与该蛋白在序列水平上相似的其他蛋白质,并按一定的顺序分组。
导出uniprot 信息
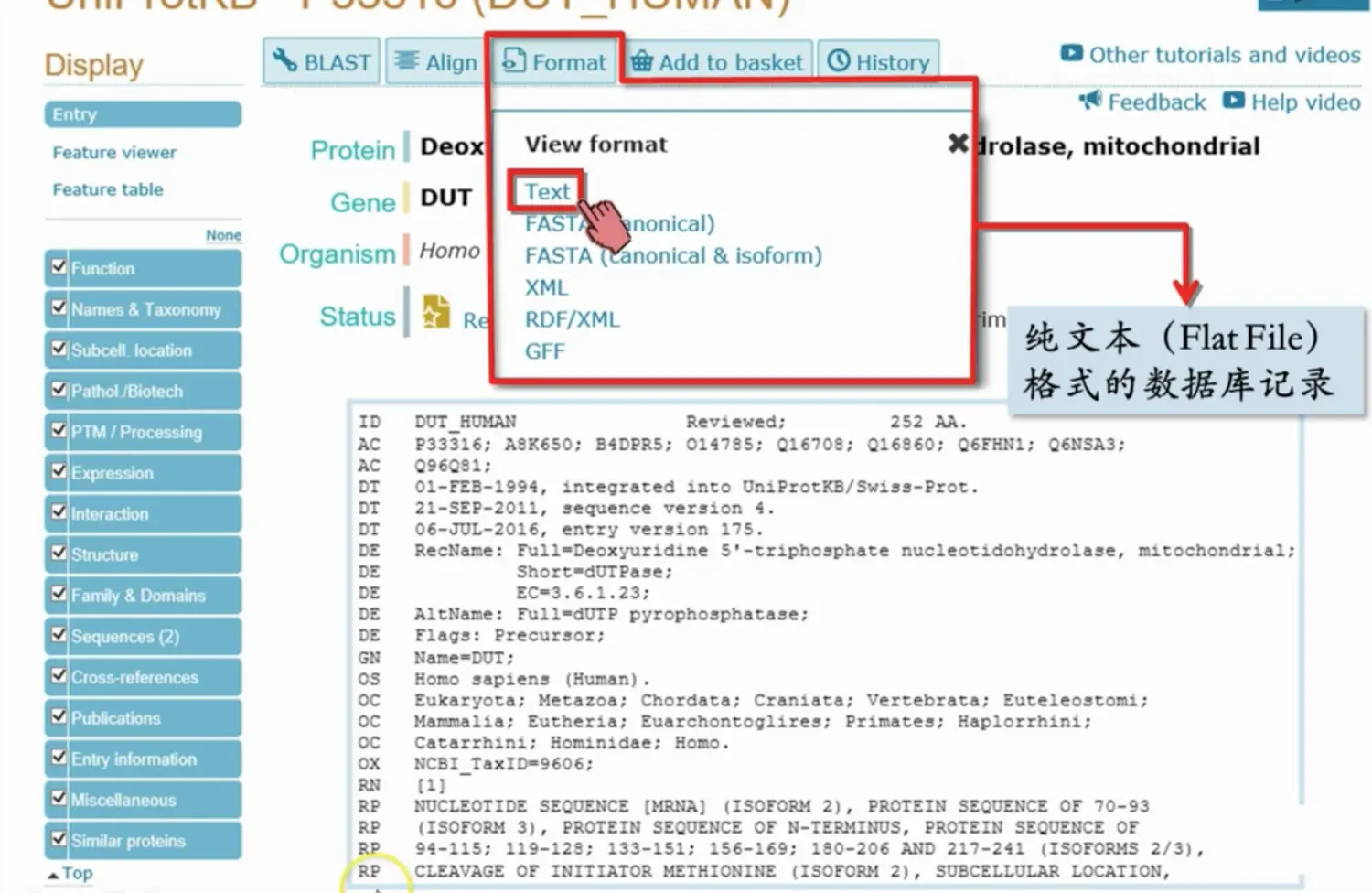
txt 格式文件会在左侧显示索引,表示不同类别的数据。
参见: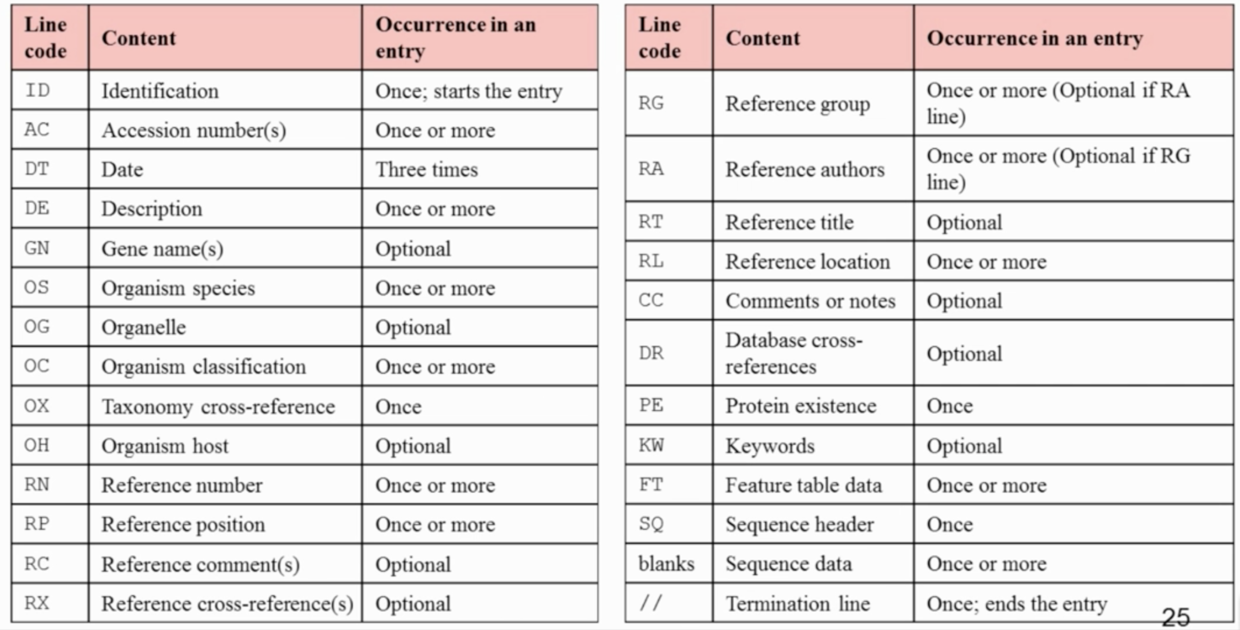
蛋白质结构数据库
蛋白质的结构
PDB 数据库
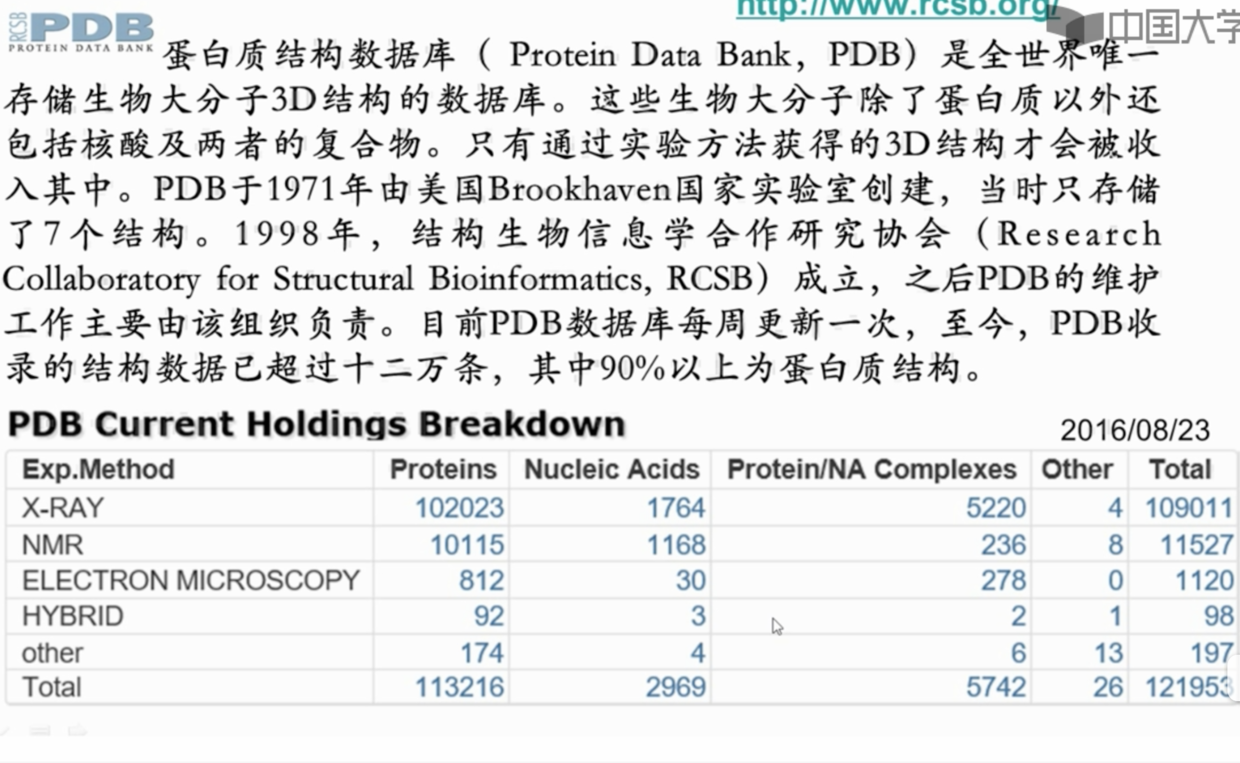
- 只有通过实验方法获得3D 结构才会被收入
周更(。。tql)
链接:http://www.rcsb.org/
搜索出来结果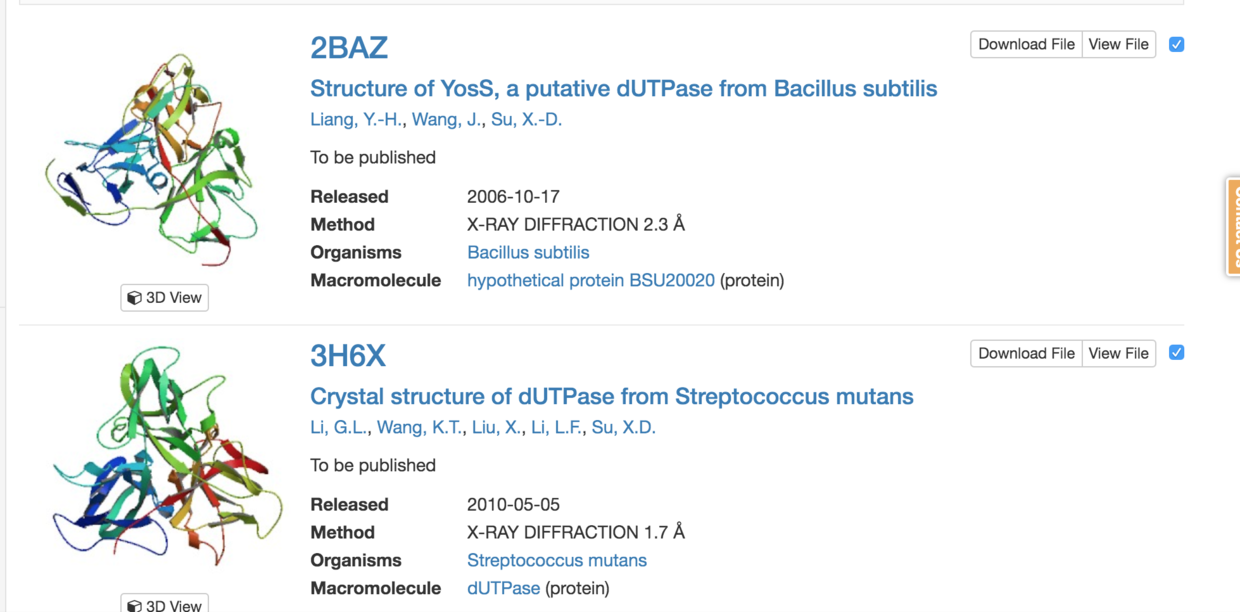
主要界面信息
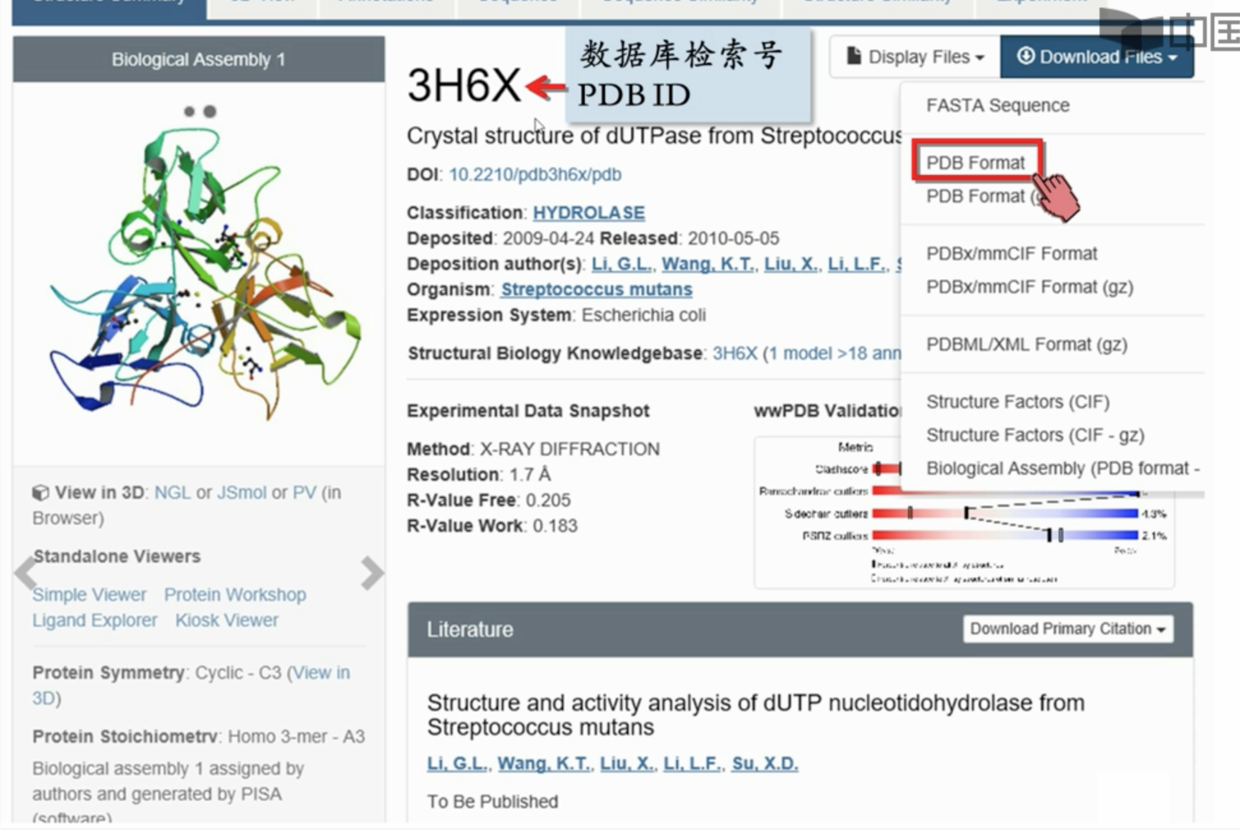
一个结构对应一个PDB ID
完全的信息存储在PDB格式的文件内。
PDB 文件信息

COMPND : 对各个分子进行描述。
SOURCE:每一个分子的实验来源。
KEYWDS:数据库搜索关键词。
EXPDTA:特定结构采用的实验方法。
JRNL:发表该结构的文献。
除此之外还有蛋白质的各级结构的具体信息。
实验参数部分
其中3D坐标部分的内容篇幅最长,也最为重要。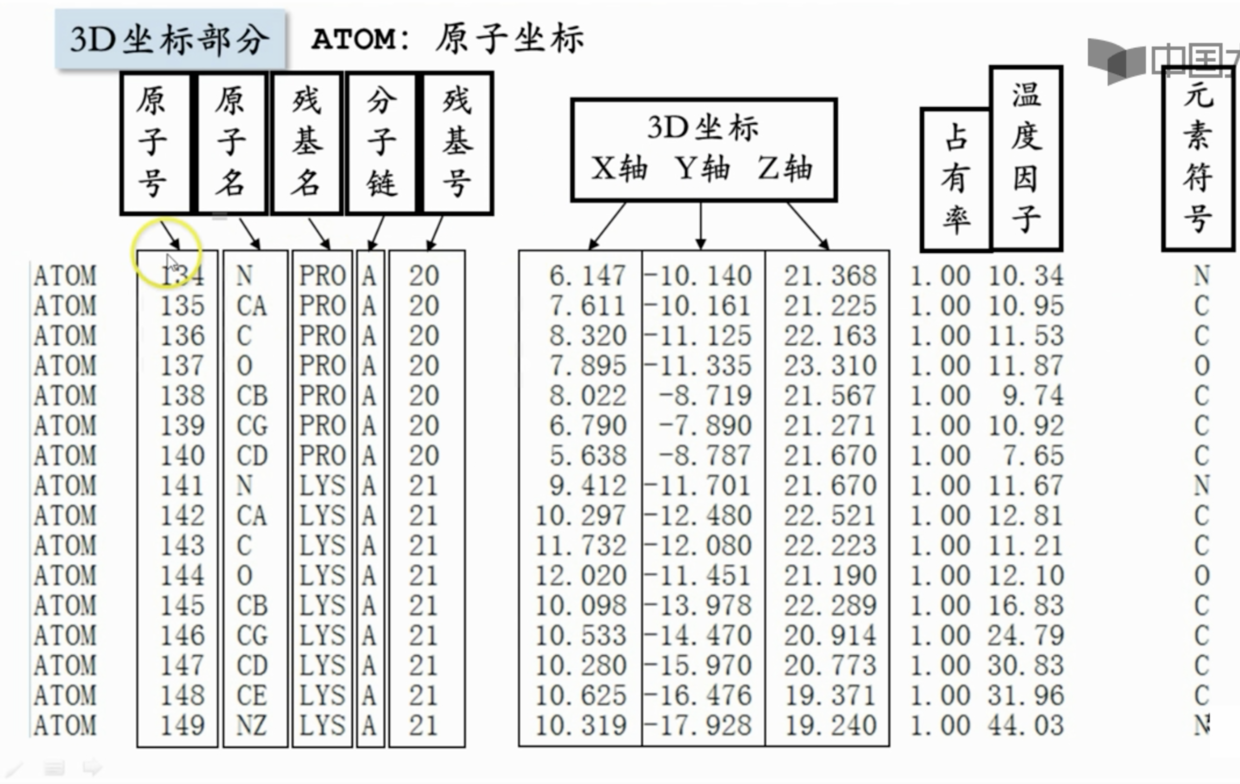
- PDB 实际存储的是3D 坐标,由此来构建3D模型。
3D可视化操作
由先前部分,知道了每一个3D 模型其实都是根据一些列的参数设定,并通过相关的化学键信息,连接而成。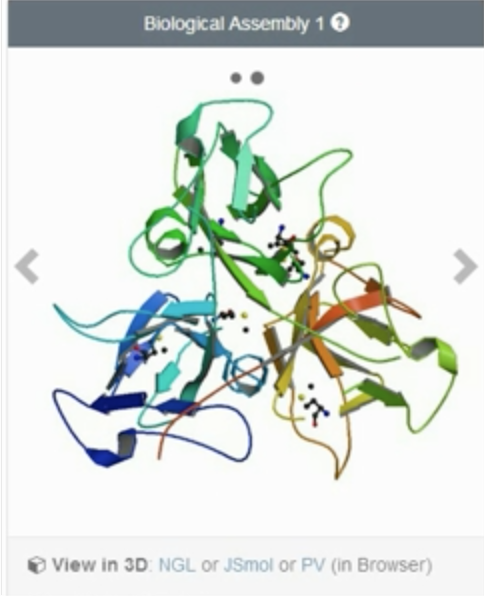
PDB提供了在线3D可视化软件(需要java 环境)
PDB 提供了一个在线查看功能结构的平台。但是功能不够全面,没有分析功能。
MDL 数据库
ACD-3D 数据库,提供世界各大化学品供应商的产品目录,可以买到具有潜在活性的候选化品。
即通过购买有潜在活性的候选中间物,避免了消耗在合成中间物上所需要的时间。

