要比较氨基酸或核酸的相似性,则需要借助替换记分矩阵。
替换记分矩阵一般分为1)DNA 替换记分矩阵;2)蛋白质替换记分矩阵。
DNA 替换记分矩阵
等价矩阵
unitary matrix,最简单的记分矩阵。其中,相同核苷酸的匹配得分为1,不同为0。由于不含有碱基理化性质和不区别对待的替换,较少使用。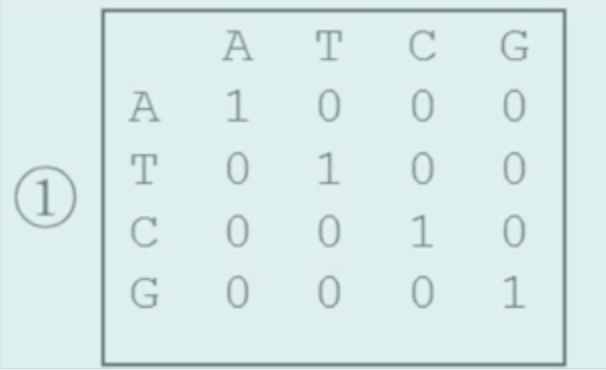
转换-颠换矩阵
transition-transversion matrix,核酸的碱基按照环结构划分两类:嘌呤(腺嘌呤A、鸟嘌呤G),有两个环;与嘧啶(胞嘧啶C、胸腺嘧啶T),只有一个环。如果环数发生变化,则称为颠换(嘌呤<->嘧啶),得-5分;如果环数不变,(C<->T,A<->G),称为转换,得-1分。而一般在进化过程中,转换发生的频率比颠换高。而相同碱基,得1分。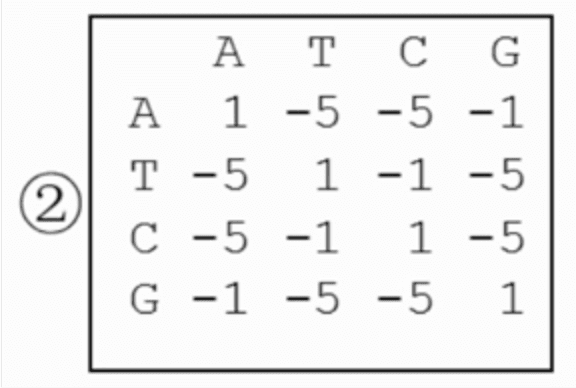
BLAST矩阵
最早应用于BLAST 软件,得名。
经过大量比对后总结出的矩阵:相同核酸为+5,不同为-4。这个矩阵被广泛使用。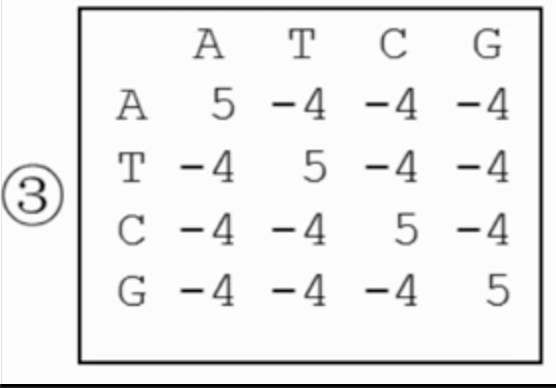
蛋白质替换记分矩阵
等价矩阵
同DNA 等价矩阵。一般也不选用。
PAM矩阵(Dayhoff突变数据矩阵)
PAM矩阵基于进化原理。如果两种氨基酸替换频繁,说明自然界容易接受这种替换,那么这对氨基酸替换得分就高。
PAM矩阵是目前蛋白质序列比较中最广泛使用的记分方法之一,基础的PAM-1 矩阵反应的是进化产生的每一百个氨基酸平均发生一个突变的量值(统计方法得到)。
PAM-1 自乘n次,可以得到PAM-n,即发生了多次突变。

BLOSUM矩阵(blocks substitution matrix)
BLOSUM 矩阵都是通过对大量符合特定要求的序列计算而来。
氨基酸替换记分矩阵选择
一般根据以下内容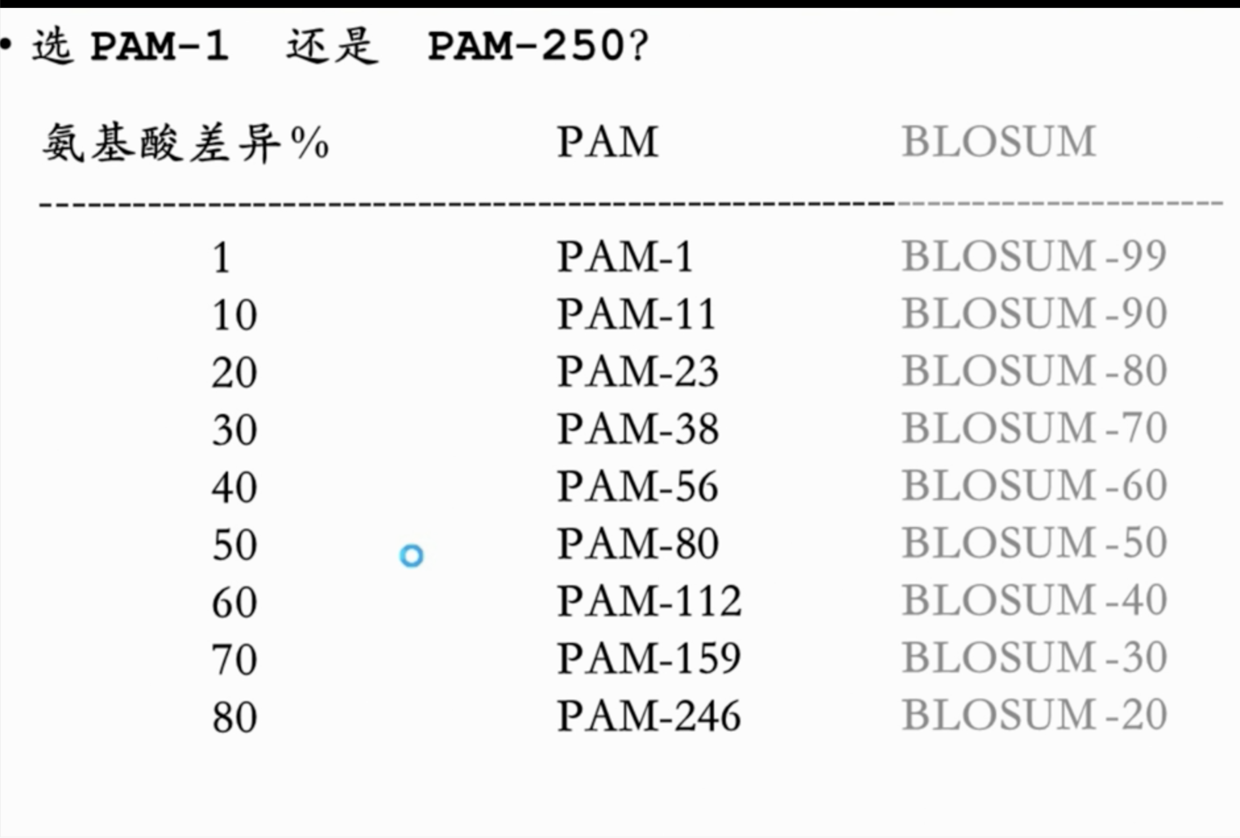
但也并不是所有的PAM 数据库都有,比如PAM-246就没有现成的,只有PAM-250。
PAM后面的数体现的是差异度,但不是直接等于差异度。
BLOSUM后面的数体现的是相似度,并且直接等于相似度。

ps: 要是听不懂替换记分矩阵,用BLOSUM-62就完事了。
其他两个蛋白质替换记分矩阵
遗传密码矩阵
genetic code matrix, GCM
疏水矩阵
根据氨基酸残基替换前后疏水性的变化而得到的得分矩阵。若一次氨基酸替换疏水特性不发生明显变化,则这种替换得分高,否则得分低。
该矩阵物理意义明确,有一定意义,适用于偏重蛋白质功能方面的序列比对。
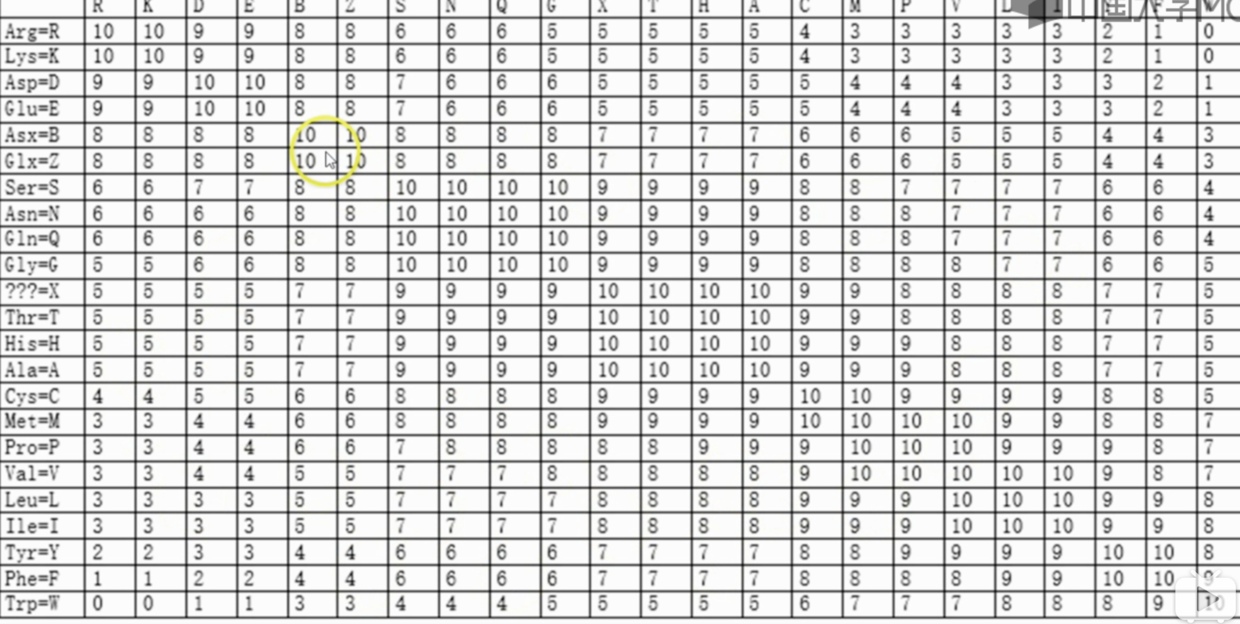
例子

- 再回到先前氨基酸序列相似度的问题。
可以通过BLOSUM-62 记分矩阵求解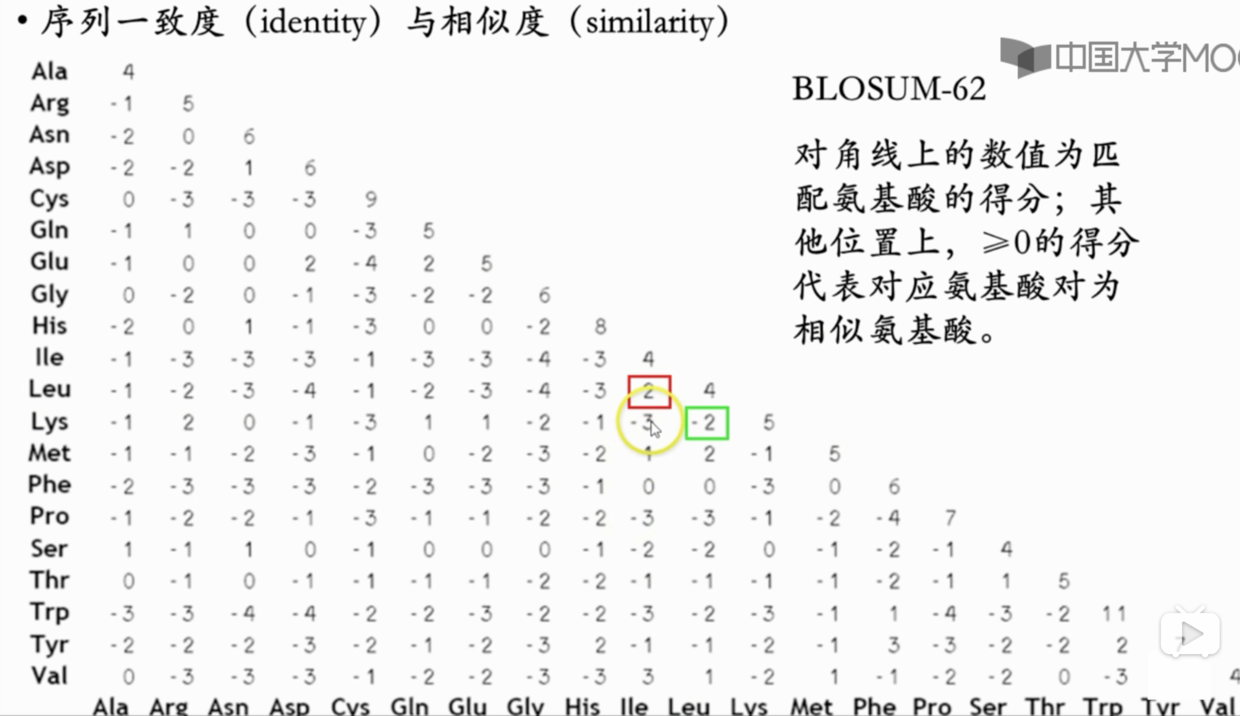
- 其中可以看到,有1个相似(正值),一个不相似。
因此相似度为(1+2)/ 4 = 75%
但新的问题又来了,知道了如何比较两个长度相同的序列,那么如何比较不同长度的序列,并求出它们的一致度与相似度呢?

