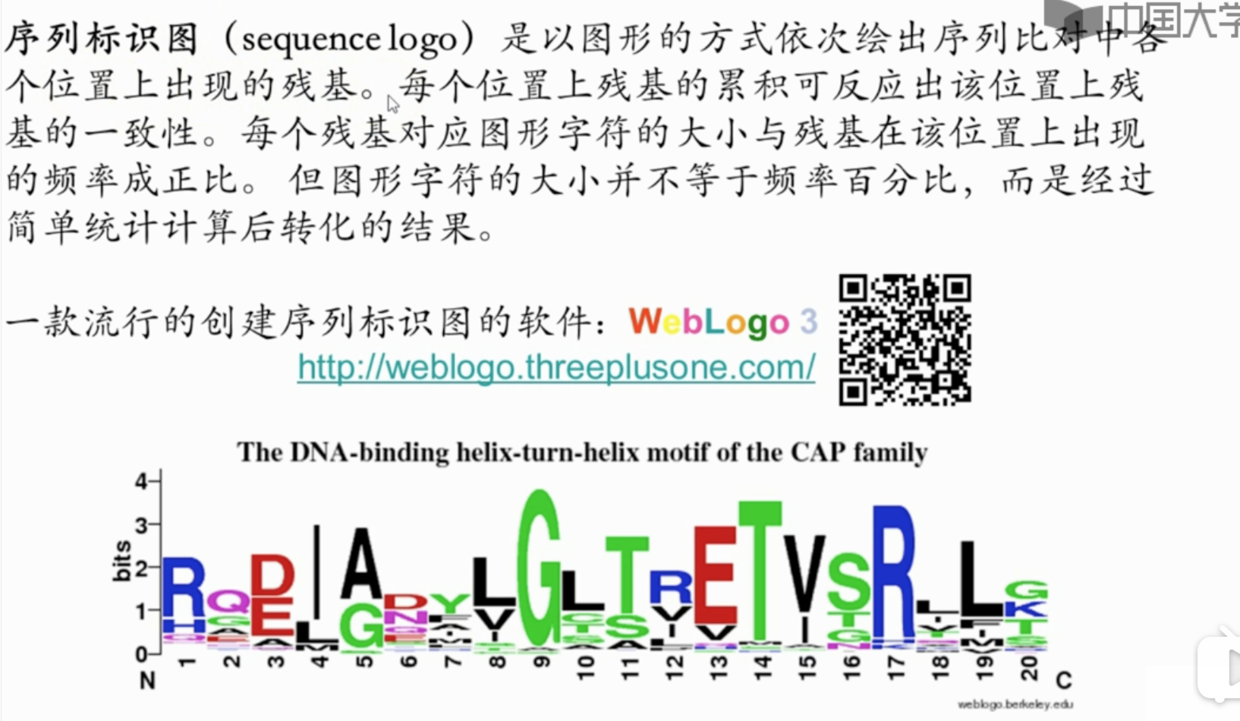
序列标识图
sequencelogo 工具使用
使用在线的工具,WebLogo3
http://weblogo.threeplusone.com/
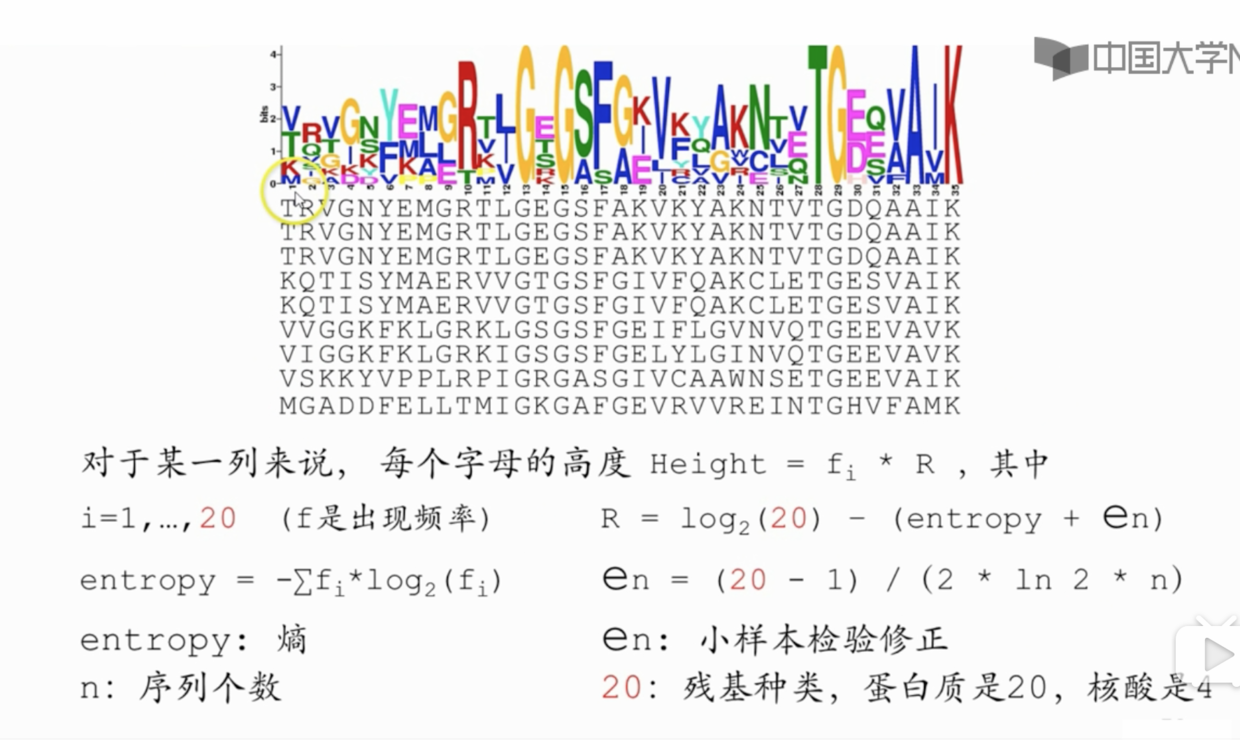
字母大小都是根据上方的运算求得的,通常来说出现次数多的字母,体积也就越大,而如果某一列只有一个大字母,说明该列非常保守。
因为在计算过程中设计了熵值,因此如果某列序列字母越混乱,熵值越大,则字母越矮。越有规律,熵值越小,字母越高。
开始比对
目前weblogo 已经支持大多数的多序列比对结果格式。
解读结果
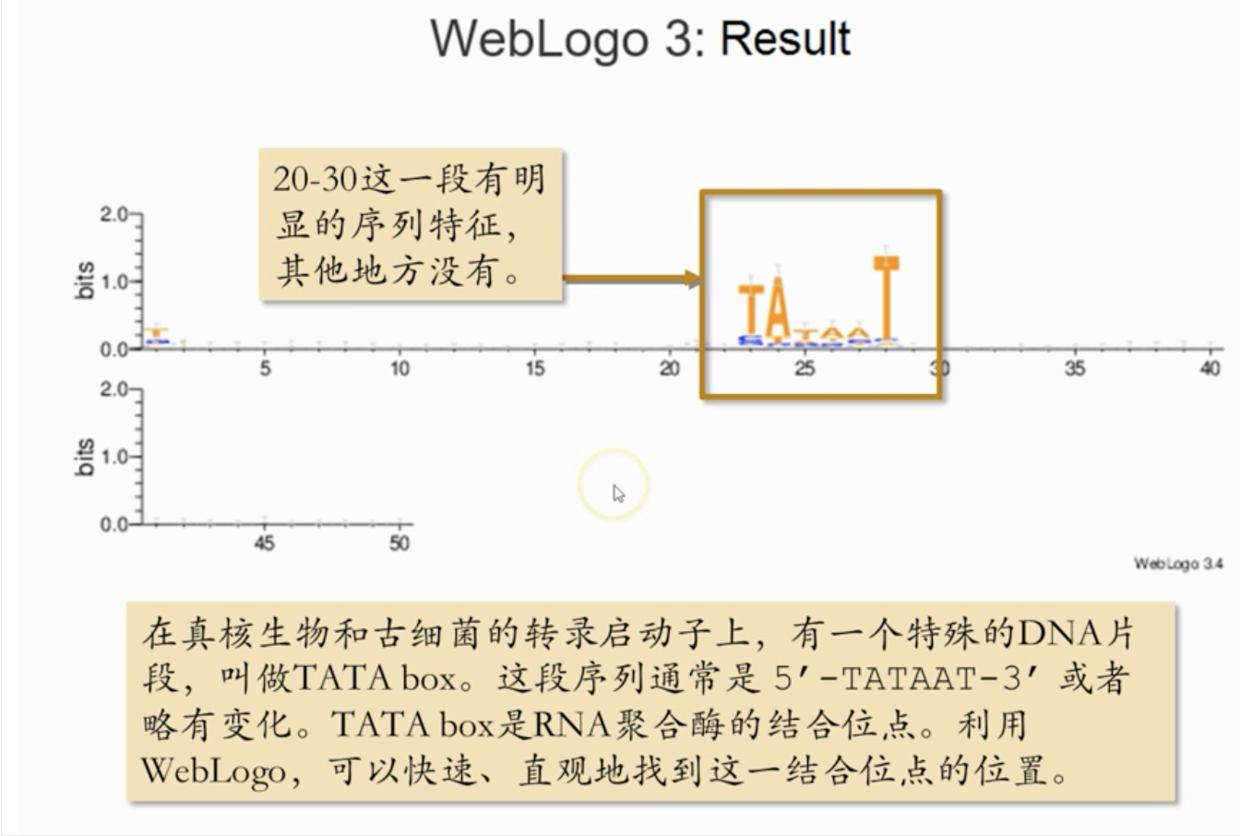
找寻保守序列的基序
在核酸/蛋白质 序列中存在有特定模式的序列片段,这些片段称为序列的基序(motif)。序列基序与序列功能密切相关。
比如在BLAST 时所说到的“精准版BLAST”,即PHI-BLAST,便是找寻序列基序。
MEME
MEME 便是一款可以自动从一组相关的DNA或蛋白质序列中发现序列基序的软件。
http://meme-suite.org/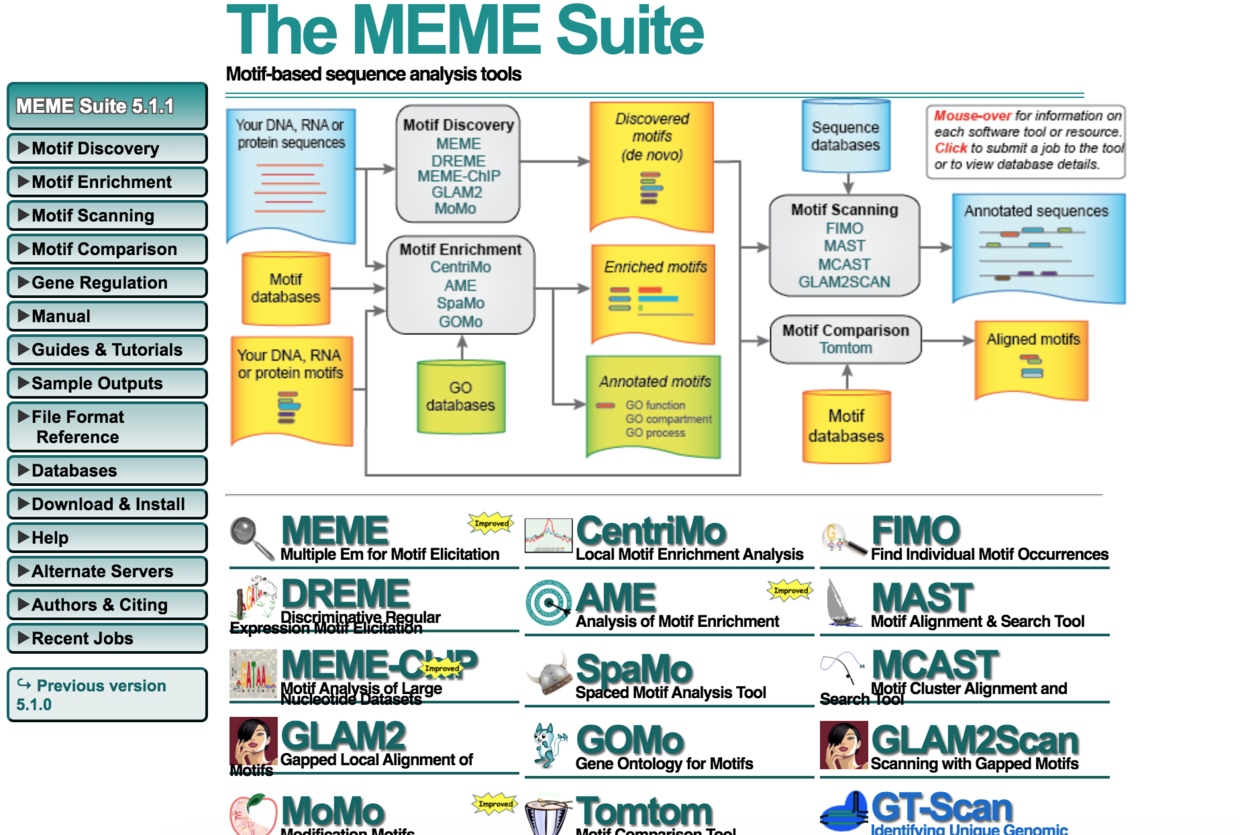
MEME 包含了各种软件,我们暂时只用MEME。
http://meme-suite.org/tools/meme
MEME 支持格式为FASTA。
输入界面介绍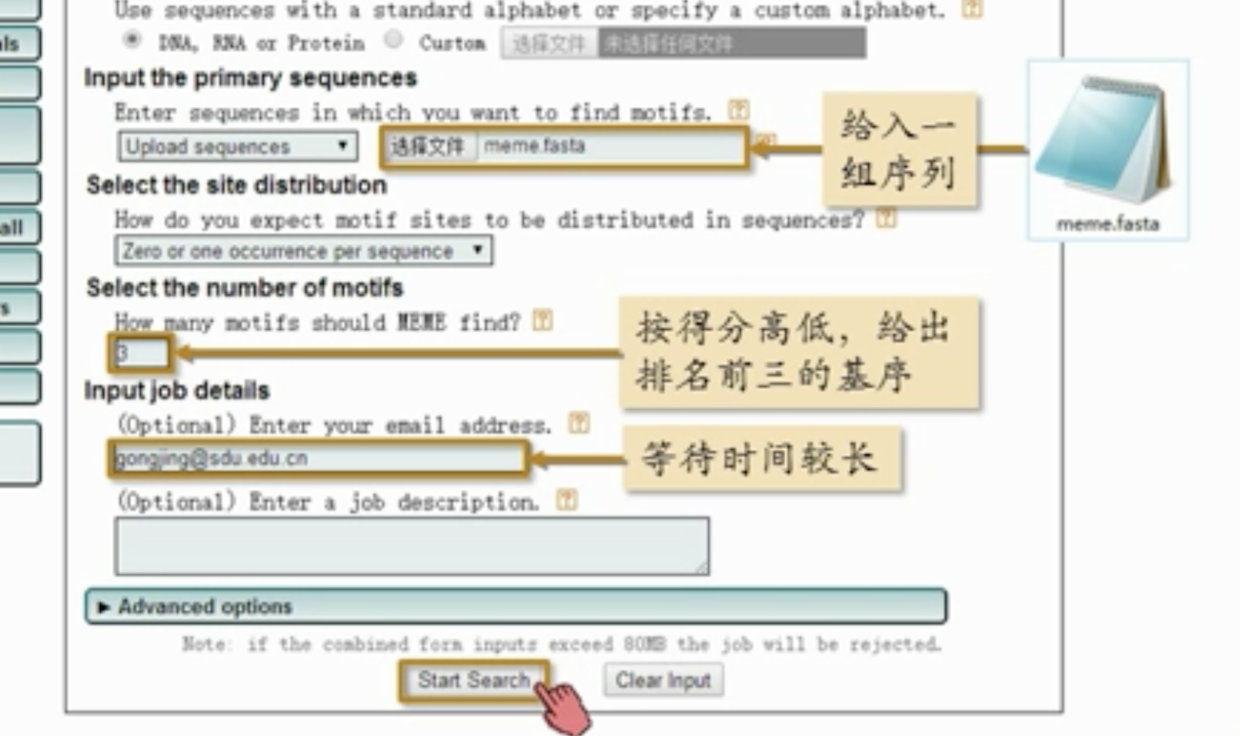
结果显示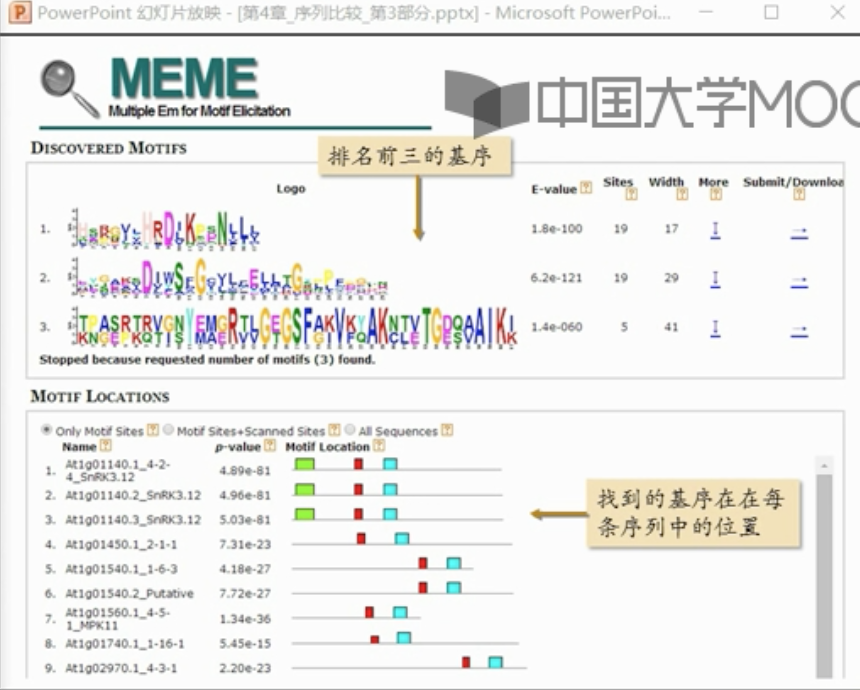
还可将基序提交到数据库,进行针对该基序的序列相似性搜索
蛋白质的指纹图谱
一个蛋白质的指纹(prints)就是一组保守的序列基序,用于刻画蛋白质家族的特征。这些基序由多序列比对结果获得,且它们在氨基酸序列上是不相邻的,但在三维结构中,它们可能紧密结合在一起。
PRINTS 是蛋白质序列指纹图谱数据库,存储了目前已发现的绝大多数的蛋白质家族的指纹图谱。对于一个陌生的蛋白质,只要看看它的序列是否符合某个家族的图谱,就可以对它进行分类并预测它的功能。
PRINT 数据库
http://130.88.97.239/PRINTS/index.php
我们可以通过文本搜索
比如我们搜索”TRANSFERRIN”
解读结果信息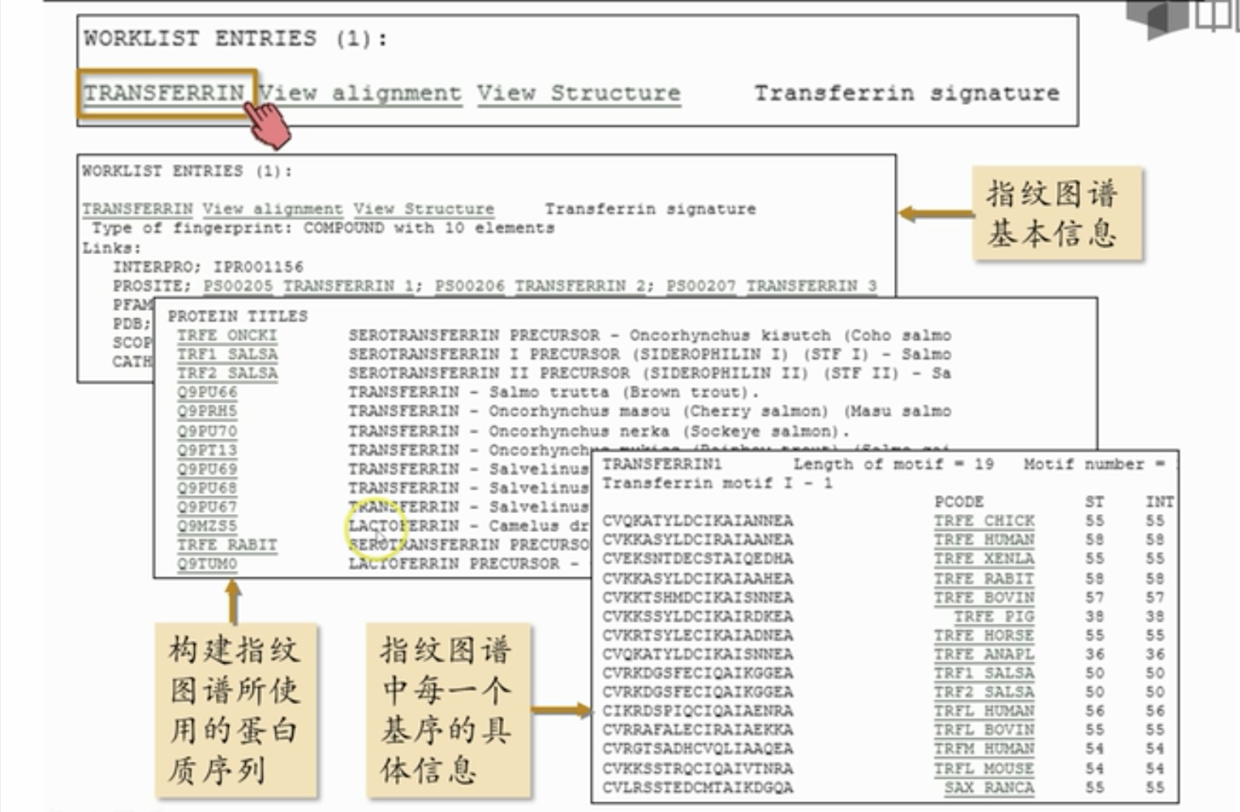
查看指纹图谱所使用的多序列比对结果
ps: 我使用的数据搜索结果栏目中,没有view structure 一项了。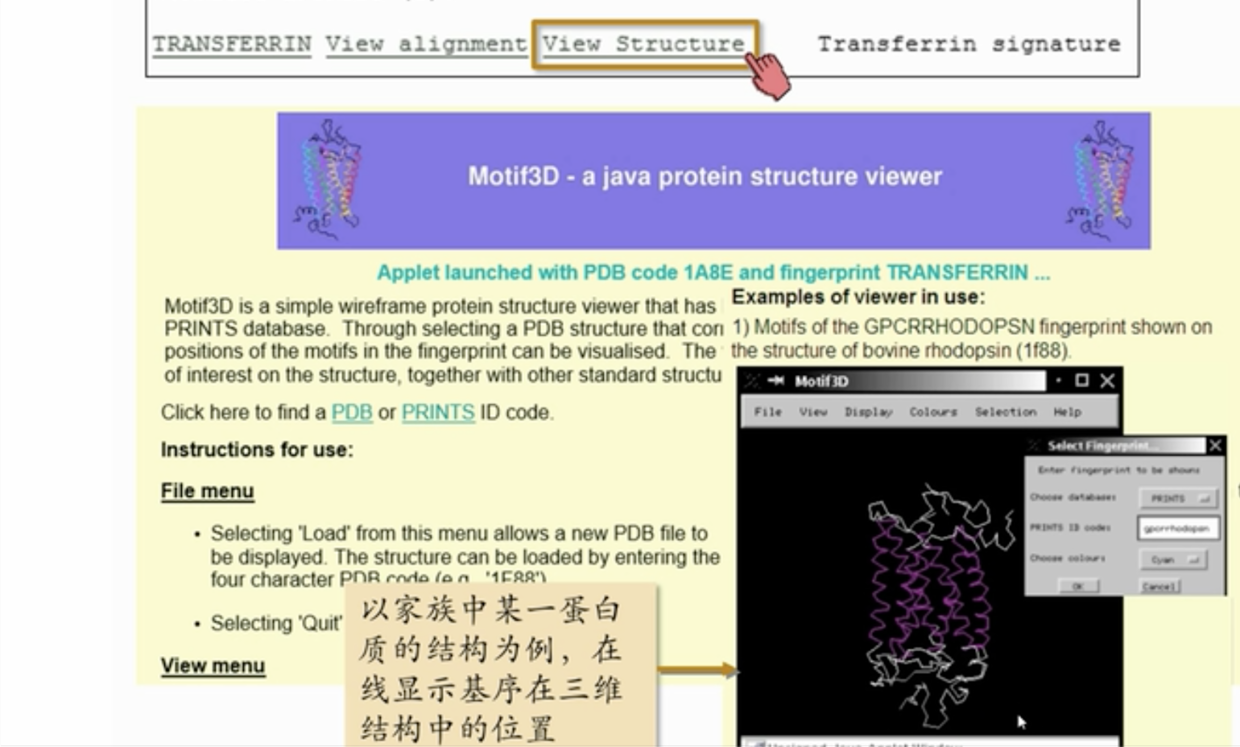
还可以进行指纹匹配
即搜索某一序列所匹配的指纹图谱
这里只能输入纯序列
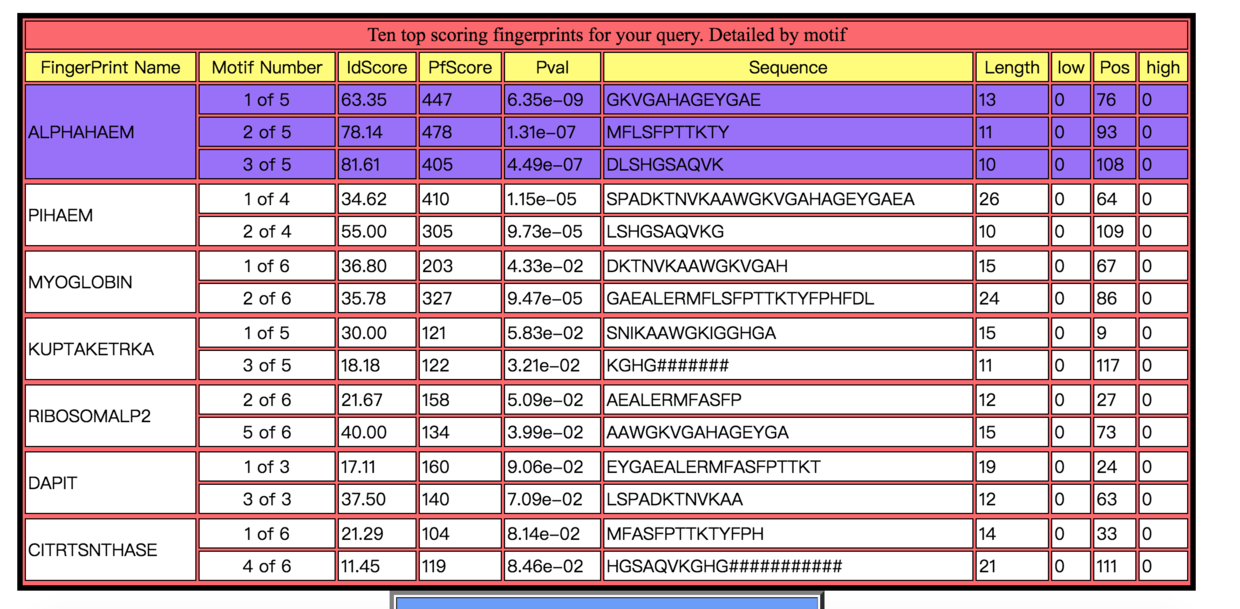
分别显示排名前三和前十的指纹图谱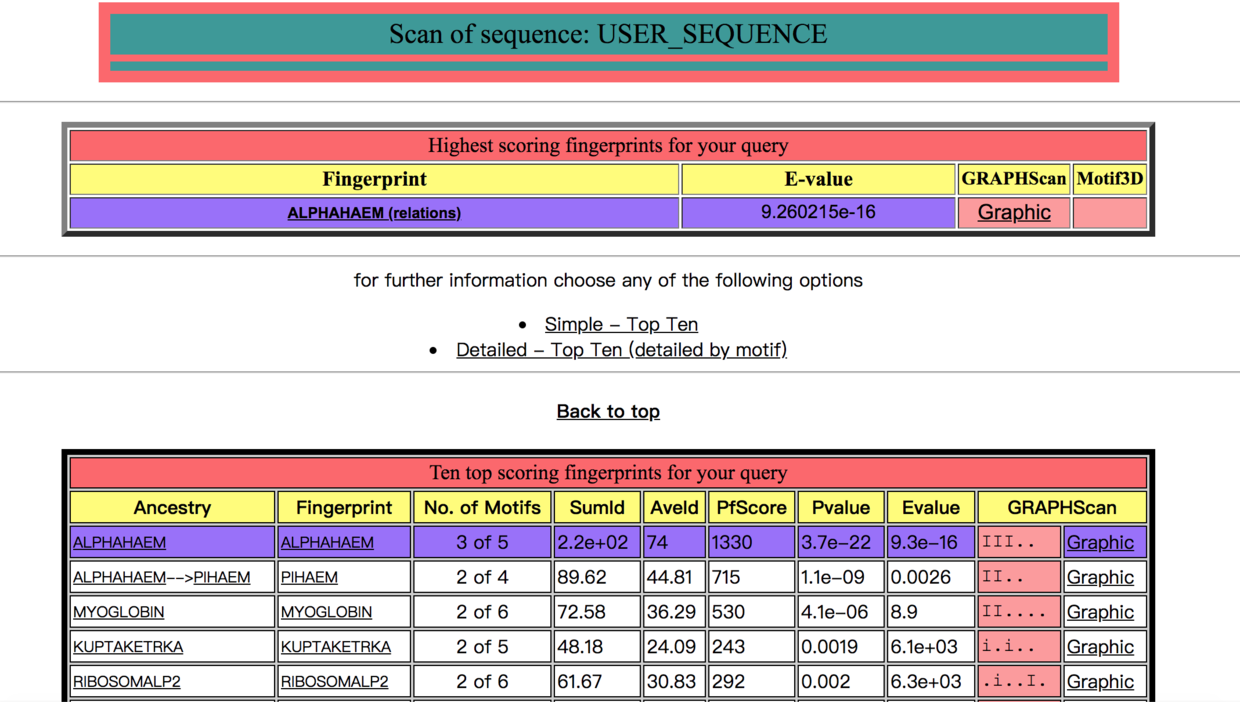
可以看到匹配的蛋白指纹图谱各个基序在输入序列中匹配的位置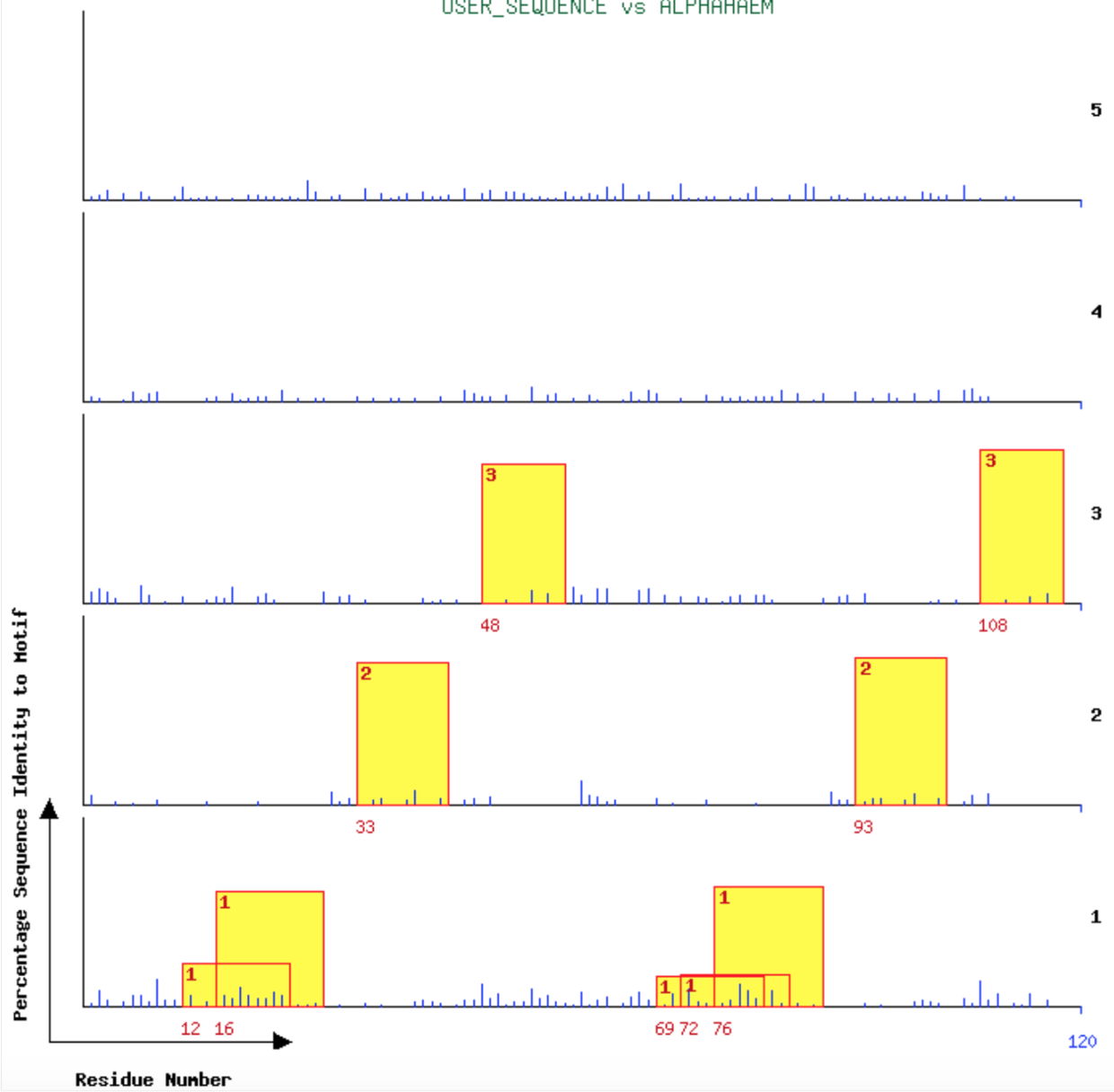
后面一个表格还提供了匹配蛋白质家族的基序在输入序列中具体的匹配片段。