virtual screening 虚拟筛选
虚拟筛选,也称生物活性筛选,即在进行生物活性筛选之前,在计算机上对化合物分子进行预筛选,以降低实际筛选化合物的数目,同时提高先导化合物的发现效率。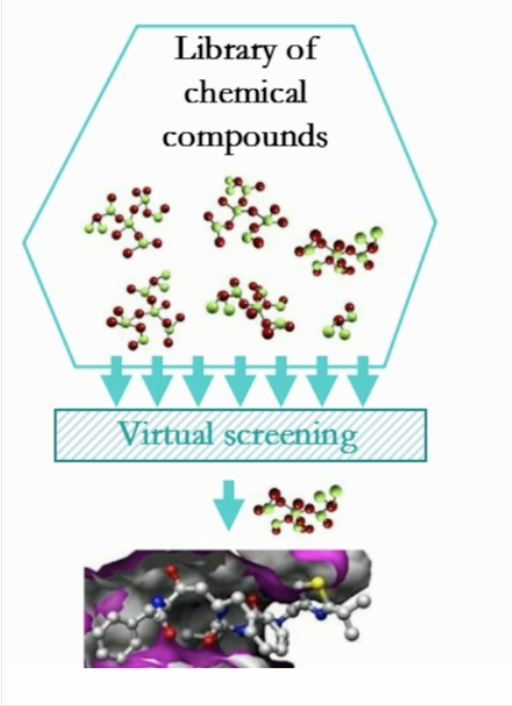
大多数虚拟筛选软件都很贵。autodock 也可以做虚拟筛选。
- zinc 提供了三千五百万个小分子以供docking
步骤
1)受体模型的建立;
可以采用同源模型建立的方法,快速获取靶标模型。
结合位点的描述;
直接从配体-受体复合物获取,或根据生物学特征手动调整。
2)建立小分子数据库;
3)对接、打分;
4)命中化合物的后处理。
根据化合物的成药性等进行筛选。
对接中存在的问题
- 分子柔性
- 溶剂化效应(人体环境影响)
- 打分函数
从ZINC 中寻找小分子
http://zinc.docking.org/substances/home/
关于zinc 15 的具体用法可参见:https://www.jianshu.com/p/39d013310950
- autodock 需要特定的保存格式

- 关于使用autodock 和zinc 进行虚拟筛选步骤可简化为以下方面:
1)批量从zinc 上筛选并下载.mol2 文件。(用于筛选的结构也需下载,可作为阳性对照)
2)从下载的.mol2 文件分离单独的结构的.mol2 文件,并将文件格式通过autodock 转换为.pdbqt 格式。
3)加工用于比较的蛋白质分子(grid box 等),也转换为.pdbqt格式。
4)将小分子和蛋白质进行对接(平行对接)。(参考:8核192次对接需要2h)
5)将对接结果排序,按照能量选择。
6)查看排名第一小分子对接的具体情况。
7)通过湿实验验证。 - autodock vina 可自定义脚本,以完成虚拟筛选。(简化了虚拟筛选步骤)
反向对接
反向对接,target fishing,是通过把一个小分子与多个靶标蛋白进行分子对接,寻找潜在的靶标。(与虚拟筛选刚好反过来)
scPDB 提供了一个蛋白靶标数据库(设计了好了grid box结合区域)。
分子动力学模拟


