朴素贝叶斯模型
贝叶斯定理
x:观察变量
C:一个潜在特性
P(C|x):后验概率
P(C):先验概率
P(xIC):似然性likelihood
对于具备n个特征的观测值x=(x1,x2,…,xn),属于第k个分类的概率记为:
P(Ck|x) = P(Cklx1,x2,… ,xn)
贝叶斯公式:
若特征间相互独立,则
贝叶斯公式的分子:
P(Ck):第K类在总体中的先验概率
P(xi|Ck):第K类中观察到X时的条件概率
1、高斯模型
X——在每一类中都是服从高斯分布的连续值。
:::info
sklearn.naive_bayes.GaussianNB对象可以构建朴素贝叶斯的高斯模型。只要把训练数据代入到fit函数即可完成模型训练。再将待分类的特征数据代入predict函数即可以获得分类结果。
:::
2、Multinomial(多项式)模型
xi——特征i发生的频次
x——n个特征的直方图
:::info
sklearn.naive_bayes.MultinomialNB对象中的fit函数和predict函数分别用来训练和预测。
:::
3、Bernoulli(伯努利)模型
xi——特征i发生/不发生
x——n个特征发生与否的二进制序列
:::info
sklearn.naive_bayes.BernoulliNB对象可以构建伯努利模型。
:::
Logistic回归——朴素贝叶斯
混淆矩阵
将数据按待分的类别分组后,统计各组中模型分类或预测结果的矩阵。
二分类问题:阳性或阴性
| 真实分类 | |||
|---|---|---|---|
| 阳性 | 阴性 | ||
| 模型分类 | 阳性 | TP(真阳性)[正确] | FP(假阳性) |
| 阴性 | FN(假阴性) | TN(真阴性)[正确] |
总例数Total:TP+FP+FN+TN
准群率: 取值:[0,1] (真阳性、召回率、敏感性、检出率)
取值:[0,1] (真阴性、特异性、选择性)
精度(阳性预测值): 取值:[0,1]
阴性预测值: 取值:[0,1]
↑
↓
特征服从高斯分布的连续值时使用高斯模型
使用scipy.stats.probplot(x, dist,plot)函数检查鸢尾花数据是否服从高斯分布
import pandas as pdimport numpy as npfrom sklearn import datasetsfrom scipy import statsimport matplotlib.pyplot as pltiris=datasets.load_iris()#print((iris))plt.figure(figsize=(12,15))5for n in range(4):print(n)for m in range(3):x=(iris.data[m*50:m*50+50,n]-iris.data[m*50:m*50+50,n].mean())/iris.data[m*50:m*50+50,n].std()plt.subplot(4,3,n*3+m+1)stats.probplot(x,dist='norm',plot=plt)plt.text(-2,2,iris.feature_names[n])if n==0:plt.title(iris.target_names[m])else:plt.title('')plt.xlim([-2.5,2.5])plt.ylim([-2.5,2.5])plt.plot([-2.5,2.5],[-2.5,2.5],c='g')plt.savefig('chap72.png')
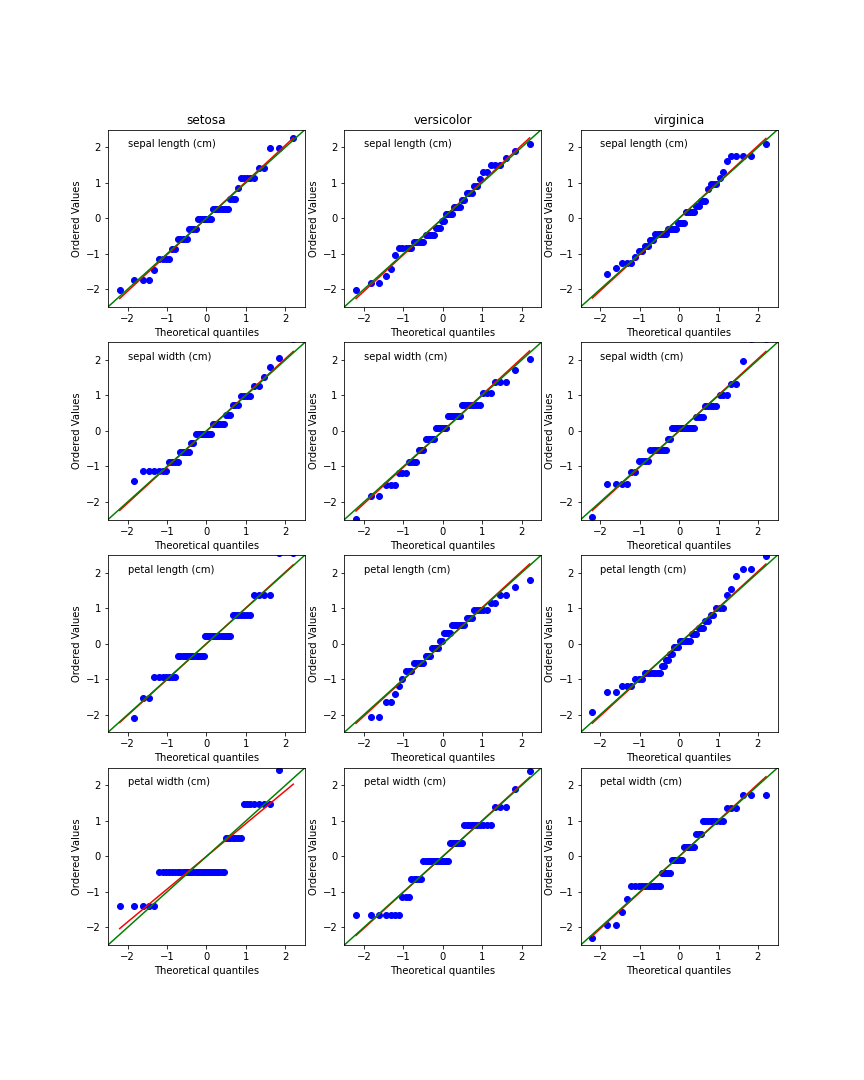
红色:probplot函数对所有散点最小二乘拟合的直线
绿色:45°对角线
可以看到speal length和speal width与高斯分布吻合的更好一些,第三个和第四个则差了一些。现在使用第一个和第二个来构建高斯模型。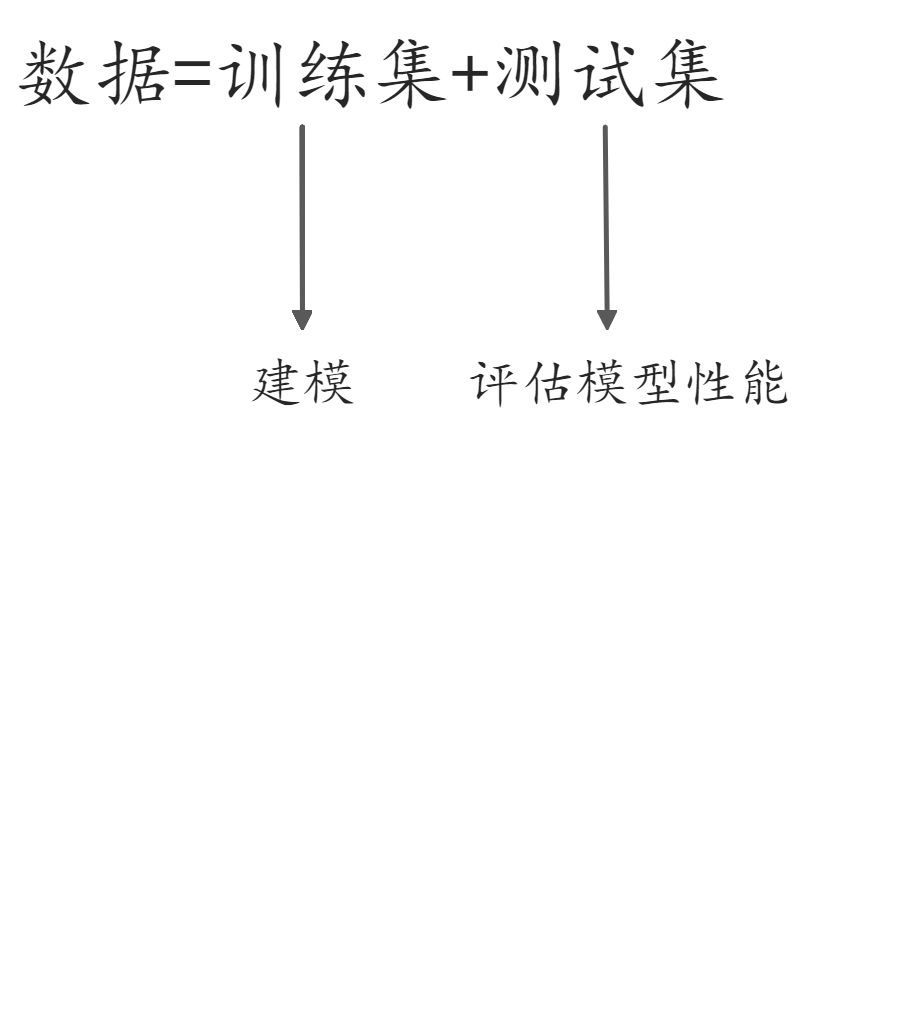
from sklearn.model_selection import train_test_split#X_train,X_test,Y_train,Y_test=train_test_split(iris.data,iris.target,test_size=0.2,random_state=0)X_train,X_test,Y_train,Y_test=train_test_split(my_data,iris.target,test_size=0.2,random_state=0)from sklearn.naive_bayes import GaussianNBclf=GaussianNB()clf.fit(X_train,Y_train)y_pred=clf.predict(X_test)Y=pd.DataFrame(np.transpose([Y_test,y_pred]),columns={'true_type','predict_type'})Y.head(30)
将测试结果和真实的结果打印出来进行对照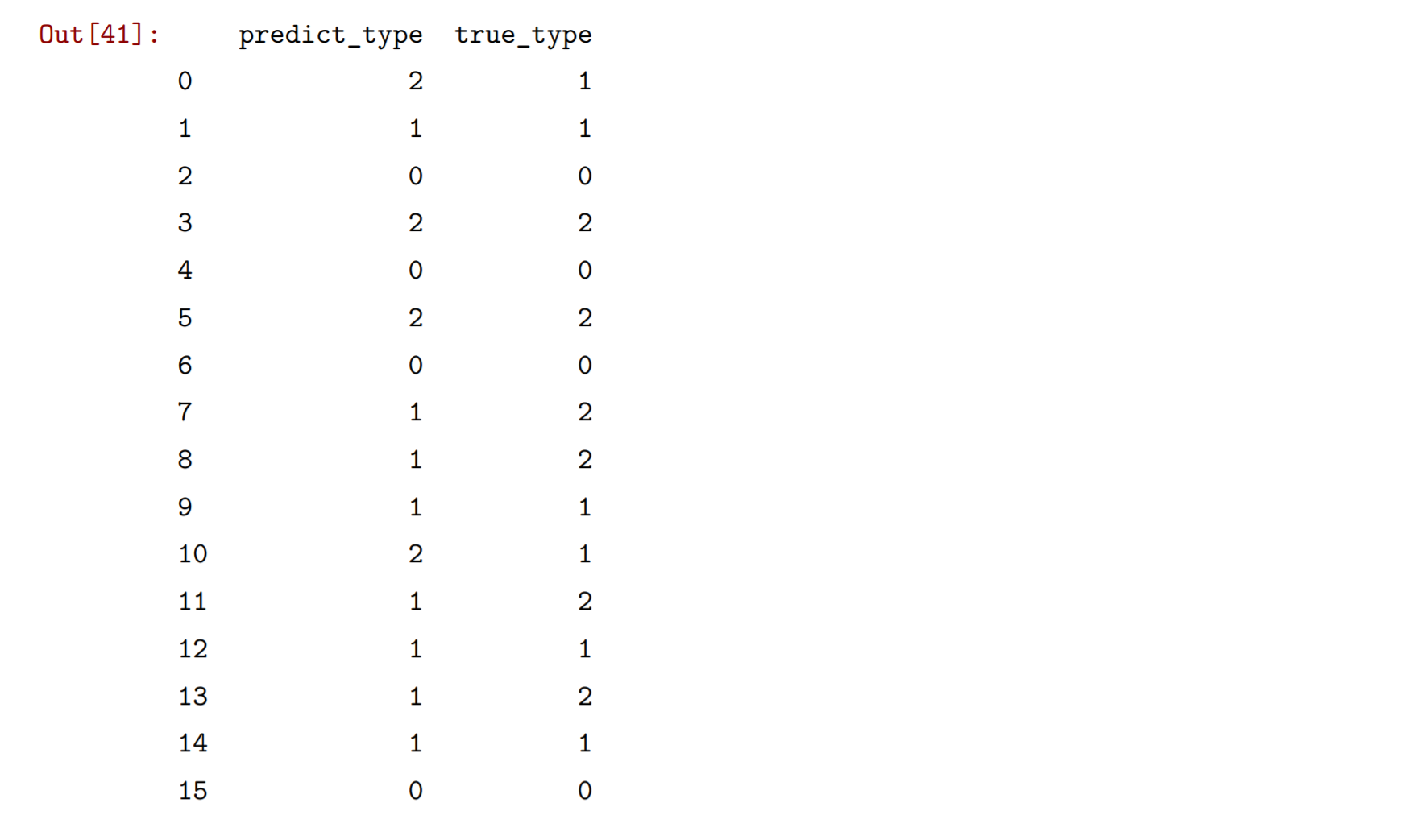
sklearn.metrics模块中的 confusion_matrix 函数将预测分类结果和真实分类结果作为参数输入可以生成混淆矩阵。
from sklearn.metrics import confusion_matrixprint(confusion_matrix(y_pred,Y_test))

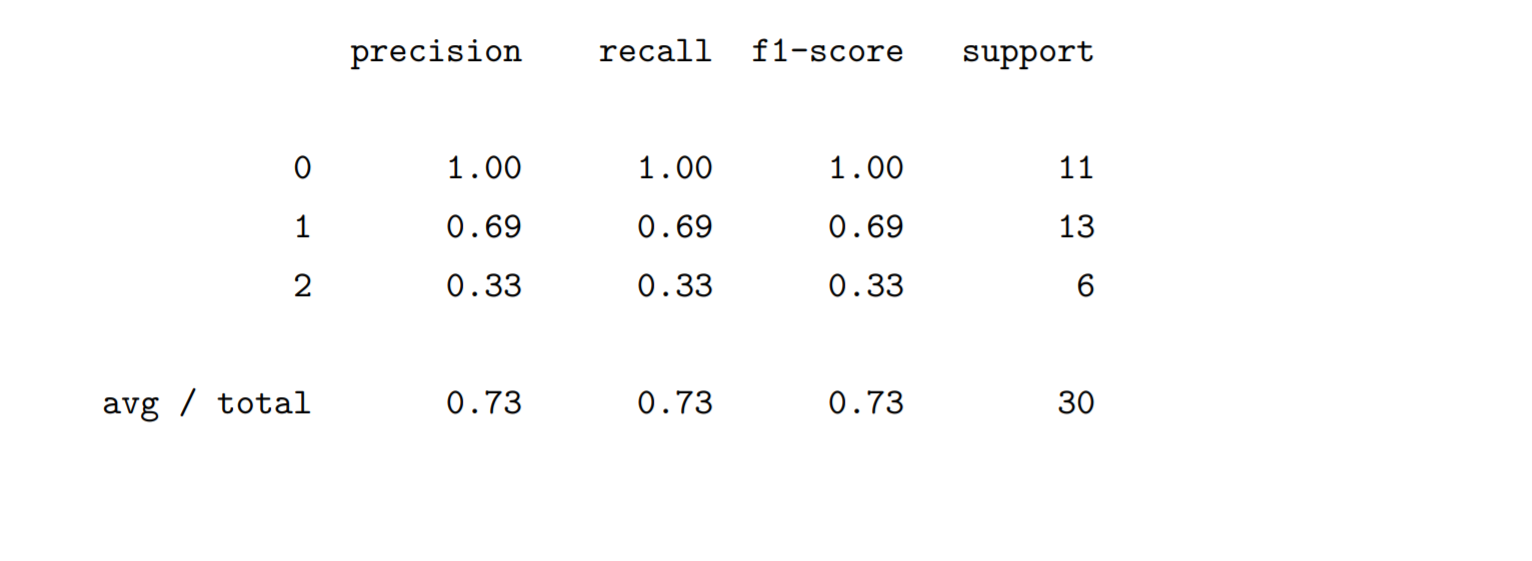
使用sklearn.metrics模块中的classification_report函数可以看到精度等评价指标。
from sklearn.metrics import classification_reportprint(classification_report(y_pred,Y_test))
分类性能评价
- 线性二分类
- 就参数的区分性能做专门评价
双样本t检验的p value作为一种评价指标,并追求小的p值。但是并不认为小的p值是一种性能指标,因为在样本容量足够大的情况下,是很容易得到一个足够小的p值。
ROC曲线(全称:接受者操作特征曲线)
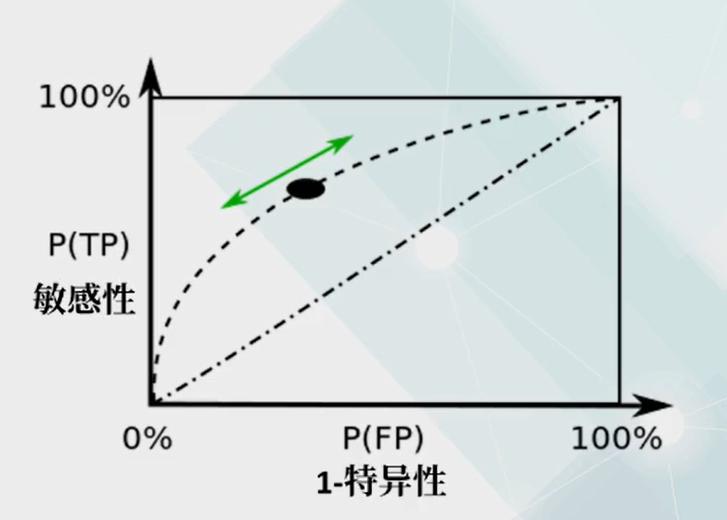
绘制方法:对同样的测试集,改变线性划分的阈值,随着阈值的改变,分类的敏感性和特异性都会随之改变,以敏感性为纵坐标,1-特异性为横坐标,将选取不同阈值的结果在图中以散点画出,散点连成的曲线就是ROC曲线。
ROC越远离45°对角线——分类性能越好
量化指标:采用曲线下面积AUC,AUCE[0,1]
- 越靠近1,参数线性区分性能越好
- 越靠近0.5,越接近随机划分
- 小于0.5,说明分类倒置
ROC优势:受两类样本数量不平衡影响较小sklearn.metrics模块中的roc_curve函数生成绘制ROC曲线所需的tpr, fpr序列,只要输入真实的分类标签和最终用于分类的square变量,可以直接说某个特征,也可以说Logistic回归中的某个概率,或者是其他分类输出用来限制二分类的峰值。函数roc_auc_score可以直接获得AUC的结果。
from sklearn.metrics import roc_curvefrom sklearn.metrics import roc_auc_scoremy_auc=[]for n in range(4):#fpr,tpr,th=roc_curve(iris.target[:100],iris.data[:,n)my_auc.append(roc_auc_score(iris.target[:100],iris.data[:100,n]))print(my_auc)#print(iris.target[:100])#print(iris.data[:100,:])
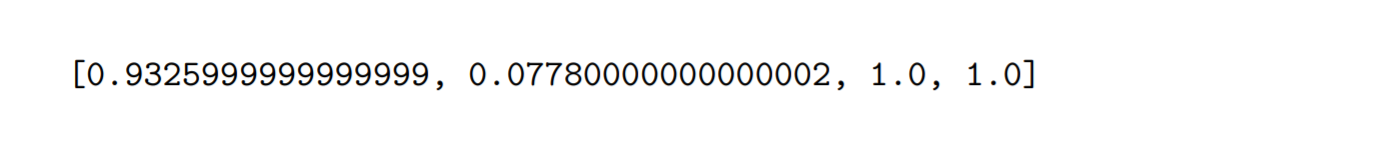
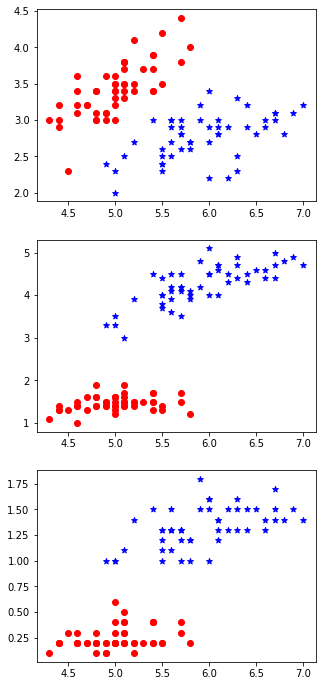
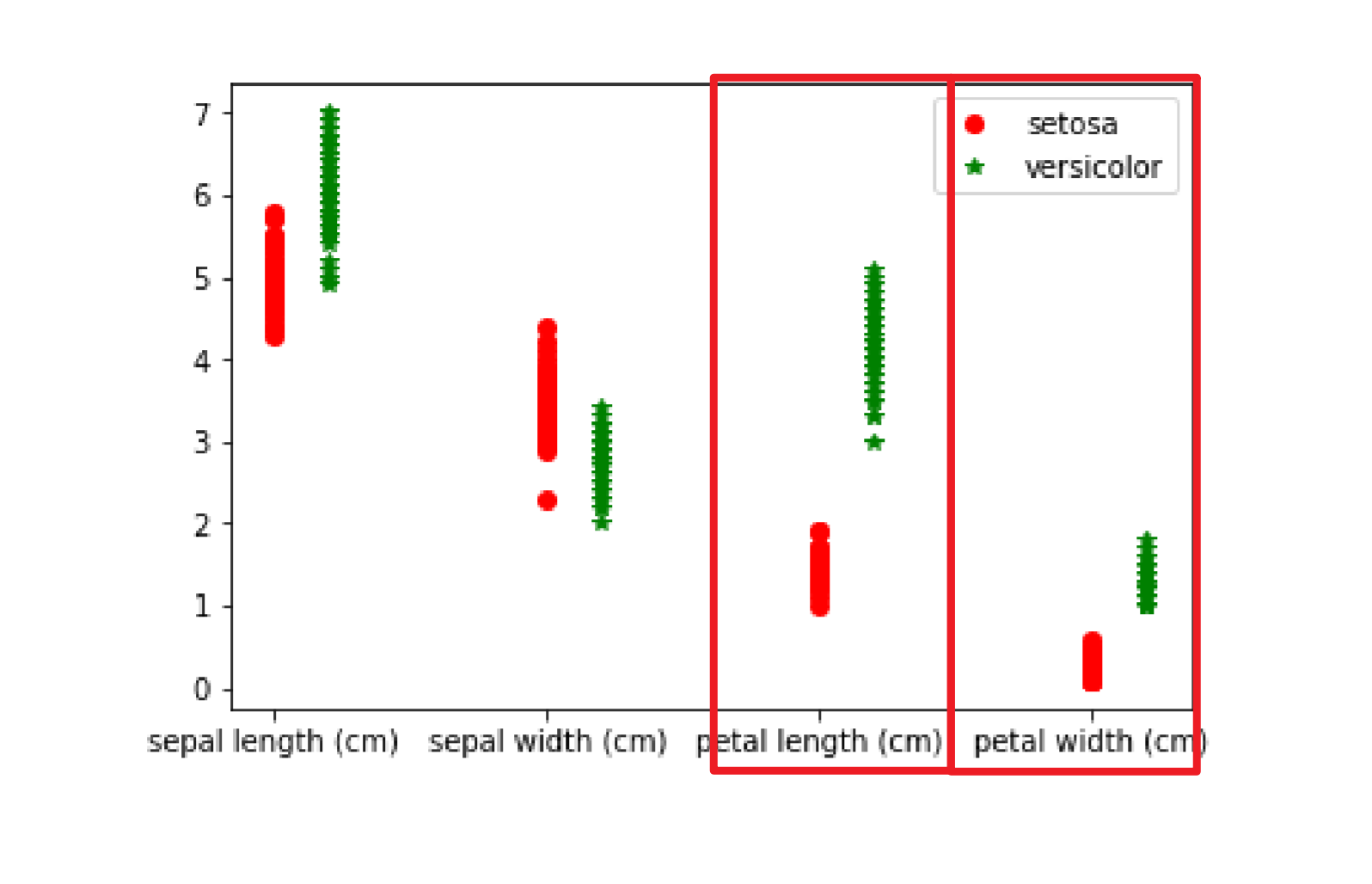
这里以鸢尾花数据的四个特征分别区分第一类和第二类。可以看到后两个参数petal length和petal width的线性区分能力都达到了1。也就是说仅单独用petal length和petal width就可以区分两种不同的鸢尾花。这和散点图的形式是完全吻合的。
plt.plot(np.ones([50,1]),iris.data[:50,0],'or')plt.plot(np.ones([50,1])+0.2,iris.data[50:100,0],'*g')plt.plot(np.ones([50,1])+1,iris.data[:50,1],'or')plt.plot(np.ones([50,1])+1.2,iris.data[50:100,1],'*g')plt.plot(np.ones([50,1])+2,iris.data[:50,2],'or')plt.plot(np.ones([50,1])+2.2,iris.data[50:100,2],'*g')plt.plot(np.ones([50,1])+3,iris.data[:50,3],'or')plt.plot(np.ones([50,1])+3.2,iris.data[50:100,3],'*g')plt.xticks([1,2,3,4],iris.feature_names)plt.legend(iris.target_names[:2])
plt.figure(figsize=(5,12))plt.subplot(3,1,1)plt.scatter(iris.data[:50,0],iris.data[:50,1],c='r',marker='o')plt.scatter(iris.data[50:100,0],iris.data[50:100,1],c='b',marker='*')plt.subplot(3,1,2)plt.scatter(iris.data[:50,0],iris.data[:50,2],c='r',marker='o')plt.scatter(iris.data[50:100,0],iris.data[50:100,2],c='b',marker='*')plt.subplot(3,1,3)plt.scatter(iris.data[:50,0],iris.data[:50,3],c='r',marker='o')plt.scatter(iris.data[50:100,0],iris.data[50:100,3],c='b',marker='*')