目的:下游分析、数据挖掘、单细胞
时间:3周 【周三、日除外】*3h
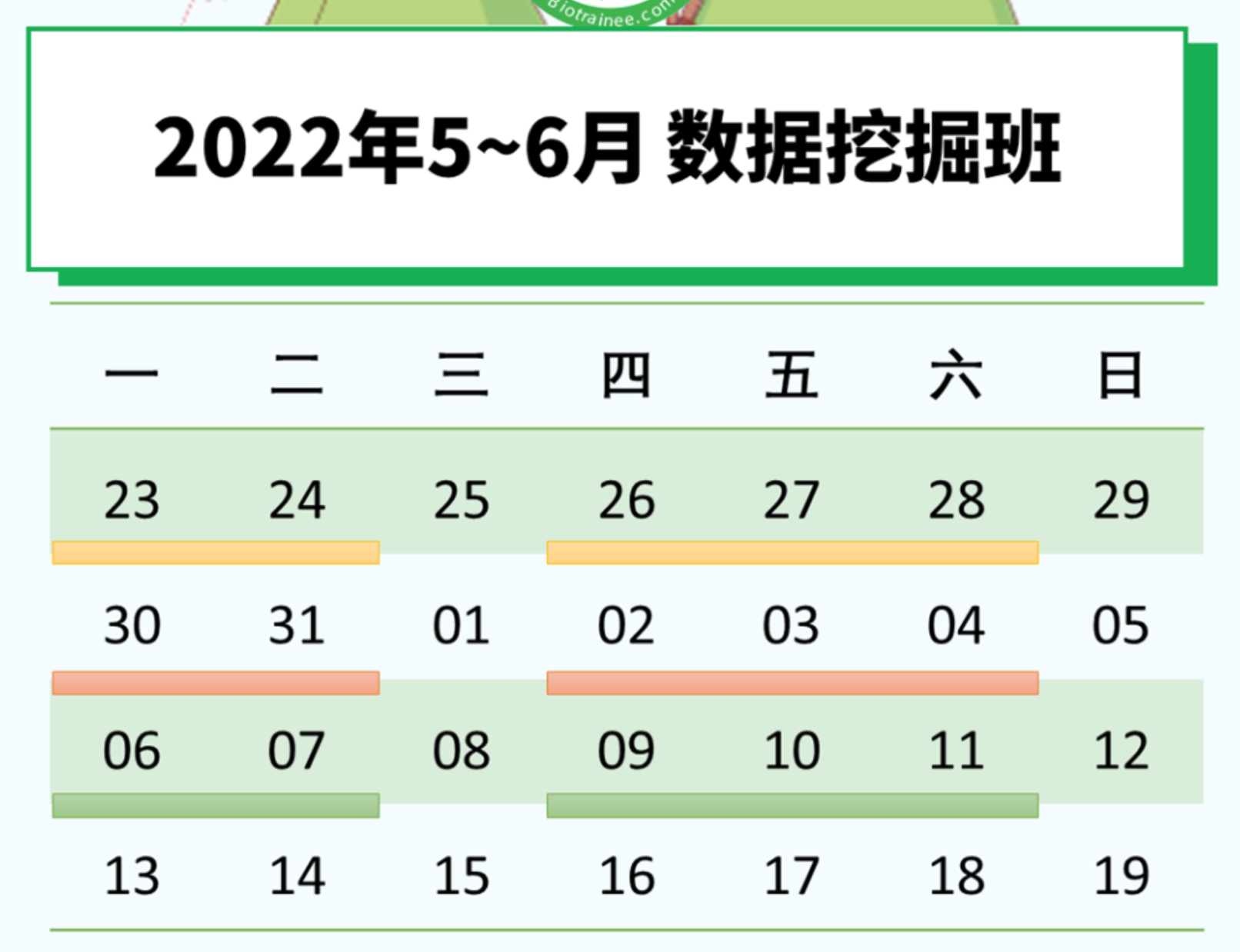
计划:
- 5.23-5.28 R
- 5.30-6.4 R+GEO
- 6.6-6.11 转录组下游分析(TCGA)+单细胞
具体内容
第一周R语言
- R语言数据类型与数据结构
- 函数与R包
- 文件读写
- R语言作图
- 拓展补充部分
第二周GEO数据挖掘
GEO数据库介绍
数据下载和质控
样本分类与芯片注释获取
差异分析和富集分析
多分组数据处理和多数据集处理
蛋白互作网络绘制
第三周Linux
服务器登录和Linux操作系统认知,基本文本文件及目录操作
文本处理三驾马车(awk,sed,grep)实战演练,基于gencode数据库human的gtf文件
conda管理生物信息学软件,环境变量
相对路径和绝对路径,通配符及参数扩展,快捷方式
批处理和脚本编程
第四周RNA-seq实战
生物信息学常见数据格式:fastq,fasta,sam,bam,vcf,gtf
转录组背景知识
转录组上游流程:文献测序数据下载到质控,比对,定量,拿到表达矩阵
转录组下游分析:3大R包走转录组表达矩阵的差异分析,富集分析,GSEA
其它NGS组学介绍
文稿内容源于公众号:生信技能树

