PPI是什么
蛋白质互作网络(Protein-Protein Interaction Networks,PPI)是由蛋白通过彼此之间的相互作用构成,来参与生物信号传递、基因表达调节、能量和物质代谢及细胞周期调控等生命过程的各个环节。网址:https://string-db.org/
界面如下: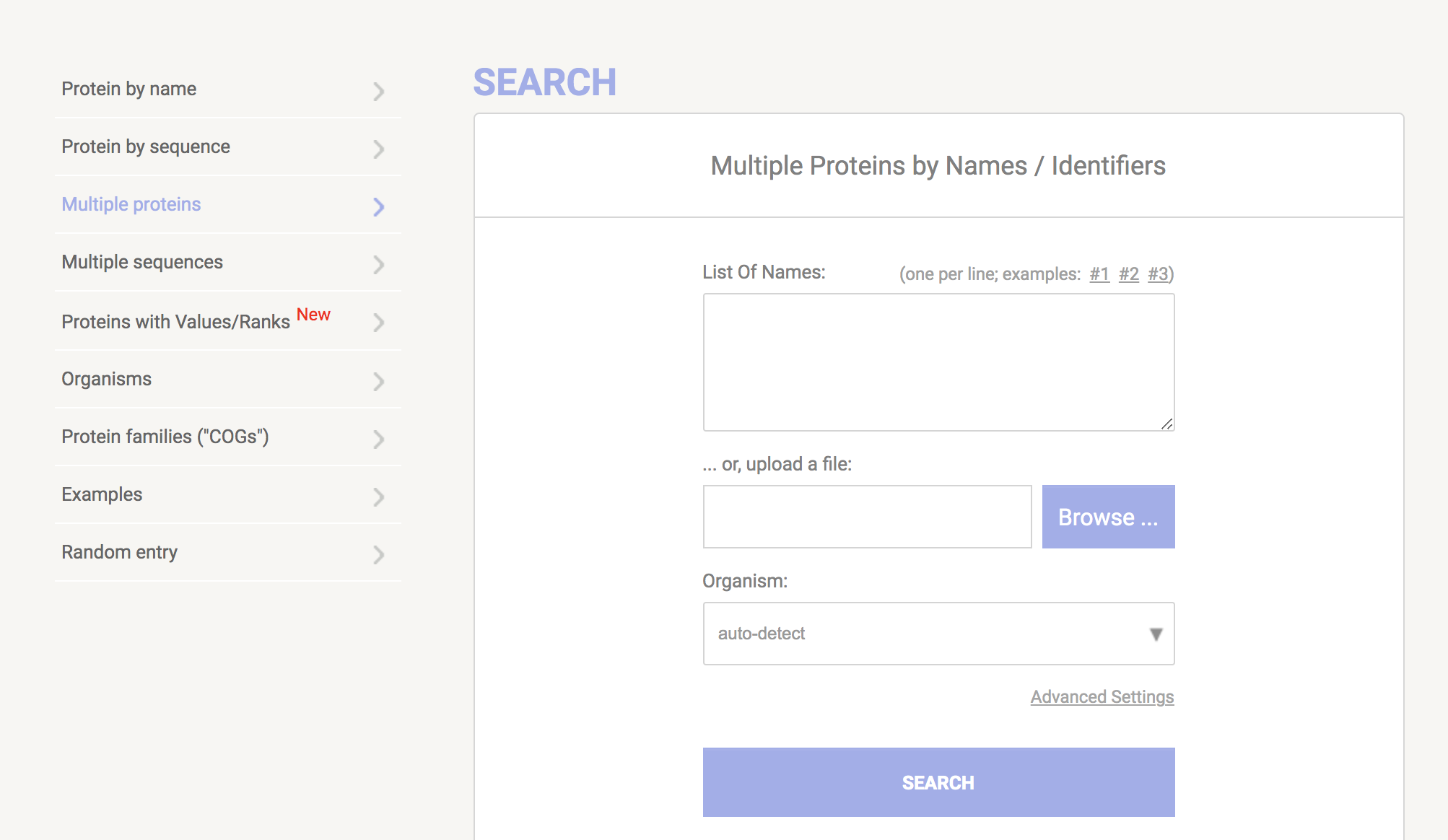
使用方法
使用基因名,选择multiple proteins,使用序列选择mulitple sequence,可以直接输入list of name也可以上传一个txt文件,然后选择物种。如果输入的是单个基因或序列则显示该基因对应的蛋白与其它蛋白的互作关系,如果输入多个基因,则显示这些基因的相互作用关系。
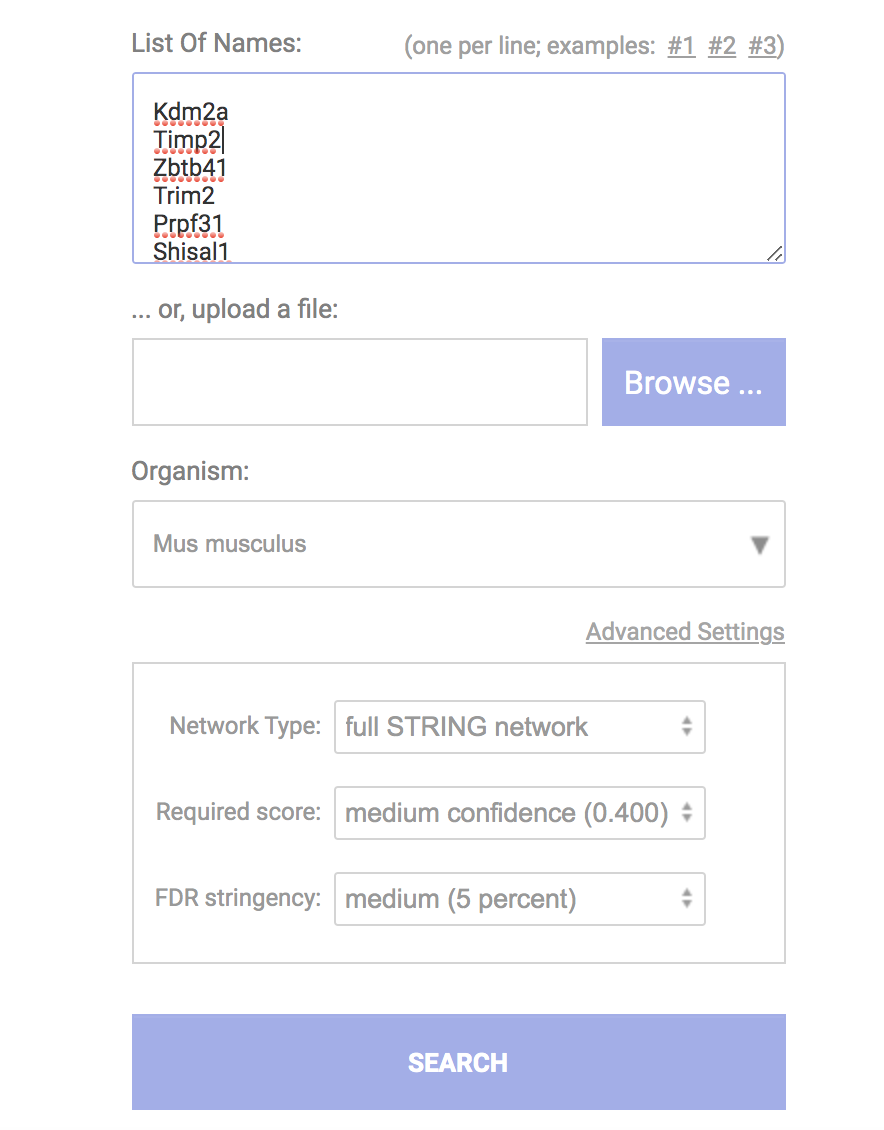
requirede score 可以根据需要调节,这里可以不管,后续仍可调节。点击searh后出现下图,点击continue。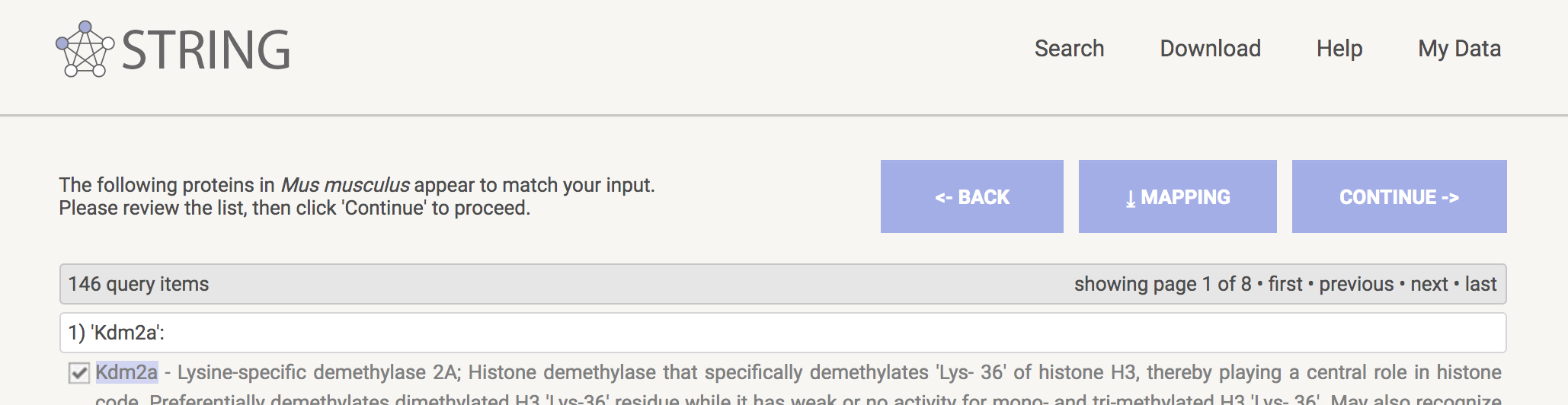
然后会出现下图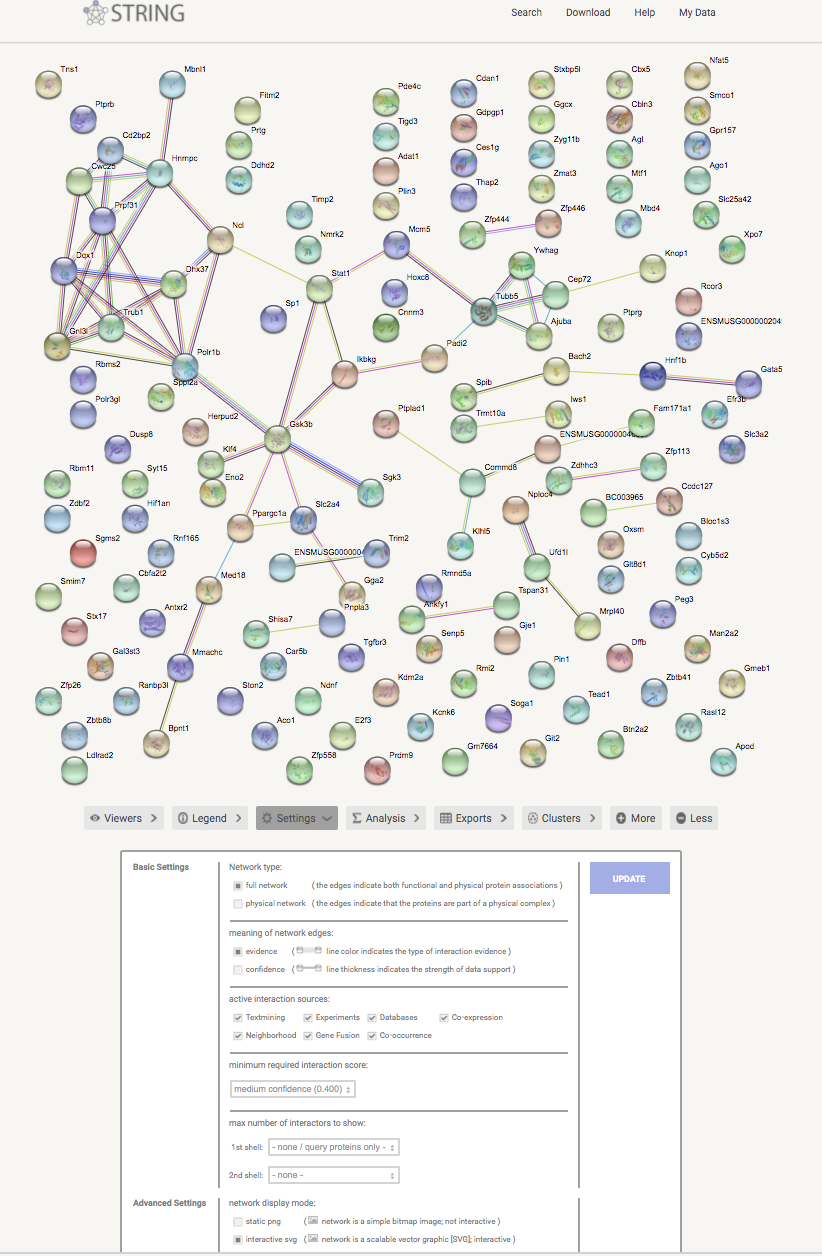
在settings里可根据需要调节置信度等。
点击:analysis,可见下图,就是一些SCI文章里的一些表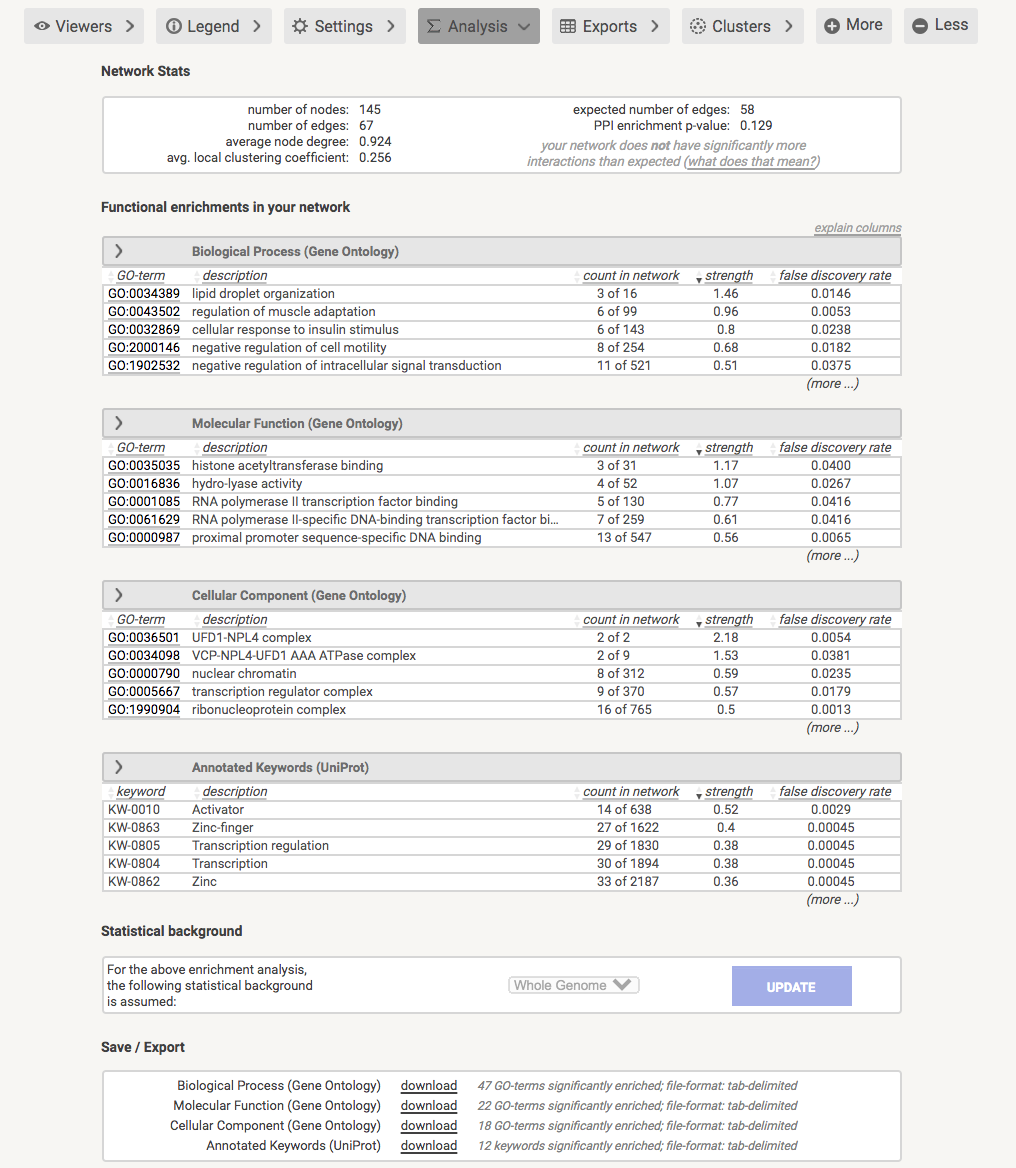
下图为一篇文章里的表格,是不是很相似,但是上图没有gene一项,这个可以点击download,在下载的表格的最后一列matching proteins in your network (labels)里有。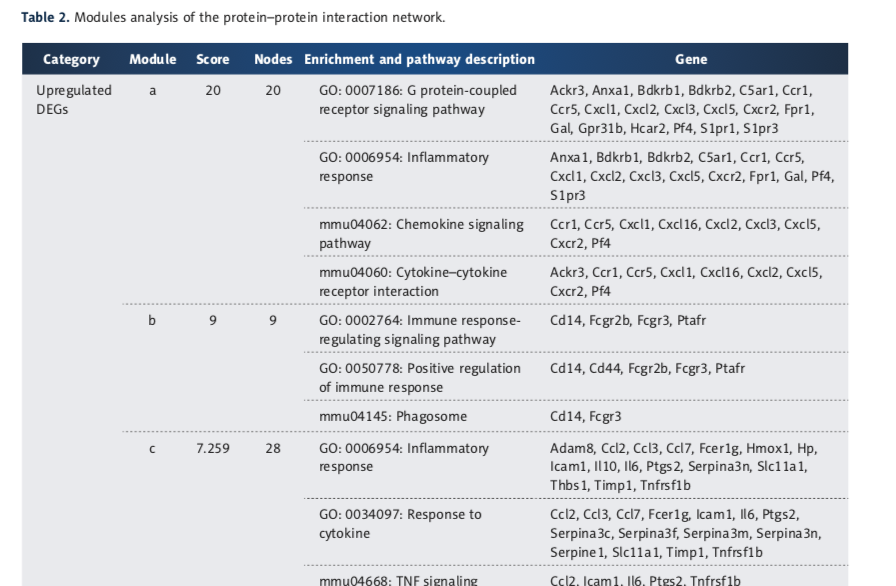
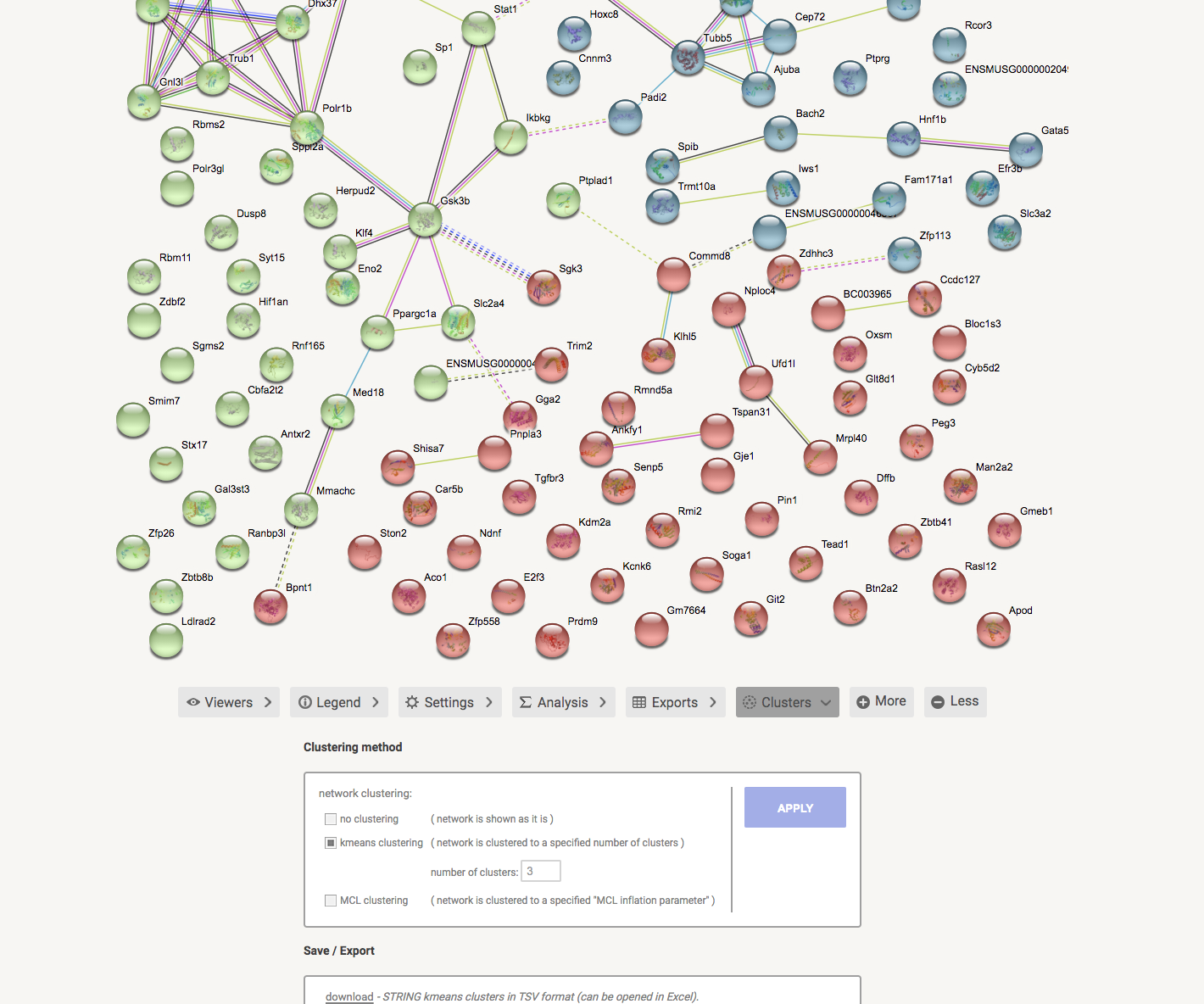
点击cluster 选择kmeans clustering可以聚类,并将数据导出到cytoscape继续分析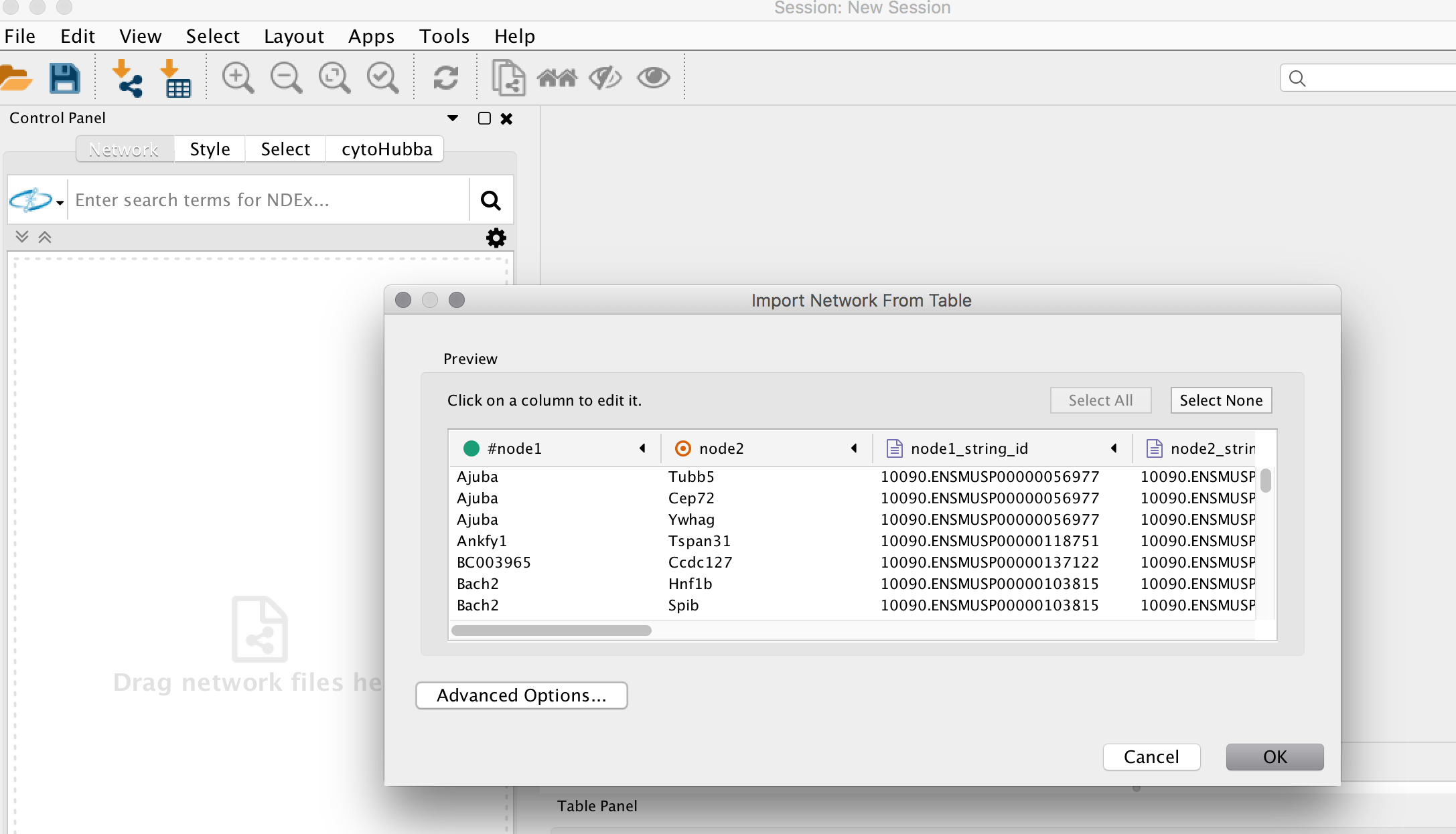
详细可参考STRING数据库的蛋白质相互作用(PPI)网络分析
cytoscape的使用
参考 :一网打尽:cytoscape详细教程
Cytoscape 是一个专注于开源网络可视化和分析的软件。它的核心是提供基础的功能布局和查询网络,并依据基本的数据的结合成可视化网络。
Cytoscape 源自系统生物学,用于将生物分子交互网络与高通量基因表达数据和其他的分子状态信息整合在一起,其最强大的功能还是用于大规模蛋白质-蛋白质相互作用、蛋白质-DNA和遗传交互作用的分析。
通过Cytoscape,可以在可视化的环境下将这些生物网络跟基因表达、基因型等各种分子状态信息整合在一起,还能将这些网络跟功能注释数据库链接在一起。
Cytoscape 的核心是网络,简单的网络图包括节点(node)和边(edge),每个节点可以是基因、miNRA或蛋白质等等;节点与节点之间的连接 (edge) 代表着这些节点之间的相互作用,包括蛋白与蛋白相互作用(pp),DNA与蛋白相互作用(pd)等。
导入数据
- 第二排第三个按钮 import network from table
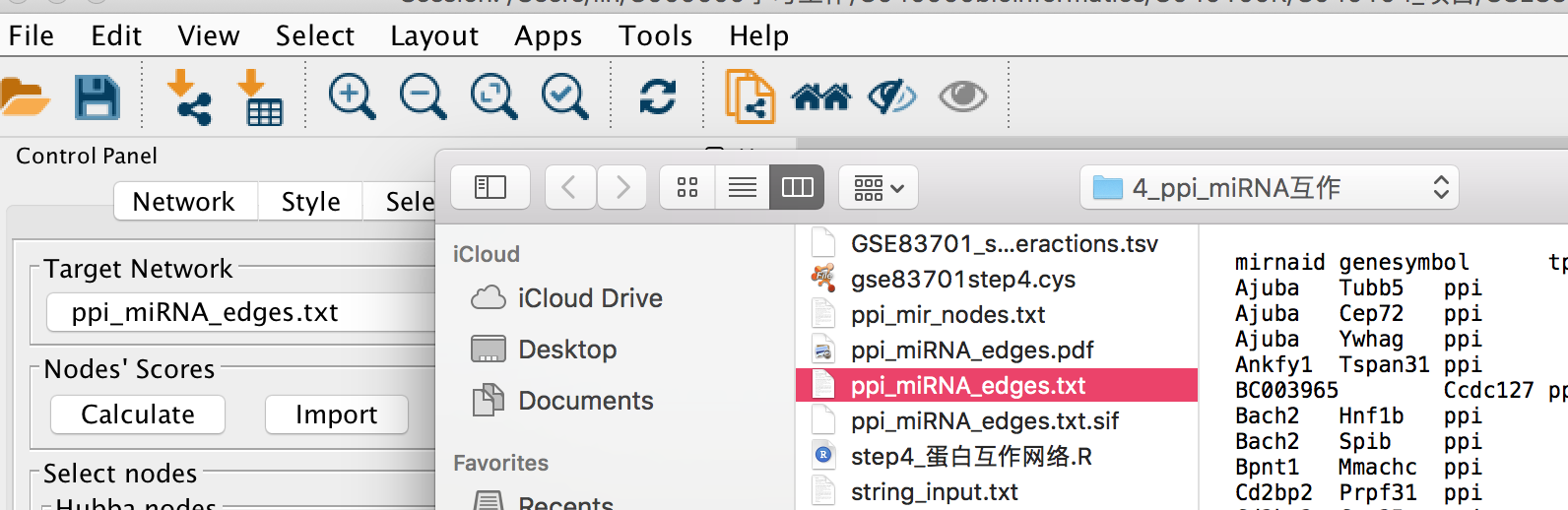
导入edgs文件,内容如上图,再如下图设置source node, target node,key。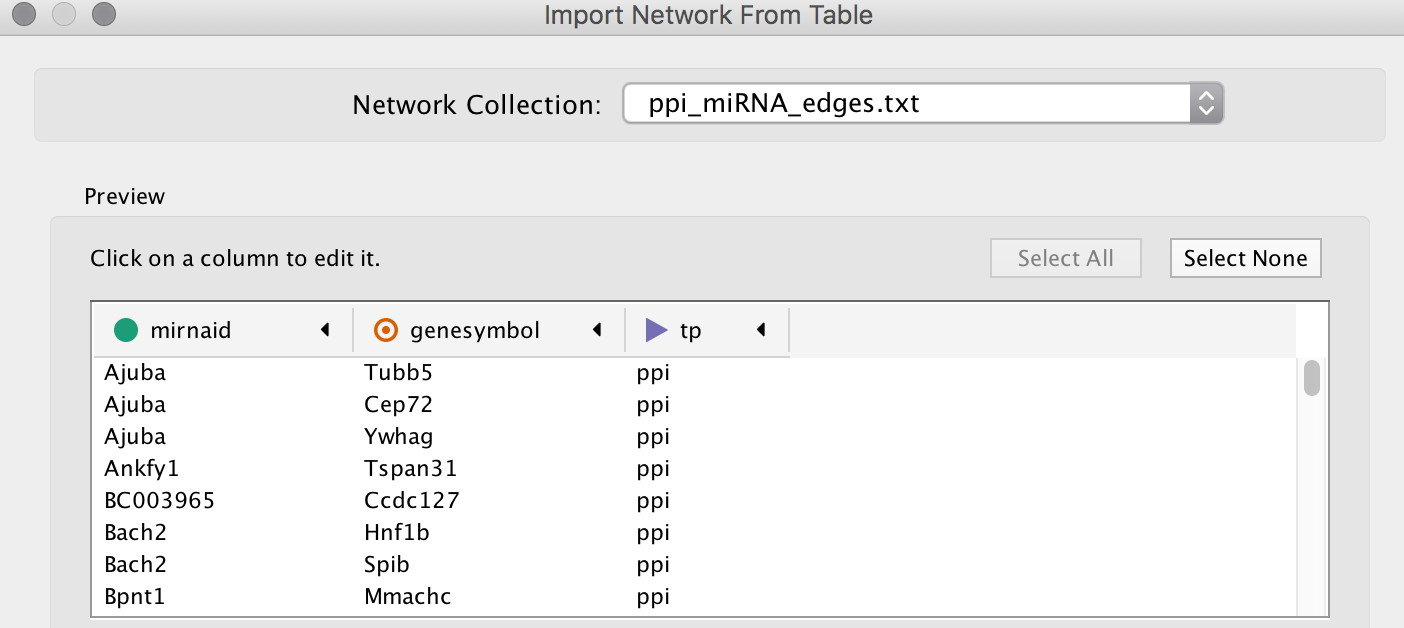
- 第二排第四个按钮 import table from file,导入nodes文件,如下
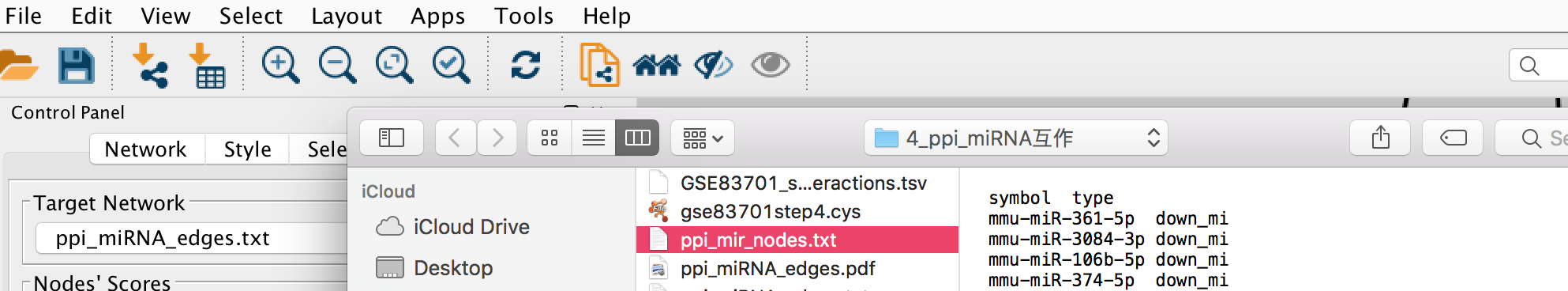
设置如下:选择第一次导入的edges,并将下面的symbol设置为key,type设置为attribute,点击小三角即可。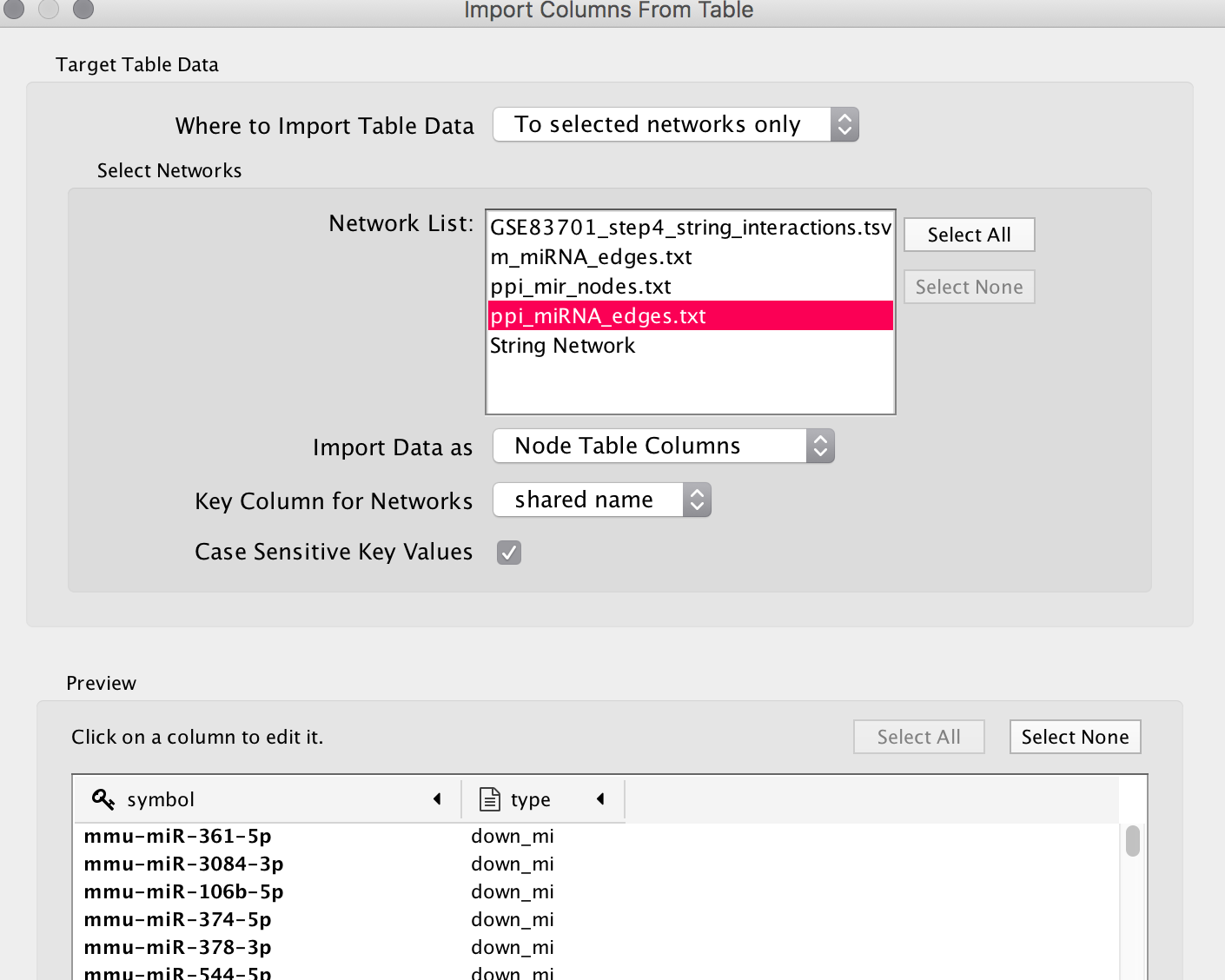
后续的设置可参考 :一网打尽:cytoscape详细教程。

