查看磁盘空间
df -hl
软件类型
一般来说,下载的linux 软件分成两类,1)二进制软件;2)源码软件。
二进制软件
一般来说,二进制软件已经是预编译的版本了,这类软件对新手非常的友好,也是最方便的,解压就可以直接使用了。(zip, gz, bzip2等等压缩类型)
源码软件
相比起二进制软件,这类软件是作者直接分享了它们的源代码,因此我们需要将它们通过编译器编译它们,从而让计算机读懂它们。
一般来说有三个步骤。
1)配置;2)编译;3)安装。
1)配置
配置部分一般的代码为
./configure --prefix= 安装路径
这个安装路径是用于指定软件编译后的可执行文件所放的目录,而默认的路径需要有root权限。
2)编译
一般通过make 命令对文件进行编译,但经常会出现库文件的缺失,比如bwa的zlib。通常来说这也是其比较头疼的地方。
3)安装
make install,直接安装到相应目录就好啦。
用conda 做你的软件管家
和苹果的app store, 手机的软件管家一样,linux 也提供了conda(miniconda)这样一个软件中心。
我们可以在里面找到各种工具,实现快速的一键式安装,而且可以通过设定不同的环境,使各自依赖的环境相互隔离,不至于使内部安装环境乱成一团。通过conda install便可实现各种软件的安装。
安装conda
下载
20-12-12 目前最新的镜像版本:
https://mirrors.tuna.tsinghua.edu.cn/anaconda/miniconda/Miniconda3-py38_4.9.2-Linux-x86_64.sh
通过wget 对应相应的下载地址,直接通过linux 下载对应下载包。
安装
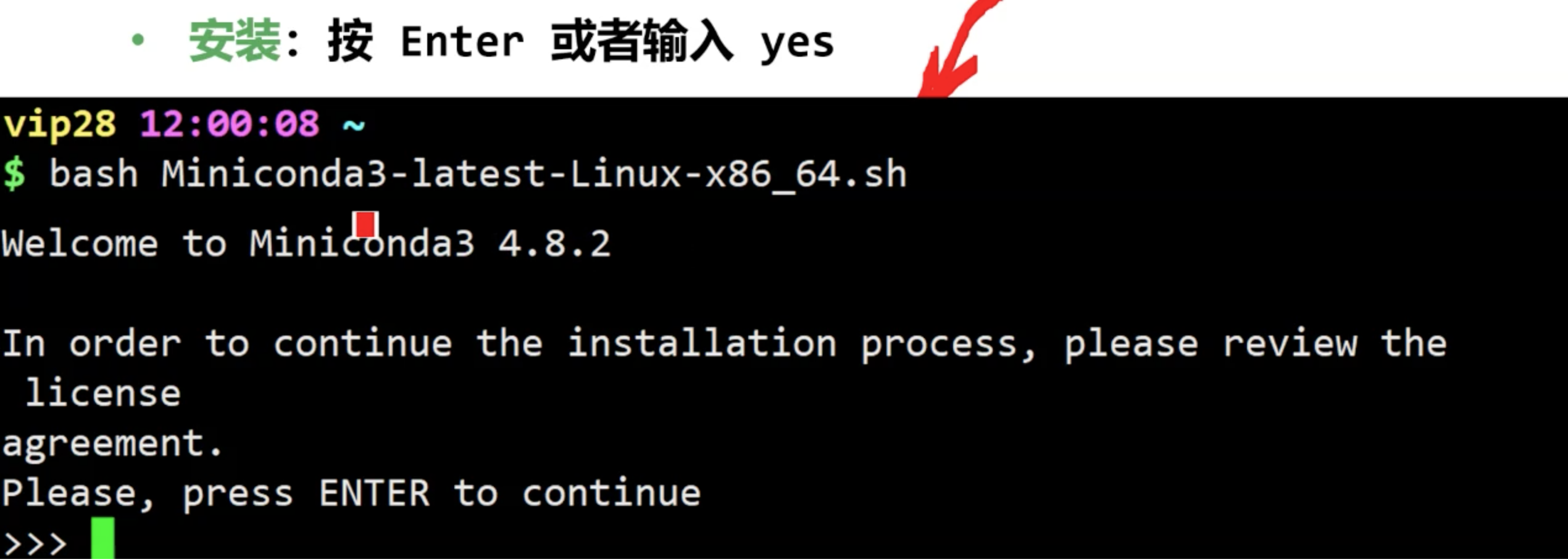
一路 yes 到底
激活环境
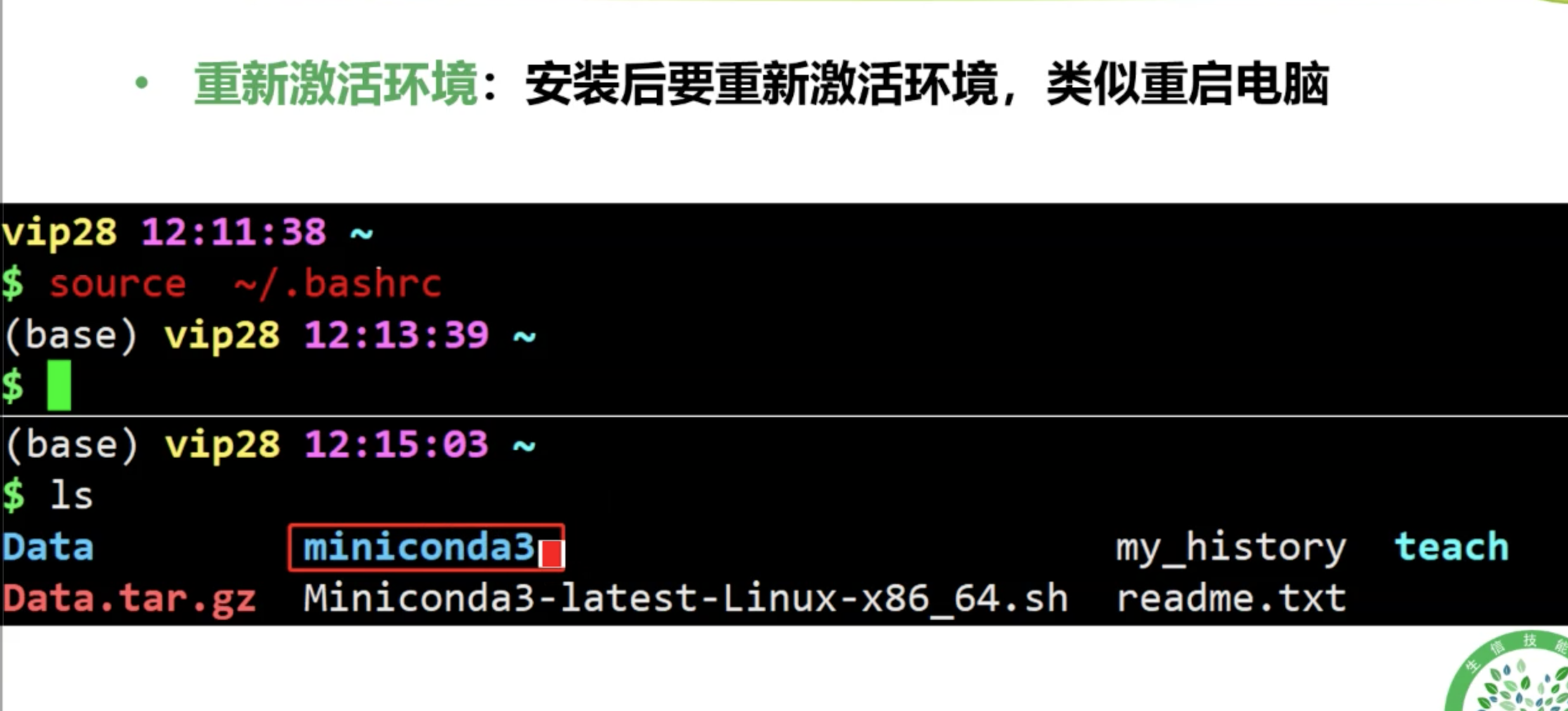
如果不希望命令行默认进入conda base 环境,可以输入:
conda config --set auto_activate_base false
source ~/.bashrc #再重新激活一下bash
配置镜像
需要注意的是,conda 从镜像下载内容存在先后顺序,它会优先检索后添加的镜像。
conda config --add channels r
conda config --add channels conda-forge
conda config --add channels bioconda
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/free/
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/main/
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud/conda-forge/
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud/bioconda/
这里再推荐一波北外的镜像,实际体验优于清华源:
conda config --add channels r
conda config --add channels conda-forge
conda config --add channels bioconda
conda config --add channels https://mirrors.bfsu.edu.cn/anaconda/cloud/bioconda/
conda config --add channels https://mirrors.bfsu.edu.cn/anaconda/cloud/conda-forge/
conda config --add channels https://mirrors.bfsu.edu.cn/anaconda/pkgs/free/
conda config --add channels https://mirrors.bfsu.edu.cn/anaconda/pkgs/main/

其他conda 选项及镜像源:
conda config --show channels # 显示所有镜像通道路径命令
conda config --remove-key channels #清除添加的镜像源
# 当然我们也可以通过cat查看
cat ~/.condarc
#添加中科大镜像源命令:
conda config --add channels https://mirrors.ustc.edu.cn/anaconda/pkgs/main/
conda config --add channels https://mirrors.ustc.edu.cn/anaconda/pkgs/free/
conda config --add channels https://mirrors.ustc.edu.cn/anaconda/cloud/bioconda/
conda config --add channels y
#添加阿里镜像源命令:
conda config --add channels https://mirrors.aliyun.com/pypi/simple/
使用conda
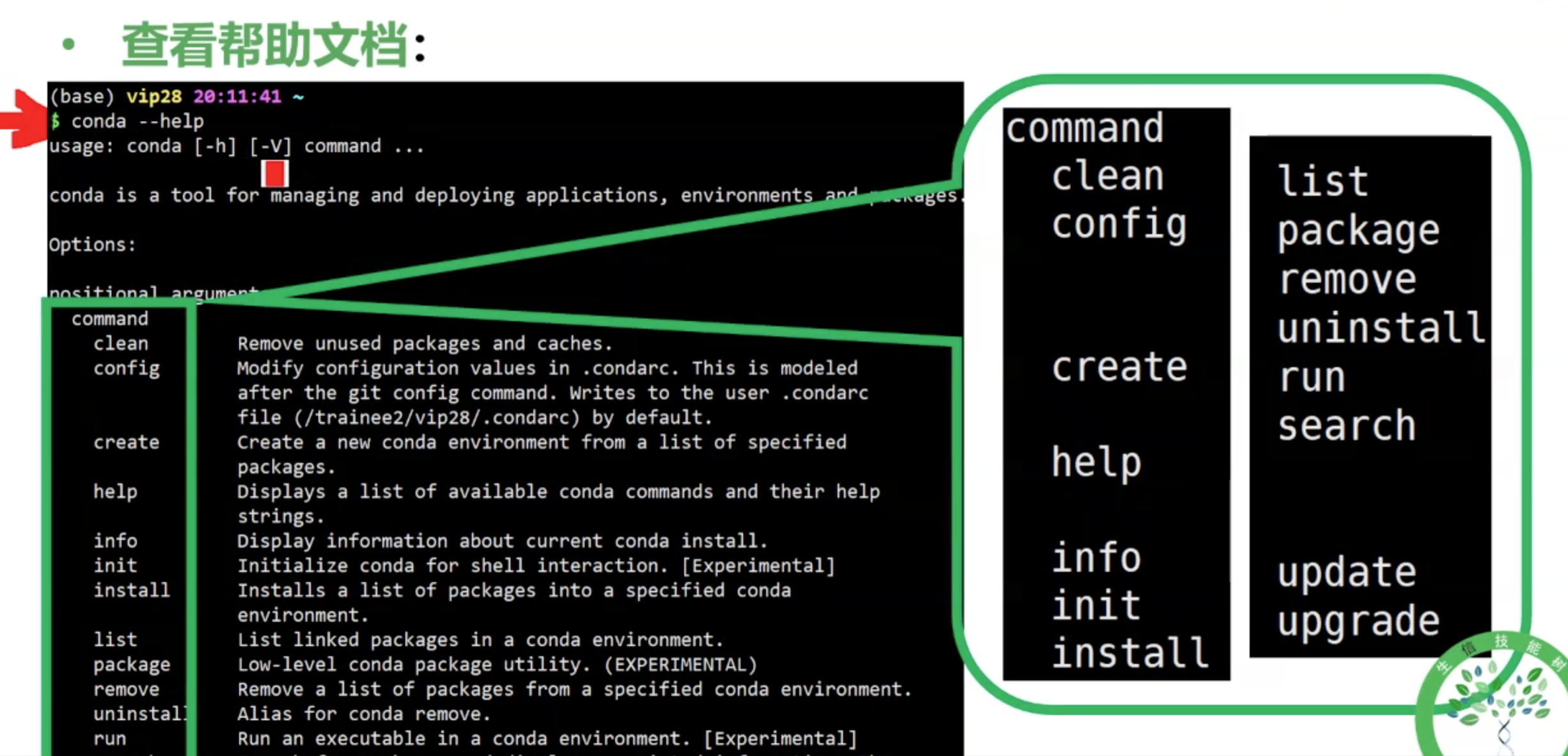
帮助文档
创建环境
创建环境的目的,是为了让依赖不同环境要求的软件可以放置在相同的环境内,以防止不同环境要求的软件因为环境变化产生冲突。
# 创建名为rna的软件环境来安装转录组学分析的生物信息学软件
conda create -y -n rna python=3
# 创建小环境成功,并成功安装python3版本
# 每建立一个小环境,安装一个python=3的软件作为依赖
# -n 指定环境名,-y 后面可以跳过确认的选项
# 查看当前conda环境
conda info -e
# 每次运行前,激活创建的小环境rna
conda activate rna
# 退出小环境
conda deactivate
conda 的其他命令

用conda 安装软件
前言
这里最好已经创建了相应的环境,并进入了相关的环境,以防止造成环境污染。
另外软件的原名可能和conda 中的名字不一样。
如 sratoolkit 与 sra-tools 。
## 小知识点
# 下载安装软件之前先搜索是否存在
http://bioconda.github.io/conda-recipe_index.html
网页搜索:conda ascp
安装
关于conda 的安装相关选项可可以参考:
https://docs.conda.io/projects/conda/en/latest/commands/install.html
# 安装 fastqc 软件
conda install fastqc
# 调出帮助文档
fastqc --help
# 可以一次安装多个软件
conda install -y sra-tools trim-galore hisat2 subread multiqc samtools salmon fastp
## 不是通过软件名来调用帮助文档,而是软件的命令
# sra-tools
prefetch --help
fastq-dump --help
which prefetch
# trim-galore
trim_galore --help
# hisat2
hisat2 -h
# subread
featureCounts
# multiqc
multiqc --help
# samtools
samtools
which samtools
# salmon
salmon
# fastp
fastp --help
其他软件安装方式
1)二进制文件安装
前言
当然我们也可以不用conda来安装软件。
创建并进入文件夹
mkdir bowtie2 && cd bowtie2
版本
软件安装提供了各种版本,因此需要注意合适的版本选择。
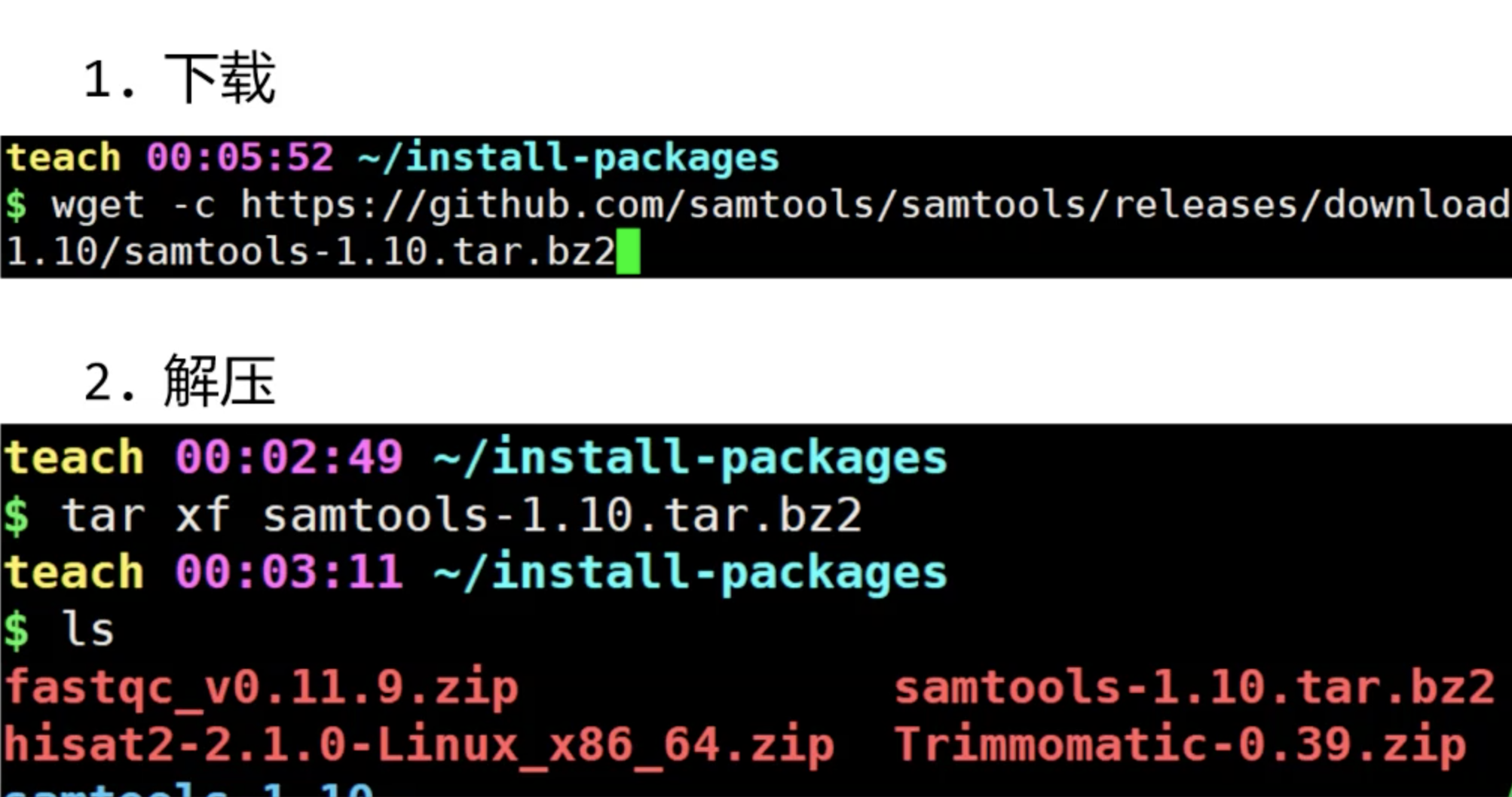
下载
可以使用wget 下载
首先进入安装目录下
mkdir xxx && cd xxx
接着安装,直接找到文件的下载地址通过wget下载即可。
wget 下载地址
通过ftp 上传文件到服务器
由于我通过wget 方式文件下载速度过于感人,我选择先将它下载至电脑上,再上传到服务器中。
- 通过ftp 连接
sftp root@your_sever_id
- 登陆完成后,通过put 上传
put local-file [remote-file]
更多内容参考
https://blog.csdn.net/q1596948728/article/details/23859879
通过FileZilla 连接远程服务器
FileZilla是一个FTP 连接服务器的软件,提供linux、mac、win 等各个平台
https://filezilla-project.org/
注意一般服务器只提供SFTP 的连接方式。
设定好服务器公网ID,连接,输入用户密码就可以登录了。
你可以直接直接将本地服务器的内容拖拽进去。
解压与使用
https://sourceforge.net/projects/bowtie-bio/files/bowtie2/2.2.9/bowtie2-2.2.9-linux-x86_64.zip
我是通过filezilla 下载了bowtie(一个短序列比对工具),并将它存放在了biosofts/bowtie2目录下。
先来解压
下载的文件是bowtie2-2.2.9-linux-x86_64.zip,直接用unzip 就好。
unzip bowtie2-2.2.9-linux-x86_64.zip
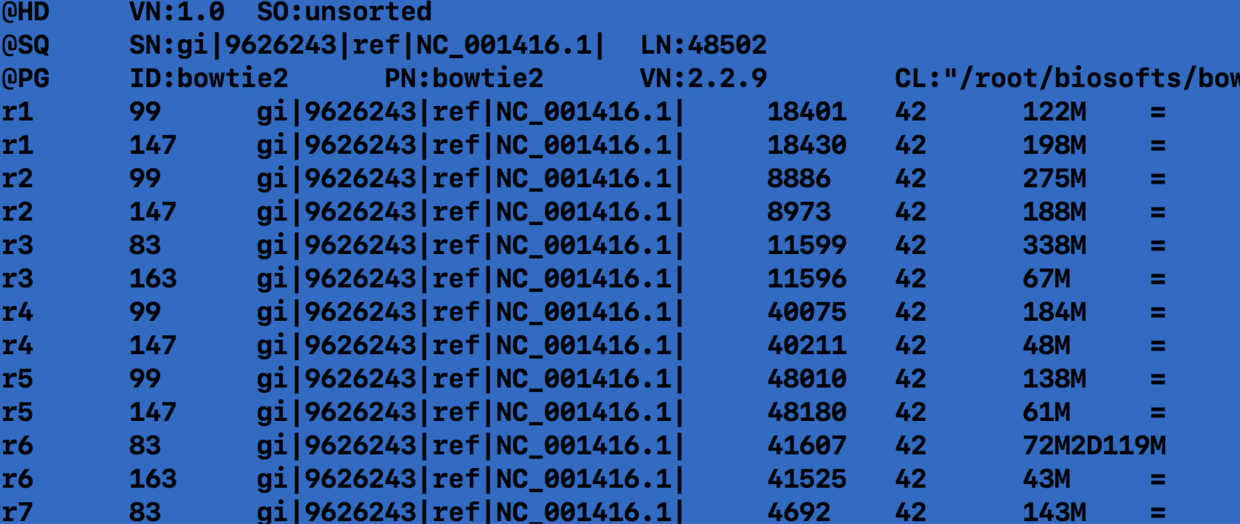
查看文件文档

可以看到,bowtie2 文件用绿色标记出来了,这表示它是一个可执行文件,因此我们可以通过输入绝对路径的方式调用它(没有添加环境),通过调用,我们可以看到bowtie2 的使用说明。
/root/biosofts/bowtie2/bowtie2-2.2.9/bowtie2 # 我的路径
# 如果不把bowtie2 文件添加到PATH中,则需要通过相对路径或绝对路径调用软件
阅读帮助文件,发现有许许多多的参数。
使用文件
添加到PATH
PATH=$PATH:/root/biosofts/bowtie2/bowtie2-2.2.9/
查看范例文件
一般来说文件里会附带一个example,可以作为练习。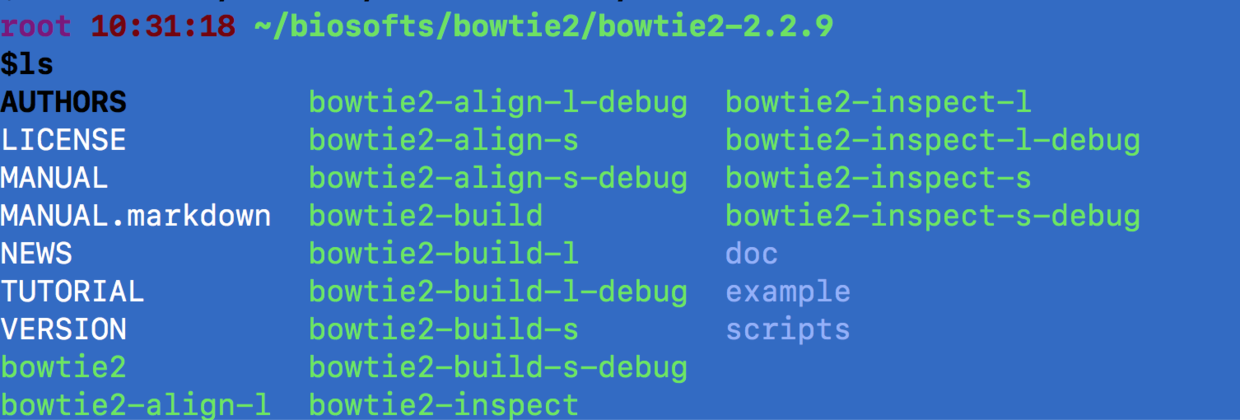
果不其然
可以查看一些文件的大小等信息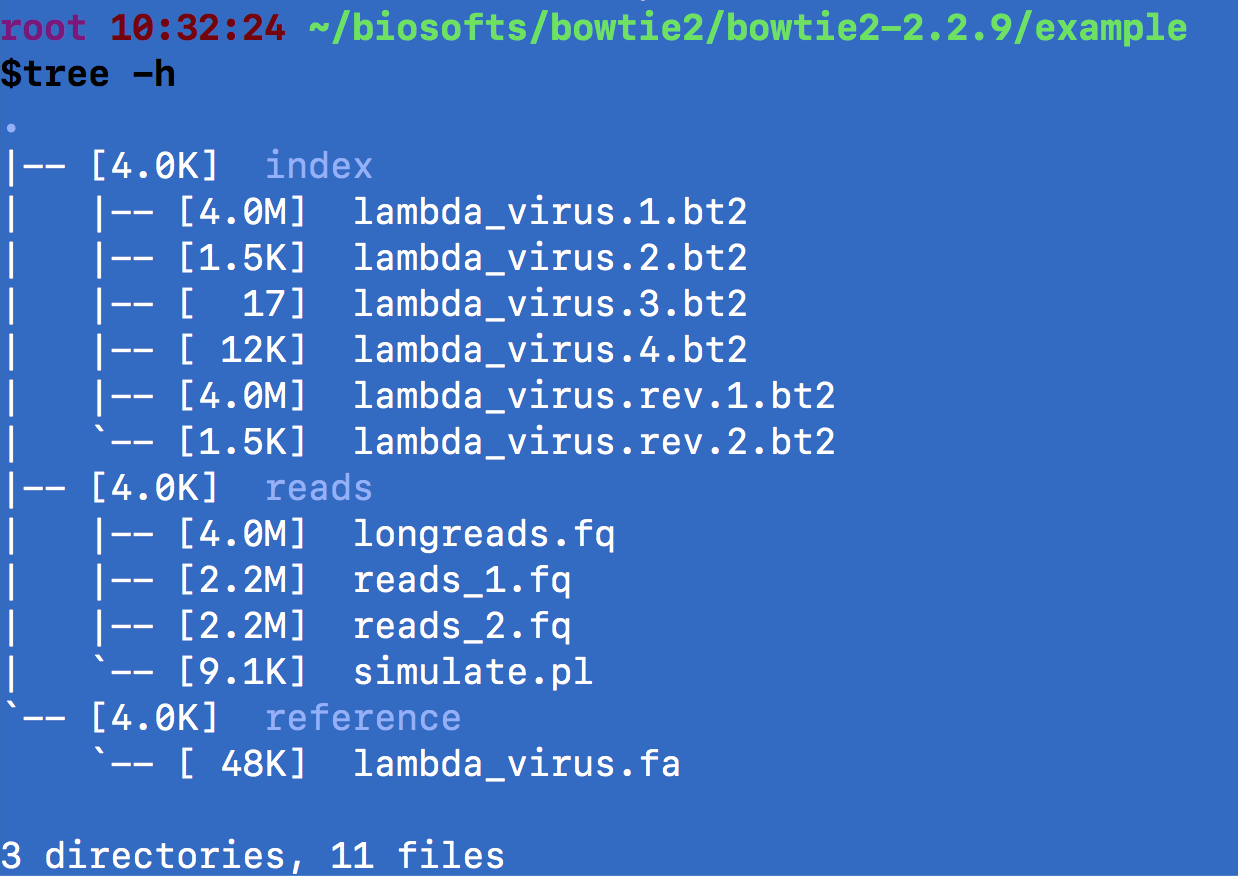
其中可以先查看其中一个reads 文件。
$less reads/reads_1.fq
通过less 可以上下滚动阅读文本。
我截取其中一个序列。至于如何解读fastq 文件,你可以参考我的笔记,
https://www.yuque.com/mugpeng/nwmnq7/ug9fg8#36bd89cf
@r1
CCAGCCGGACTTCAGGCCTGCCATCCAGTTCCCGCGAAGCTGGTCTTCAGCCGCCCAGGTCTTTTTCTGCTCTGACACGACGTTATTCAGCGCCAGCGGATTATCGCCATACTGTTCCTTCAGGCGCTGTTCCGTGGCTTCCCGTTCTGCCTGCCGGTCAGTCAGCCCCCGGCTTTTCGCATCAATGGCGGCCC
+
7%&%E,3@->*/4>0):4)17/4A=H<3!72"$97HDB(*66B(,&$+41&A,8+=,7/216,C=2#B6!>)"?&D44/6+@))30A91BA&=@(%0&-E.1-=959!B,",,#FA94,7.B+-)2@5H;6E**#EA&;"3F920A>1:<:8F-1.<A4.-/HE$02%1>;0HA6.*@(=37<:.E93+;52+8
bowtie 的使用规范
首先可以看看版本和帮助界面。
bowtie2 --help ## 帮助文件,且我已经设定了bowtie 的alias。
bowtie2 --version ## 查看版本。
bowtie2 是一个用于短序列比对的工具。
http://bowtie-bio.sourceforge.net/index.shtml
Usage:
bowtie2 [options]* -x <bt2-idx> {-1 <m1> -2 <m2> | -U <r>} [-S <sam>]
<bt2-idx> Index filename prefix (minus trailing .X.bt2).
NOTE: Bowtie 1 and Bowtie 2 indexes are not compatible.
<m1> Files with #1 mates, paired with files in <m2>.
Could be gzip'ed (extension: .gz) or bzip2'ed (extension: .bz2).
<m2> Files with #2 mates, paired with files in <m1>.
Could be gzip'ed (extension: .gz) or bzip2'ed (extension: .bz2).
<r> Files with unpaired reads.
Could be gzip'ed (extension: .gz) or bzip2'ed (extension: .bz2).
<sam> File for SAM output (default: stdout)
<m1>, <m2>, <r> can be comma-separated lists (no whitespace) and can be
specified many times. E.g. '-U file1.fq,file2.fq -U file3.fq'.
Options (defaults in parentheses):
Input:
-q query input files are FASTQ .fq/.fastq (default)
--qseq query input files are in Illumina's qseq format
-f query input files are (multi-)FASTA .fa/.mfa
-r query input files are raw one-sequence-per-line
-c <m1>, <m2>, <r> are sequences themselves, not files
-s/--skip <int> skip the first <int> reads/pairs in the input (none)
-u/--upto <int> stop after first <int> reads/pairs (no limit)
-5/--trim5 <int> trim <int> bases from 5'/left end of reads (0)
-3/--trim3 <int> trim <int> bases from 3'/right end of reads (0)
--phred33 qualities are Phred+33 (default)
--phred64 qualities are Phred+64
--int-quals qualities encoded as space-delimited integers

使用bowtie2构建索引
$bowtie2-build reference/lambda_virus.fa temp

我们可以通过md5sum 比较一下生成的index 和默认的index 的文件差距。md5sum 是一套linux 下的算法,可以被用来验证网络文件传输的完整性,防止被他们篡改。MD5 全称是报文摘要算法(Message-Digest Algorithm 5)。
$md5sum index/lambda_virus.1.bt2
9c033d93b13e5106347b4d39ccf33af8 index/lambda_virus.1.bt2
$md5sum temp.1.bt2
9160769174cc604412693aff33b76f88 temp.1.bt2
其实二者还是有区别的。
进行序列比对
完成索引的构建后,便可以按照usage 的方式进行序列比对。
$bowtie2 -x temp -1 reads/reads_1.fq -2 reads/reads_2.fq -S temp.sam
查看比对结果
less -S temp.sam
2)源代码安装
下载并解压
编译三部曲
构建—编译—安装
后续
后面的操作就和二进制文件一样了,将文件添加到PATH,就可以直接调用它了。
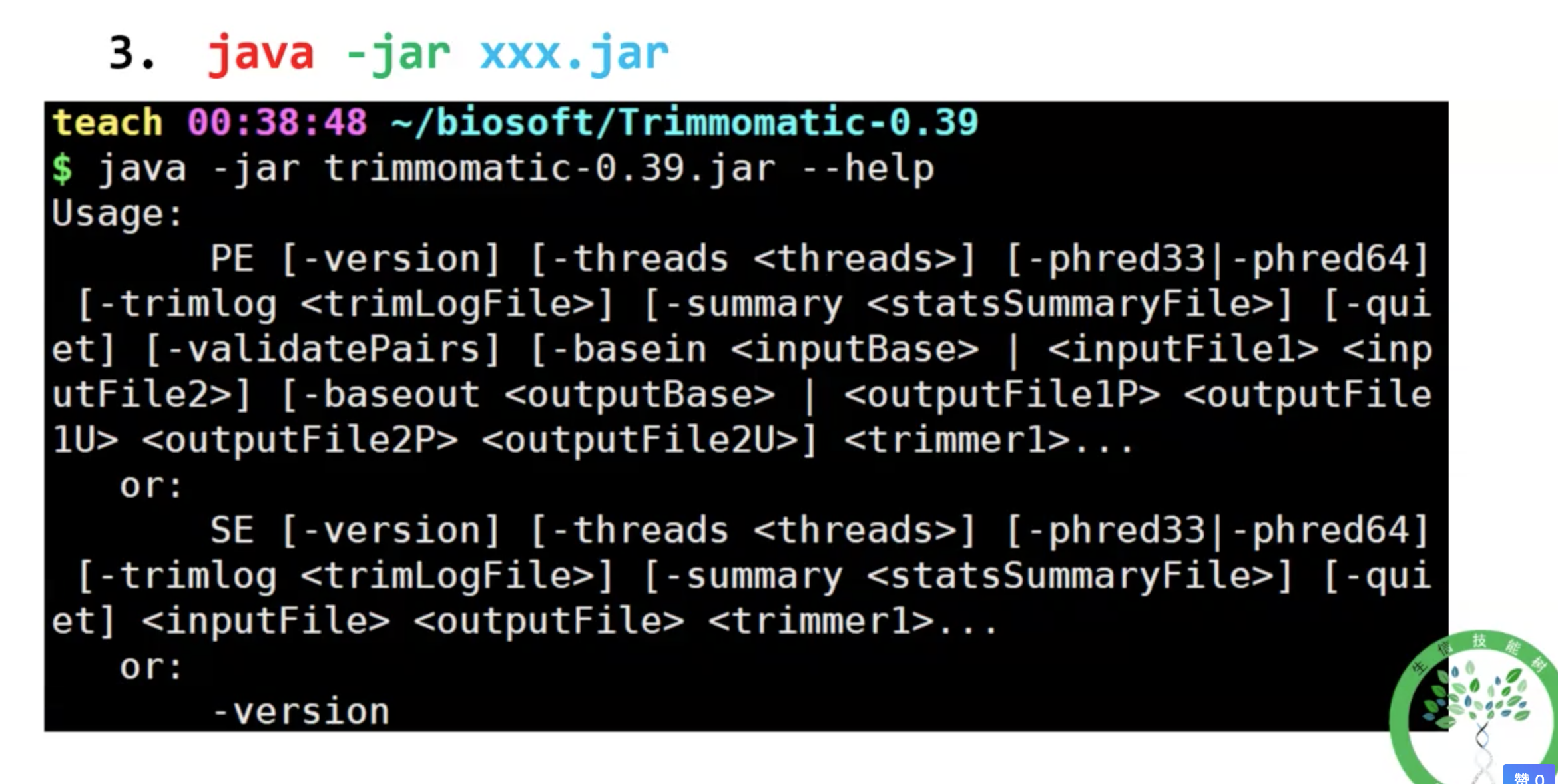
3)java 编译软件

下载并解压
使用
其他安装方法
apt-get
sudo apt-get install w3m
w3m 是一个简易网页浏览器。
w3m https://www.lanqiao.cn/faq
apt 包介绍
APT 是 Advance Packaging Tool(高级包装工具)的缩写,是 Debian 及其派生发行版的软件包管理器,APT 可以自动下载,配置,安装二进制或者源代码格式的软件包,因此简化了 Unix 系统上管理软件的过程。APT 最早被设计成 dpkg 的前端,用来处理 deb 格式的软件包。现在经过 APT-RPM 组织修改,APT 已经可以安装在支持 RPM 的系统管理 RPM 包。这个包管理器包含以 apt- 开头的多个工具,如 apt-get apt-cache apt-cdrom 等,在 Debian 系列的发行版中使用。
常用工具
| 工具 | 说明 |
|---|---|
install |
其后加上软件包名,用于安装一个软件包 |
update |
从软件源镜像服务器上下载/更新用于更新本地软件源的软件包列表 |
upgrade |
升级本地可更新的全部软件包,但存在依赖问题时将不会升级,通常会在更新之前执行一次update |
dist-upgrade |
解决依赖关系并升级(存在一定危险性) |
remove |
移除已安装的软件包,包括与被移除软件包有依赖关系的软件包,但不包含软件包的配置文件 |
autoremove |
移除之前被其他软件包依赖,但现在不再被使用的软件包 |
purge |
与 remove 相同,但会完全移除软件包,包含其配置文件 |
clean |
移除下载到本地的已经安装的软件包,默认保存在/var/cache/apt/archives/ |
autoclean |
移除已安装的软件的旧版本软件包 |
常用参数
| 参数 | 说明 |
|---|---|
-y |
自动回应是否安装软件包的选项,在一些自动化安装脚本中使用这个参数将十分有用 |
-s |
模拟安装 |
-q |
静默安装方式,指定多个q或者-q=#,#表示数字,用于设定静默级别,这在你不想要在安装软件包时屏幕输出过多时很有用 |
-f |
修复损坏的依赖关系 |
-d |
只下载不安装 |
--reinstall |
重新安装已经安装但可能存在问题的软件包 |
--install-suggests |
同时安装 APT 给出的建议安装的软件包 |
安装与升级与卸载
sudo apt-get --reinstall install w3m ,需要使用参数。
# 更新软件源
sudo apt-get update
# 升级没有依赖问题的软件包
sudo apt-get upgrade
# 升级并解决依赖关系
sudo apt-get dist-upgrade
sudo apt-get remove w3m
# 不保留配置文件的移除
$ sudo apt-get purge w3m
# 或者 sudo apt-get --purge remove
# 移除不再需要的被依赖的软件包
$ sudo apt-get autoremove
# 在本地查找是否存在某个软件
sudo apt-cache search softname1 softname2 softname3……
dpkg
dpkg 可以从本地磁盘安装 deb 软件包。
dpkg 本身是一个底层的工具。上层的工具,像是 APT,被用于从远程获取软件包以及处理复杂的软件包关系。”dpkg”是”Debian Package”的简写。
我们经常可以在网络上见到以deb形式打包的软件包,就需要使用dpkg命令来安装。
常用参数
安装相关内容
可以使用-d 选项,使apt 下载但不安装。
sudo apt-get update
sudo apt-get -d install -y emacs
emacs 是非常有名的代码编辑器。
接着便可以通过dpkg 安装deb 包。
sudo dpkg -i emacs24_24.5+1-6ubuntu1.1_amd64.deb
由于dpkg 命令不会帮助我们解决依赖包的问题,所以有时安装会出错,因此需要apt 帮助我们下载相关的依赖包。
sudo apt-get -f install -y
如果想知道下载的软件包安装在了什么位置,可以通过dpkg 查询:
sudo dpkg -L emacs24