0x01 数据归一化
1.1 为什么要数据归一化
在实际应用中,样本的不同特征的单位不同,会在求距离时造成很大的影响。比如:在两个样本中肿瘤大小的分别为1cm和5cm,发现时间分别为100天和200天,那么在求距离时,时间差为100、大小差为4,那么其结果会被时间所主导,因为肿瘤大小的差距太小了。但是如果我们把时间用年做单位,0.27年与0.55年的差距又远小于肿瘤大小的差距,结果又会被大小主导了。
我们发现,在量纲不同的情况下,以上的情况,不能反映样本中每一个特征的重要程度。这就需要数据归一化了。
一般来说,我们的解决方案是:把所有的数据都映射到同一个尺度(量纲)上。
常用的数据归一化有两种:
- 最值归一化(normalization): 把所有数据映射到0-1之间。最值归一化的使用范围是特征的分布具有明显边界的(分数0~100分、灰度0~255),受outlier的影响比较大
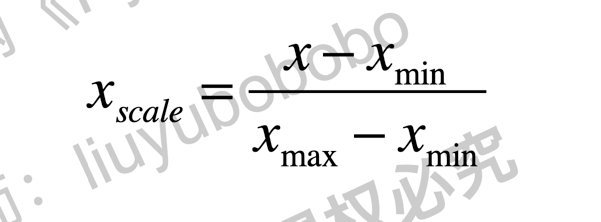
- 均值方差归一化(standardization): 把所有数据归一到均值为0方差为1的分布中。适用于数据中没有明显的边界,有可能存在极端数据值的情况.
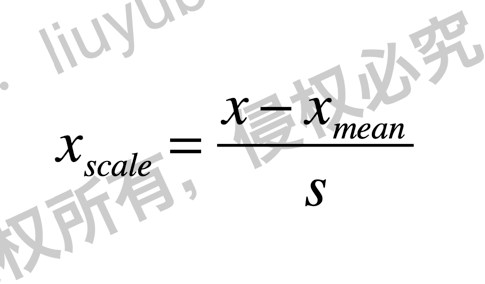
1.2 最值归一化实现
import numpy as npimport matplotlib.pyplot as pltx = np.random.randint(0, 100, 100)# 最值归一化(向量)# 最值归一化公式,映射到0,1之间(x - np.min(x)) / (np.max(x) - np.min(x))# 最值归一化(矩阵)# 0~100范围内的50*2的矩阵X = np.random.randint(0, 100, (50, 2))X = np.array(X, dtype=float) # 将矩阵改为浮点型# 最值归一化公式,对于每一个维度(列方向)进行归一化。# X[:,0]第一列,第一个特征X[:, 0] = (X[:, 0] - np.min(X[:, 0])) / (np.max(X[:, 0]) - np.min(X[:, 0]))# X[:,1]第一列,第二个特征X[:, 1] = (X[:, 1] - np.min(X[:, 1])) / (np.max(X[:, 1]) - np.min(X[:, 1]))”“”# 如果有n个特征,可以写个循环:for i in range(0, n):X[:,i] = (X[:,i]-np.min(X[:,i])) / (np.max(X[:,i] - np.min(X[:,i])))“”“np.mean(X[:, 0]) # 0.4716326530612245np.std(X[:, 0]) # 0.3248636660191091np.mean(X[:, 1]) # 0.4942268041237113np.std(X[:, 1]) # 0.2936853396994547plt.scatter(X[:,0], X[:,1])plt.show()
1.3 均值方差归一化实现
X2 = np.random.randint(0, 100, (50, 2))X2 = np.array(X2, dtype=float)”“”# 方式一: 直接对每一列做均值方差归一化X2[:,0] = (X2[:,0] - np.mean(X2[:,0])) / np.std(X2[:,0])X2[:,1] = (X2[:,1] - np.mean(X2[:,1])) / np.std(X2[:,1])“”“# 方式二: 套用公式,对每一列做均值方差归一化for i in range(0,2):X2[:,i]=(X2[:,i]-np.mean(X2[:,i])) / np.std(X2[:,i])np.mean(X2[:,0]) # -9.769962616701378e-17np.std(X2[:,0]) # 0.9999999999999999np.mean(X2[:,1]) # 7.993605777301127e-17np.std(X2[:,1]) # 1.0plt.scatter(X2[:,0], X2[:,1])plt.show()

1.4 Scikit-learn中的Scaler
首先我们来看一个在实际使用归一化时的一个小陷阱。
我们在建模时要将数据集划分为训练数据集&测试数据集。
训练数据集进行归一化处理,需要计算出训练数据集的均值mean_train和方差std_train。
问题是:我们在对测试数据集进行归一化时,要计算测试数据的均值和方差么?
答案是否定的。在对测试数据集进行归一化时,仍然要使用训练数据集的均值train_mean和方差std_train。这是因为测试数据是模拟的真实环境,真实环境中可能无法得到均值和方差,对数据进行归一化。只能够使用公式(x_test - mean_train) / std_train并且,数据归一化也是算法的一部分,针对后面所有的数据,也应该做同样的处理.
因此我们要保存训练数据集中得到的均值和方差。
在sklearn中专门的用来数据归一化的方法:StandardScaler。
import numpy as npfrom sklearn import datasetsiris = datasets.load_iris()X = iris.datay = iris.targetfrom sklearn.model_selection import train_test_splitX_train, X_test, y_train, y_test = train_test_split(iris.data, iris.target, test_size=0.2, random_state=666)X[:10,:]
| # X[:10,:] array([[5.1, 3.5, 1.4, 0.2], [4.9, 3. , 1.4, 0.2], [4.7, 3.2, 1.3, 0.2], [4.6, 3.1, 1.5, 0.2], [5. , 3.6, 1.4, 0.2], [5.4, 3.9, 1.7, 0.4], [4.6, 3.4, 1.4, 0.3], [5. , 3.4, 1.5, 0.2], [4.4, 2.9, 1.4, 0.2], [4.9, 3.1, 1.5, 0.1]]) |
|---|
# scikit-learn中的StandardScalerstandardScaler = StandardScaler()standardScaler.fit(X_train) # StandardScaler(copy=True, with_mean=True, with_std=True)standardScaler.mean_ # array([5.83416667, 3.08666667, 3.70833333, 1.17 ])standardScaler.scale_ # array([0.81019502, 0.44327067, 1.76401924, 0.75317107])standardScaler.transform(X_train)X_train[:10,:]array([[5.1, 3.5, 1.4, 0.2],[4.9, 3. , 1.4, 0.2],[5.7, 2.8, 4.1, 1.3],[6.2, 3.4, 5.4, 2.3],[5.1, 2.5, 3. , 1.1],[7. , 3.2, 4.7, 1.4],[6.1, 2.6, 5.6, 1.4],[7.6, 3. , 6.6, 2.1],[5.2, 4.1, 1.5, 0.1],[6.2, 2.2, 4.5, 1.5]])X_train = standardScaler.transform(X_train)X_train[:10,:]array([[-0.90616043, 0.93246262, -1.30856471, -1.28788802],[-1.15301457, -0.19551636, -1.30856471, -1.28788802],[-0.16559799, -0.64670795, 0.22203084, 0.17260355],[ 0.45153738, 0.70686683, 0.95898425, 1.50032315],[-0.90616043, -1.32349533, -0.40154513, -0.09294037],[ 1.43895396, 0.25567524, 0.56216318, 0.30537551],[ 0.3281103 , -1.09789954, 1.0723617 , 0.30537551],[ 2.1795164 , -0.19551636, 1.63924894, 1.23477923],[-0.78273335, 2.2860374 , -1.25187599, -1.42065998],[ 0.45153738, -2.00028272, 0.44878573, 0.43814747]])X_test_standard = standardScaler.transform(X_test)
# 使用归一化后的数据进行knn分类
from sklearn.neighbors import KNeighborsClassifier
knn_clf = KNeighborsClassifier(n_neighbors=3)
knn_clf.fit(X_train, y_train)
# KNeighborsClassifier(algorithm='auto', leaf_size=30, metric='minkowski', metric_params=None, n_jobs=None, n_neighbors=3, p=2, weights='uniform')
knn_clf.score(X_test_standard, y_test) # 归一化后的测试数据
# 1.0
knn_clf.score(X_test, y_test) # 注意,此时不能传入没有归一化的数据!
# 0.3333333333333333
1.5 自己实现均值方差归一化
import numpy as np
class StandardScaler:
def __init__(self):
self.mean_ = None
self.scale_ = None
def fit(self, X):
"""根据训练数据集X获得数据的均值和方差"""
assert X.ndim == 2, "The dimension of X must be 2"
self.mean_ = np.array([np.mean(X[:, i]) for i in range(X.shape[1])])
self.scale_ = np.array([np.std(X[:, i]) for i in range(X.shape[1])])
return self
def transform(self, X):
"""将X根据这个StandardScaler进行均值方差归一化处理"""
assert X.ndim == 2, "The dimension of X must be 2"
assert self.mean_ is not None and self.scale_ is not None, "must fit before transform"
assert X.shape[1] == len(self.mean_), "the feature number of X must be equal to mean_ and std_"
resX = np.empty(shape=X.shape, dtype=float)
for col in range(X.shape[1]):
resX[:, col] = (X[:, col] - self.mean_[col]) / self.scale_[col]
return resX
X_train, X_test, y_train, y_test = train_test_split(iris.data, iris.target, test_size=0.2, random_state=666)
from playML.preprocessing import StandardScaler
my_standardScalar = StandardScaler()
my_standardScalar.fit(X_train) # <playML.preprocessing.StandardScaler at 0x1286ae438>
my_standardScalar.mean_ # array([5.83416667, 3.08666667, 3.70833333, 1.17 ])
my_standardScalar.scale_ # array([0.81019502, 0.44327067, 1.76401924, 0.75317107])
X_train = my_standardScalar.transform(X_train)
X_train[:10,:]
array([[-0.90616043, 0.93246262, -1.30856471, -1.28788802],
[-1.15301457, -0.19551636, -1.30856471, -1.28788802],
[-0.16559799, -0.64670795, 0.22203084, 0.17260355],
[ 0.45153738, 0.70686683, 0.95898425, 1.50032315],
[-0.90616043, -1.32349533, -0.40154513, -0.09294037],
[ 1.43895396, 0.25567524, 0.56216318, 0.30537551],
[ 0.3281103 , -1.09789954, 1.0723617 , 0.30537551],
[ 2.1795164 , -0.19551636, 1.63924894, 1.23477923],
[-0.78273335, 2.2860374 , -1.25187599, -1.42065998],
[ 0.45153738, -2.00028272, 0.44878573, 0.43814747]])
X_test_standard = standardScaler.transform(X_test)
X_test_standard[:10,:]
array([[-0.28902506, -0.19551636, 0.44878573, 0.43814747],
[-0.04217092, -0.64670795, 0.78891808, 1.63309511],
[-1.0295875 , -1.77468693, -0.23147896, -0.22571233],
[-0.04217092, -0.87230374, 0.78891808, 0.96923531],
[-1.52329579, 0.03007944, -1.25187599, -1.28788802],
[-0.41245214, -1.32349533, 0.16534211, 0.17260355],
[-0.16559799, -0.64670795, 0.44878573, 0.17260355],
[ 0.82181859, -0.19551636, 0.8456068 , 1.10200727],
[ 0.57496445, -1.77468693, 0.39209701, 0.17260355],
[-0.41245214, -1.09789954, 0.39209701, 0.03983159]])
0x02 KNN的优缺点
KNN的主要优点有:
- 理论成熟,思想简单,既可以用来做分类也可以用来做回归
- 天然解决多分类问题,也可用于回归问题
- 和朴素贝叶斯之类的算法比,对数据没有假设,准确度高,对异常点不敏感
- 由于KNN方法主要靠周围有限的邻近的样本,而不是靠判别类域的方法来确定所属类别的,因此对于类域的交叉或重叠较多的待分样本集来说,KNN方法较其他方法更为适合
KNN的主要缺点有:
- 计算量大,效率低。即使优化算法,效率也不高。
- 高度数据相关,样本不平衡的时候,对稀有类别的预测准确率低
- 相比决策树模型,KNN模型可解释性不强
- 维度灾难:随着维度的增加,“看似相近”的两个点之间的距离越来越大,而knn非常依赖距离
| 维数 | 点到点 | 距离 |
|---|---|---|
| 1维 | 0到1的距离 | 1 |
| 2维 | (0,0)到(1,1)的距离 | 1.414 |
| 3维 | (0,0,0)到(1,1,1)的距离 | 1.73 |
| 64维 | (0,0,…0)到(1,1,…1) | 8 |
| 10000维 | (0,0,…0)到(1,1,…1) | 100 |
大家感觉一万维貌似很多,但实际上就是100*100像素的黑白灰图片。

