获取更多R语言知识,请关注公众号:医学和生信笔记
医学和生信笔记 公众号主要分享:1.医学小知识、肛肠科小知识;2.R语言和Python相关的数据分析、可视化、机器学习等;3.生物信息学学习资料和自己的学习笔记!
介绍
不同于普通的相关性热图,此相关性热图的2个变量是不同的,比如展示10种免疫细胞和20个基因彼此之间的相关性,此时 corrplot 包无法满足要求,需要使用 ggplot2 进行展示。
准备数据
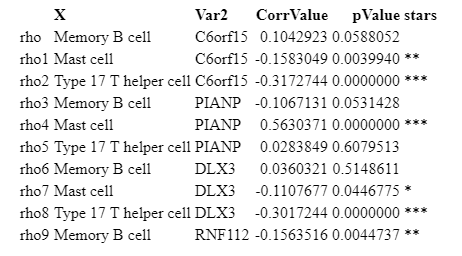
## 首先你需要获得类似于这种结构的数据,一共5列,第一列和第二列是不同的2个变量,第3列是相关系数,第4## 列是相关系数的P值,第5列是根据P值表示的星号(第5列需要自己构造)# load(file = "correlationHeatmap.RData")knitr::kable(longformData[1:10,])
画图
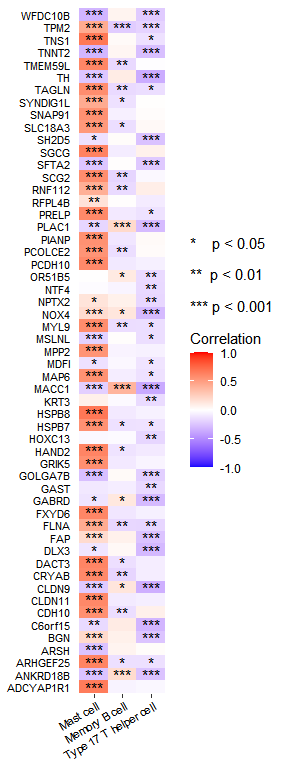
## 使用 geom_tile画图library(ggplot2)ggplot(longformData, aes(X,Var2))+geom_tile(aes(fill=CorrValue))+geom_text(aes(label=stars), color="black", size=4)+scale_fill_gradient2(low='blue', high='red',mid = 'white',limit=c(-1,1),name=paste0("* p < 0.05","\n\n","** p < 0.01","\n\n","*** p < 0.001","\n\n","Correlation"))+labs(x=NULL,y=NULL)+theme(axis.text.x = element_text(size=8,angle = 30,hjust = 1,color = "black"),axis.text.y = element_text(size=8,color = "black"),axis.ticks.y = element_blank(),panel.background=element_blank())
获取更多R语言知识,请关注公众号:医学和生信笔记
医学和生信笔记 公众号主要分享:1.医学小知识、肛肠科小知识;2.R语言和Python相关的数据分析、可视化、机器学习等;3.生物信息学学习资料和自己的学习笔记!