获取更多R语言知识,请关注公众号:医学和生信笔记
医学和生信笔记 公众号主要分享:1.医学小知识、肛肠科小知识;2.R语言和Python相关的数据分析、可视化、机器学习等;3.生物信息学学习资料和自己的学习笔记!
rm(list = ls())library(survival)library(survminer)
使用R包自带数据集lung进行演示
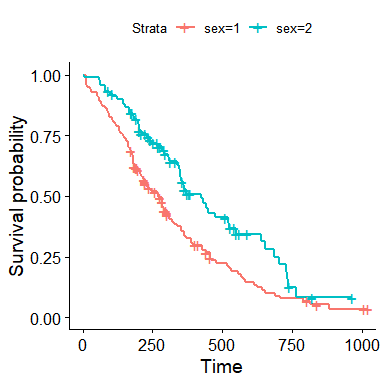
data(lung)## 创建生存对象fit <- survfit(Surv(time, status) ~ sex, data = lung)fit# summary(fit)
ggsurvplot(fit, data = lung)
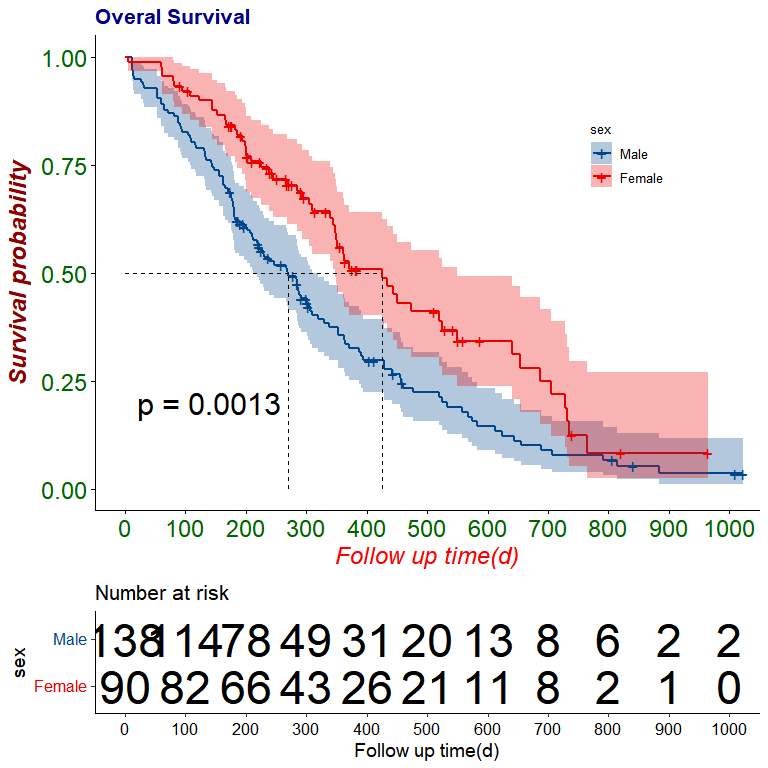
ggsurvplot(fit, # 创建的拟合对象data = lung, # 指定变量数据来源conf.int = TRUE, # 显示置信区间pval = TRUE, # 添加P值pval.size = 8,surv.median.line = "hv", # 添加中位生存时间线risk.table = TRUE, # 添加风险表risk.table.fontsize = 12,xlab = "Follow up time(d)", # 指定x轴标签legend = c(0.8,0.75), # 指定图例位置legend.title = "sex", # 设置图例标题legend.labs = c("Male", "Female"), # 指定图例分组标签break.x.by = 100,palette = "lancet",title = "Overal Survival",font.main = c(16, "bold", "darkblue"),font.x = c(18, "italic", "red"),font.y = c(18, "bold.italic", "darkred"),font.tickslab = c(18, "plain", "darkgreen")) # 设置轴刻度间距
主要参数
ggsurvplot(fit, data = NULL, fun = NULL, color = NULL,palette = NULL, linetype = 1, conf.int = FALSE,pval = FALSE, pval.method = FALSE,test.for.trend = FALSE, surv.median.line = "none",risk.table = FALSE, cumevents = FALSE,cumcensor = FALSE, tables.height = 0.25,group.by = NULL, facet.by = NULL, add.all = FALSE,combine = FALSE, ggtheme = theme_survminer(),tables.theme = ggtheme, ...)
参数解释
fit # 拟合的生存曲线对象data # 用来拟合生存曲线的数据集fun # 常用三个字符参数;”event”绘制累计事件;“cumhaz”绘制累计风险,”pct”绘制生存概率(用百分比表示)color # 设置生存曲线的颜色。如果只有1条曲线,则直接设置color=”blue”;如果有多条曲线,默认color=”strata”,按分组为生存曲线着色;也可以 # 自定义调色板来设置曲线颜色。palette # 调色板,默认”hue”。可选调色板有”grey”,”npg”,”aaas”,”lancet”, “jco”, “ucscgb”,”uchicago”,”simpsons”和”rickandmorty”.linetype = 1 # 设置曲线线型。可以按”strata”设置线型;或按数字向量c(1, 2)或按字符向量c(“solid”, “dashed”)设置conf.int # 逻辑词;默认FASLE;为TRUE则绘制曲线置信区间pval = FALSE # 逻辑词;为TRUE则将统计检验计算的p值添加到图上;为数字,则直接指定P值大小,如pval = 0.03; # 为字符串,则添加字符串到图上,如pval = “p-value: 0.031”pval.method # 逻辑词,是否添加计算p值的统计方法的文本;只有当 pval = TRUE时, 才会在图上添加检验方法文本test.for.trend # 逻辑词,默认为FALSE;为TRUE则返回趋势p值的检验,趋势检验旨在检验生存曲线的有序差异surv.median.line # 在中位生存时间点处绘制水平或垂直线的字符向量;可用值有”none”、”hv”、”h”、”v”;其中v绘制垂直线,h绘制水平线。risk.table = FALSE # 逻辑词,图上是否添加风险表;“absolute” 显示处于风险中的绝对数量;“percentage” 显示处于风险中的百分比数量;# “abs_pct” 显示处于风险中的绝对数量和百分比cumevents # 逻辑词,是否添加累积事件表cumcensor # 逻辑词,是否添加累积删失表tables.height = 0.25 # 生存曲线图下所有生存表的高度,数值0-1之间group.by # 包含分组变量名称的字符向量,向量长度≤2facet.by # 字符向量,指定绘制分面生存曲线的分组变量(应≤2)的名称ggtheme=theme_survminer() # 设置ggplot2主题,如theme_bw()tables.theme # 作用于生存表的ggplot2主题名称,有theme_survminer、theme_cleantable()add.all = FALSE # 逻辑词;是否添加总患者生存曲线到主生存图中
图标题和坐标轴标签
title # 图表标题xlabylab # 分别指x轴和y轴标签
图例标题和位置
legend # 指定图例位置的字符向量:"top"(默认),"bottom","left","right","none"等。legend="none"表示移除图例;# 图例位置也可用数字向量c(x,y)指定,x和y的值应在0到1之间。legend.title # 图例标题,如legend.title = "Sex"。legend.labs # 指定图例标签的字符向量, 替换fit中strata的名称,顺序应与strata一致。# 如 legend.labs = c("Male","Female")
坐标轴范围、刻度间距
break.time.by # 设定坐标轴刻度间距break.x.by # 设定x轴刻度的间距,如break.x.by = 100break.y.by # 设定y轴刻度的间距,如break.y.by = 0.2surv.scale # 生存曲线的比例转换。允许的值为"default"或"percent"。xlimylim # 指定x轴和y轴的范围,如xlim = c(0,30), ylim = c(0,1)axes.offset # 逻辑词,默认为TRUE。为FALSE,则生存曲线图的坐标轴从原点开始。
置信区间
# 以下只有在conf.int = TRUE时才生效conf.int.fill # 设置置信区间填充的颜色conf.int.style # 设置置信区间的类型,有"ribbon"(默认),"step"两种。conf.int.alpha # 数值,指定置信区间填充颜色的透明度;# 数值在0-1之间,0为完全透明,1为不透明。
P值文本大小和位置
# 以下只有在pval = TRUE时才生效pval.size # 指定p值文本大小的数字,默认为 5。pval.coord # 长度为2的数字向量,指定p值位置x、y,如pval.coord=c(x,y)。pval.method.size # 指定检验方法 log.rank 文本的大小pval.method.coord # 指定检验方法 log.rank 文本的坐标log.rank.weights # 计算log-rank检验p值的权重类型的名称。
删失点
censor # 逻辑词,默认为TRUE,在图上绘制删失点。censor.shape # 数值或字符,用于指定删失点的形状;默认为"+"(3), 可选"|"(124)。censor.size # 指定删失点形状的大小,默认为4.5。
生存表
tables.col # 生存曲线图下所有表的颜色,默认为"black";# 按strata显示则tables.col="strata".fontsize # 指定风险表和累积事件表的字体大小。font.family # 指定文字字体的字符向量,如font.family="Courier New".tables.y.text # 逻辑词,默认显示生存表的y轴刻度标签;为FALSE则刻度标签被隐藏tables.y.text.col # 逻辑词,默认FALSE;为TRUE,则表的y刻度标签将按strata着色。tables.height # 指定所有生存表的高度,数值在0-1之间,默认为0.25.
风险表
risk.table.title # 风险表的标题risk.table.pos # 指定风险表位置的字符向量;有两种,"out"在生存图外(默认),"in"在生存图内。risk.table.colrisk.table.fontsizerisk.table.y.textrisk.table.y.text.colrisk.table.height# 这五个和上面生存表的常规参数意义是一样的# 但是只应用在风险表中。
累积事件表
cumevents.title # 累积事件表的标题cumevents.colcumevents.y.textcumevents.y.textcumevents.height# 这四个和上面生存表的常规参数意义是一样的# 但是只应用于累积事件表中。
累积删失表
cumcensor.title # 累积删失表的标题cumcensor.colcumcensor.y.textcumcensor.y.text.colcumcensor.height# 这四个和上面生存表的常规参数意义是一样的# 但是只应用于累积删失表中。
生存图高度
surv.plot.height # 生存图的高度,默认为0.75;# 当risk.table = FALSE时忽略
字体样式
font.title # 标题字体font.subtitle # 副标题字体font.captionfont.xfont.y # x轴、y轴字体font.tickslab # 刻度标签字体font.legend # 图例字体# 用长度为3的向量分别指定大小、类型、颜色。如:# font.main = c(16, "bold", "darkblue")# font.x = c(14, "italic", "red")# font.y = c(14, "bold.italic", "darkred")# font.tickslab = c(12, "plain", "darkgreen")# font.x = 14 则只改变字体的大小# font.x = "bold" 则只改变字体类型
获取更多R语言知识,请关注公众号:医学和生信笔记
医学和生信笔记 公众号主要分享:1.医学小知识、肛肠科小知识;2.R语言和Python相关的数据分析、可视化、机器学习等;3.生物信息学学习资料和自己的学习笔记!