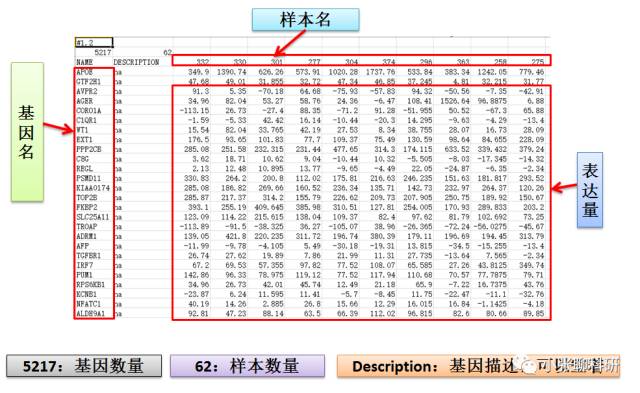
在前面的《GSEA简介》中,简单介绍GSEA,以及GSEA分析调用的后台数据库MsigDB。下面简单介绍GSEA的使用。
第一步、下载GSEA软件:
下载地址:http://software.broadinstitute.org/gsea/downloads.jsp
下载后安装打开(JAVA运行环境大家按照软件提示来安装就可),软件初始界面是: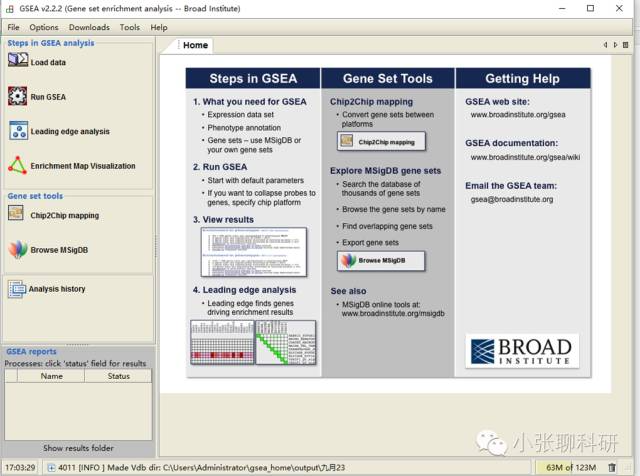 **
**
第二步、准备数据
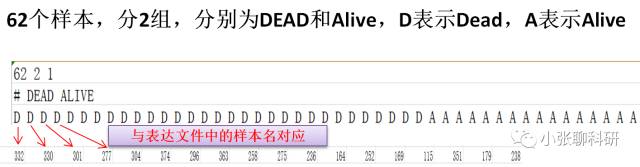
在分析以前我们需要准备两个文件,两个文件可以用Excel打开:
(下载链接: https://pan.baidu.com/s/1pLvZQbx 密码: dfng)
接下来把两个文件导入软件: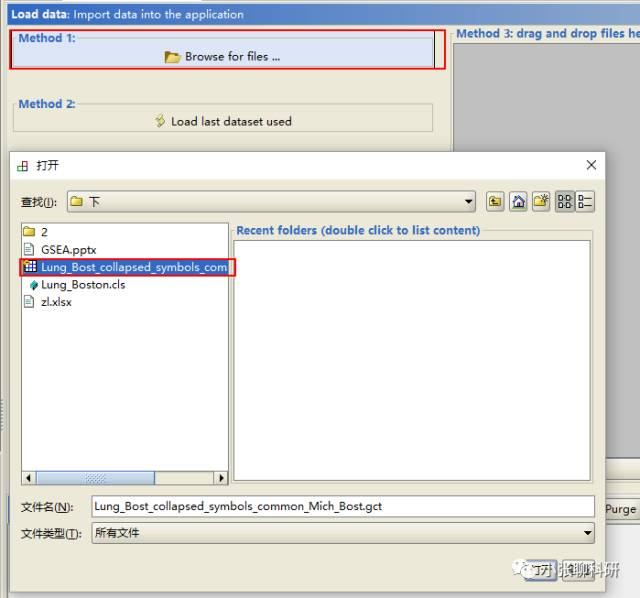
好了以后是这样的: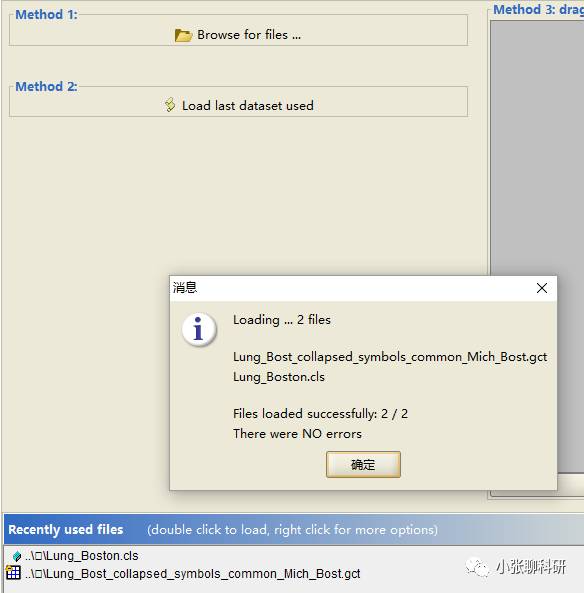
三、数据分析
下面开始设置参数: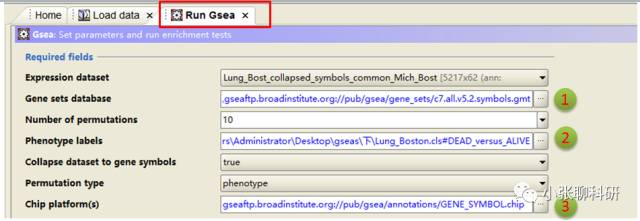
Number of permutations这里最多可以选择1000次(次数越多结果相对越可靠,但是占用CPU越多),permutation type是Geneset,其中1,2和3处的地方设置为: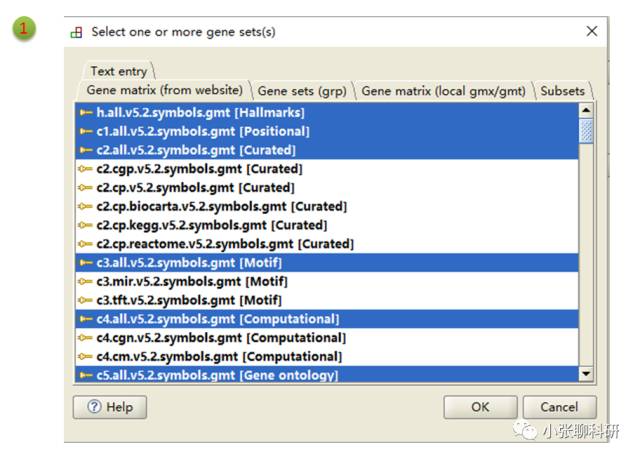
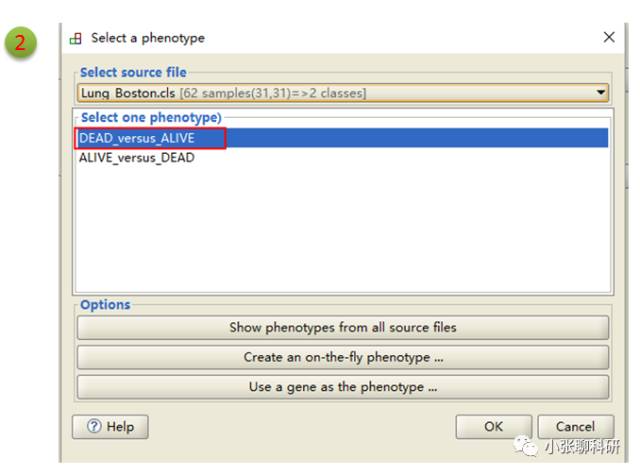
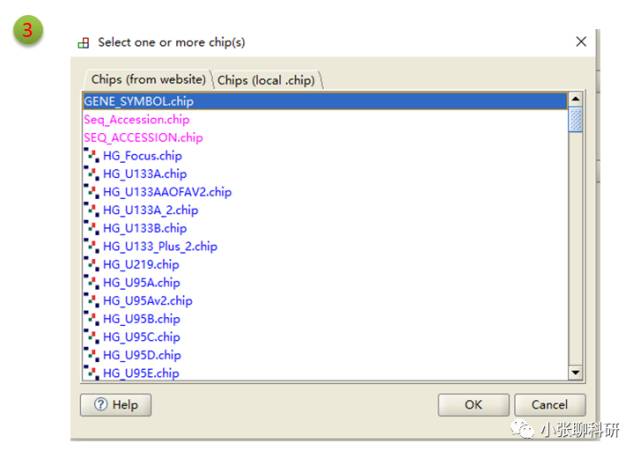
然后运行就好了,运行模式有low和normal两种,是说对CPU的占用率的。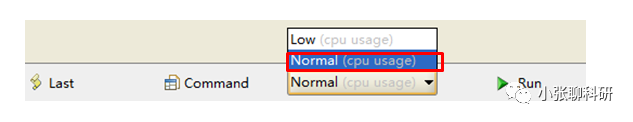
这里我们选择Normal模式,然后单击Run,就可以了:
然后就好了:
这里大家看到,小张在第一次运行的时候也遇到了问题,经过调整第二次才好,同样的道理,大家在用我们推荐的软件或者网站的时候也要多试一下,特别是一些网站,换个浏览器就好了。
四、结果解读
这里如果我们直接单击show results folder,出现这个文件夹:
打开文件夹是这样的:
看的都晕掉了,乱七八糟的什么东东啊!
我们换种方式看:单击success,
会链接到一个网页,这里给出的是所有的结果的说明:
包括在两组样本中上调和下调的基因组合(Geneset)数量,
富集结果的概览和详细信息:
富集结果的概览: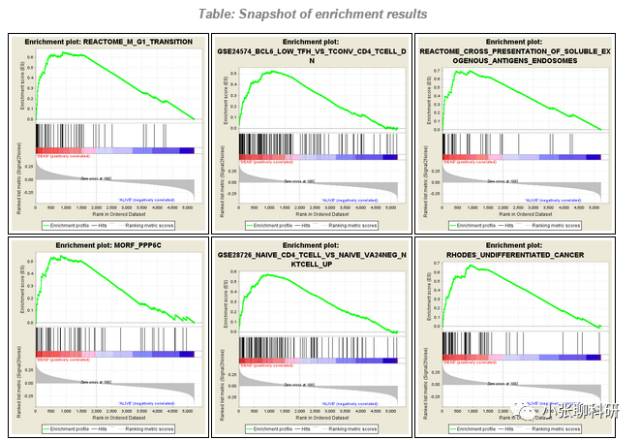
富集结果的详细信息(点看看大图)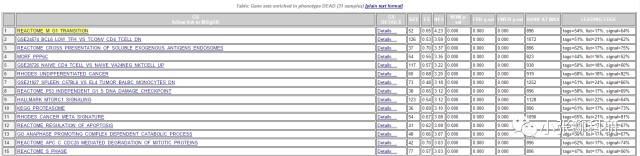
然后每个富集的基因组合都可以点开,看到关于基因的信息。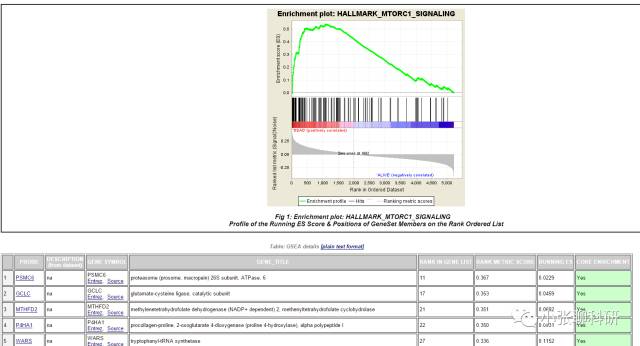
接下来我们看这个Enrichment Plot,在文章里面出现的是最多的,下面的图说的是这个基因组合:
REACTOME_P53_INDEPENDENT_G1_S_DNA_DAMAGE_CHECKPOINT
REACTOME数据库中的(P53非依赖的G1_S期DNA损伤检查点)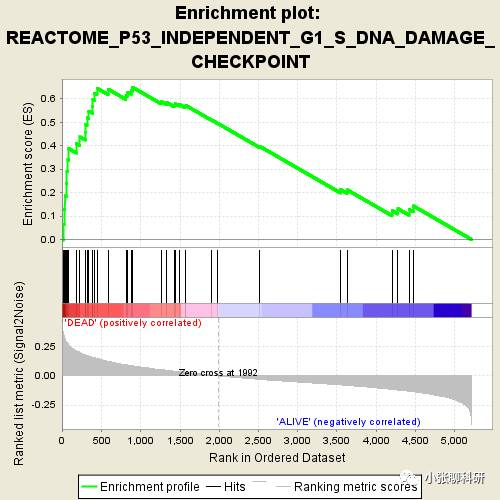
每条黑线代表一个基因,所有的图富集分数(Enrichment Score)一开始都是0。在所有的基因中,如果出现一个基因属于这个组合并且表达量在DEAD组里面表达高于ALIVE组,富集分数就增加,反之就下降。
上面的图,对应到热图里就是下面这个图(单击看大图):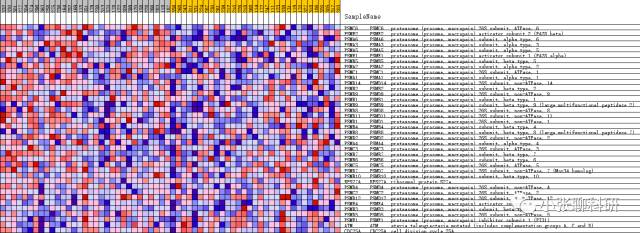
五:补充说明
好了,最后我们说一下这个GSEA分析的一个重要特点:
在我们的输入的文件“基因表达文件”中,给出的不仅是基因名,还有所有基因的表达值。这与我们进行GO/Pathway分析时输入差异表达的基因是不同的,我们在做GO/Pathway富集分析的时候,是首先判断差异表达基因,然后再看差异表达的基因所参与的功能;而GSEA分析则根据一组基因的整体表达趋势来看该组基因是否有差异。
比如:常规的GO/Pathway分析是这样:先从10000个基因中找到差异基因800个(倍数>1.5倍),然后再分析功能;而GSEA则把10000个基因全部放进来,不管差异倍数是1.5还是1.1,统统进行考量和富集。再极端一点,如果某条通路的分子大部分都被上调了,但是倍数只有1.3倍,常规的分析会遗漏该通路,而GSEA分析则能找出来。这一点是GSEA与常规富集分析最大的区别。
- 版权声明 本文源自 小张聊科研, XP 整理
- 转载请务必保留本文链接:https://www.plob.org/article/10815.html