在利用RNA-seq数据比较分析两个样品中同一个基因是否存在差异表达的时候,一般选取两个标准:
i)FoldChange
FoldChange,很容易理解了。就是两样品中同一个基因表达水平的变化倍数。可以用RPKM值来计算,关于RPKM的计算方法,请参考
ii)FDR校正后的p-value,即q-value
FDR值的计算方法如下:
1)对每个基因进行p-value的计算
假设观测到基因A对应的reads数为x,已知在一个大文库中,每个基因的表达量只占所有基因表达量的一小部分,在这种情况下,p(x)的分布服从泊松分布。已知样本一中唯一比对到基因组的总reads数为N1,样本二中唯一比对到基因组的总reads数为N2,样本一中唯一比对到基因A的总reads数为x,样本二中唯一比对到基因A的总reads数为y,则基因A在两样本中表达量相等的概率可由以下公式计算: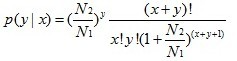
2)用FDR错误控制法对p-value作多重假设检验校正
FDR错误控制法是Benjamini于1995年提出一种方法,通过控制FDR(False Discovery Rate)来决定P值的域值. 假设你挑选了R个差异表达的基因,其中有S个是真正有差异表达的,另外有V个其实是没有差异表达的,是假阳性的。实践中希望错误比例Q=V/R平均而言不能超过某个预先设定的值(比如0.05),在统计学上,这也就等价于控制FDR不能超过5%.
对所有候选基因的p值进行从小到大排序,则若想控制fdr不能超过q,则只需找到最大的正整数i,使得 p(i)<= (iq)/m.然后,挑选对应p(1),p(2),…,p(i)的基因做为差异表达基因,这样就能从统计学上保证fdr不超过q。 因此,FDR的计算公式如下:
q-value(i)=p(i)length(p)/rank(p)
参考文献:
1.Audic, S. and J. M. Claverie (1997). The significance of digital gene expression profiles. Genome Res 7(10): 986-95.
2.Benjamini, Y. and D. Yekutieli (2001). The control of the false discovery rate in multiple testing under dependency. The Annals of Statistics. 29: 1165-1188.
- 本文由 ybzhao
- 转载请务必保留本文链接:https://www.plob.org/article/2361.html

