用基因芯片的手段来探针基因表达量的技术虽然已经在逐步被RNA-seq技术取代,但毕竟经历了十多年的发展了,在GEO或arrayexpress数据库里面存储的全球研究者数据都已经超过了50PB了!实在是很可观,里面还是有非常多等待挖掘的地方!
现在我们要讲的就是基因表达芯片数据的一种分析方式,差异分析,主角就是limma这个package,虽然一直都有各种差异分析统计方法提出来,但是limma包绝对是其中的佼佼者,我们也不多说废话了,直接上教程!
使用这个包需要三个数据:
- 表达矩阵
- 分组矩阵
- 差异比较矩阵
而且总共也只有三个步骤:
- lmFit
- eBayes
- topTable
首先是表达矩阵,这个非常简单了,你可以自己从芯片原始的cel文件开始用affy包读取,然后用rma或者mas5函数做归一化处理,最后得到表达矩阵。也可以直接下载表达矩阵然后用read.table等函数读取到R里面。或者直接下载一个含有表达矩阵的数据对象!
suppressPackageStartupMessages(library(CLL))data(sCLLex)exprSet=exprs(sCLLex) ##sCLLex是依赖于CLL这个package的一个对象samples=sampleNames(sCLLex)pdata=pData(sCLLex)group_list=as.character(pdata[,2])dim(exprSet)## [1] 12625 22exprSet[1:5,1:5]## CLL11.CEL CLL12.CEL CLL13.CEL CLL14.CEL CLL15.CEL## 1000_at 5.743132 6.219412 5.523328 5.340477 5.229904## 1001_at 2.285143 2.291229 2.287986 2.295313 2.662170## 1002_f_at 3.309294 3.318466 3.354423 3.327130 3.365113## 1003_s_at 1.085264 1.117288 1.084010 1.103217 1.074243## 1004_at 7.544884 7.671801 7.474025 7.152482 6.902932group_list ##这个也是存储在sCLLex对象里面的信息,描述了样本的状态,需要根据它来进行样本分类## [1] "progres." "stable" "progres." "progres." "progres." "progres."## [7] "stable" "stable" "progres." "stable" "progres." "stable"## [13] "progres." "stable" "stable" "progres." "progres." "progres."## [19] "progres." "progres." "progres." "stable"
可以看到我们得到了表达矩阵exprSet,它列是各个样本,行是每个探针ID,一个纯粹的表达矩阵,必须是数字型的! 我们可以简单做该表达矩阵做一个QC检测。
par(cex = 0.7)n.sample=ncol(exprSet)if(n.sample>40) par(cex = 0.5)cols <- rainbow(n.sample*1.2)boxplot(exprSet, col = cols,main="expression value",las=2)
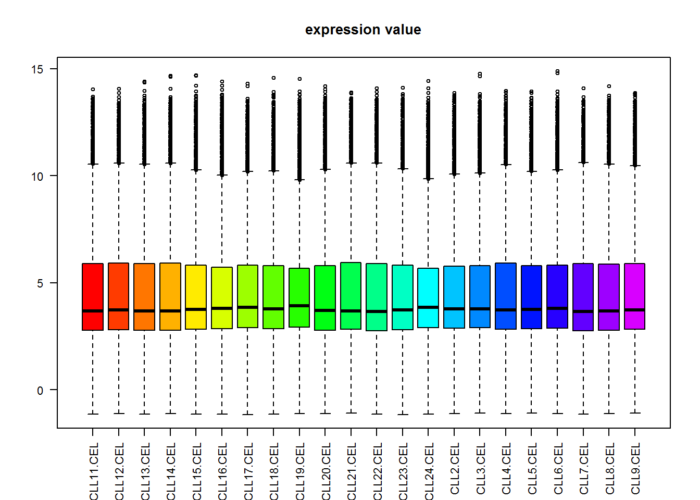
通过这些boxplot可以看到各个芯片直接数据还算整齐,可以进行差异比较
然后我们制作分组矩阵
suppressMessages(library(limma))## Warning: package 'limma' was built under R version 3.1.1design <- model.matrix(~0+factor(group_list))colnames(design)=levels(factor(group_list))rownames(design)=colnames(exprSet)design## progres. stable## CLL11.CEL 1 0## CLL12.CEL 0 1## CLL13.CEL 1 0## CLL14.CEL 1 0## CLL15.CEL 1 0## CLL16.CEL 1 0## CLL17.CEL 0 1## CLL18.CEL 0 1## CLL19.CEL 1 0## CLL20.CEL 0 1## CLL21.CEL 1 0## CLL22.CEL 0 1## CLL23.CEL 1 0## CLL24.CEL 0 1## CLL2.CEL 0 1## CLL3.CEL 1 0## CLL4.CEL 1 0## CLL5.CEL 1 0## CLL6.CEL 1 0## CLL7.CEL 1 0## CLL8.CEL 1 0## CLL9.CEL 0 1## attr(,"assign")## [1] 1 1## attr(,"contrasts")## attr(,"contrasts")$`factor(group_list)`## [1] "contr.treatment"
这个design就是我们制作好的分组矩阵,需要根据我们下载的芯片数据的实验设计方案来,此处例子是CLL疾病的探究,22个样本分成了两组,你们自己的数据只需要按照同样的方法制作即可! 最后一个差异比较矩阵,此处不能省略,即使这里的数据只分成了两组,所以肯定是它们两组直接进行比较啦!如果分成多于两个组,那么就需要制作更复杂的差异比较矩阵。
contrast.matrix<-makeContrasts(paste0(unique(group_list),collapse = "-"),levels = design)contrast.matrix ##这个矩阵声明,我们要把progres.组跟stable进行差异分析比较## Contrasts## Levels progres.-stable## progres. 1## stable -1
那么我们已经制作好了必要的输入数据,下面开始讲如何使用limma这个包来进行差异分析了!
##step1fit <- lmFit(exprSet,design)##step2fit2 <- contrasts.fit(fit, contrast.matrix) ##这一步很重要,大家可以自行看看效果fit2 <- eBayes(fit2) ## default no trend !!!##eBayes() with trend=TRUE##step3tempOutput = topTable(fit2, coef=1, n=Inf)nrDEG = na.omit(tempOutput)#write.csv(nrDEG2,"limma_notrend.results.csv",quote = F)head(nrDEG)## logFC AveExpr t P.Value adj.P.Val B## 39400_at -1.0284628 5.620700 -5.835799 8.340576e-06 0.03344118 3.233915## 36131_at 0.9888221 9.954273 5.771526 9.667514e-06 0.03344118 3.116707## 33791_at 1.8301554 6.950685 5.736161 1.048765e-05 0.03344118 3.051940## 1303_at -1.3835699 4.463438 -5.731733 1.059523e-05 0.03344118 3.043816## 36122_at 0.7801404 7.259612 5.141064 4.205709e-05 0.10619415 1.934581## 36939_at 2.5471980 6.915045 5.038301 5.362353e-05 0.11283285 1.736846
可以看到就三个步骤!最后输出的nrDEG就是我们的差异分析结果! 至于结果该如何解释,大家可以仔细阅读说明书啦!
来自:http://www.bio-info-trainee.com/
- 版权声明 本文源自 生信菜鸟团, ybzhao 整理
- 转载请务必保留本文链接:https://www.plob.org/article/9963.html

