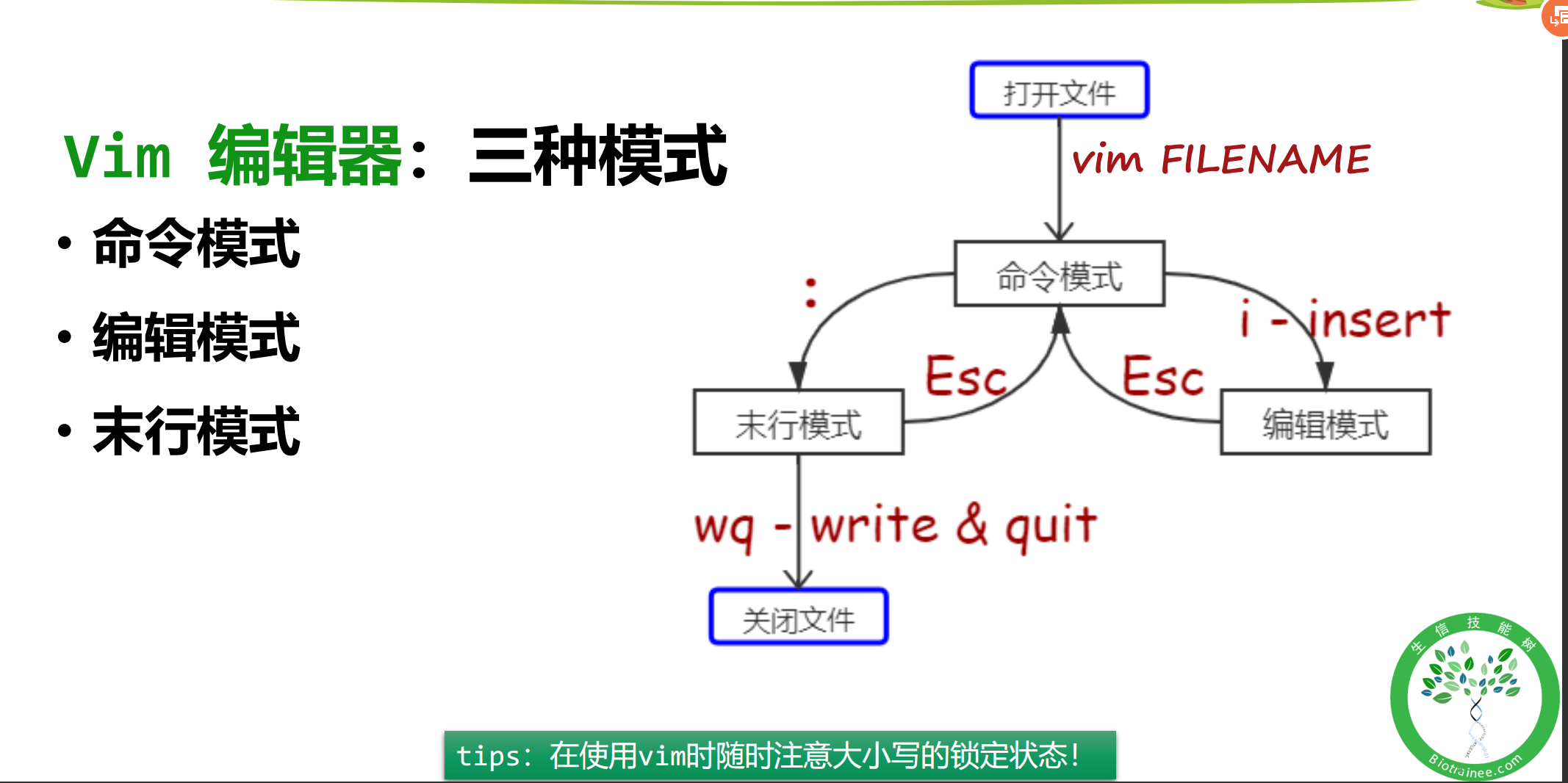
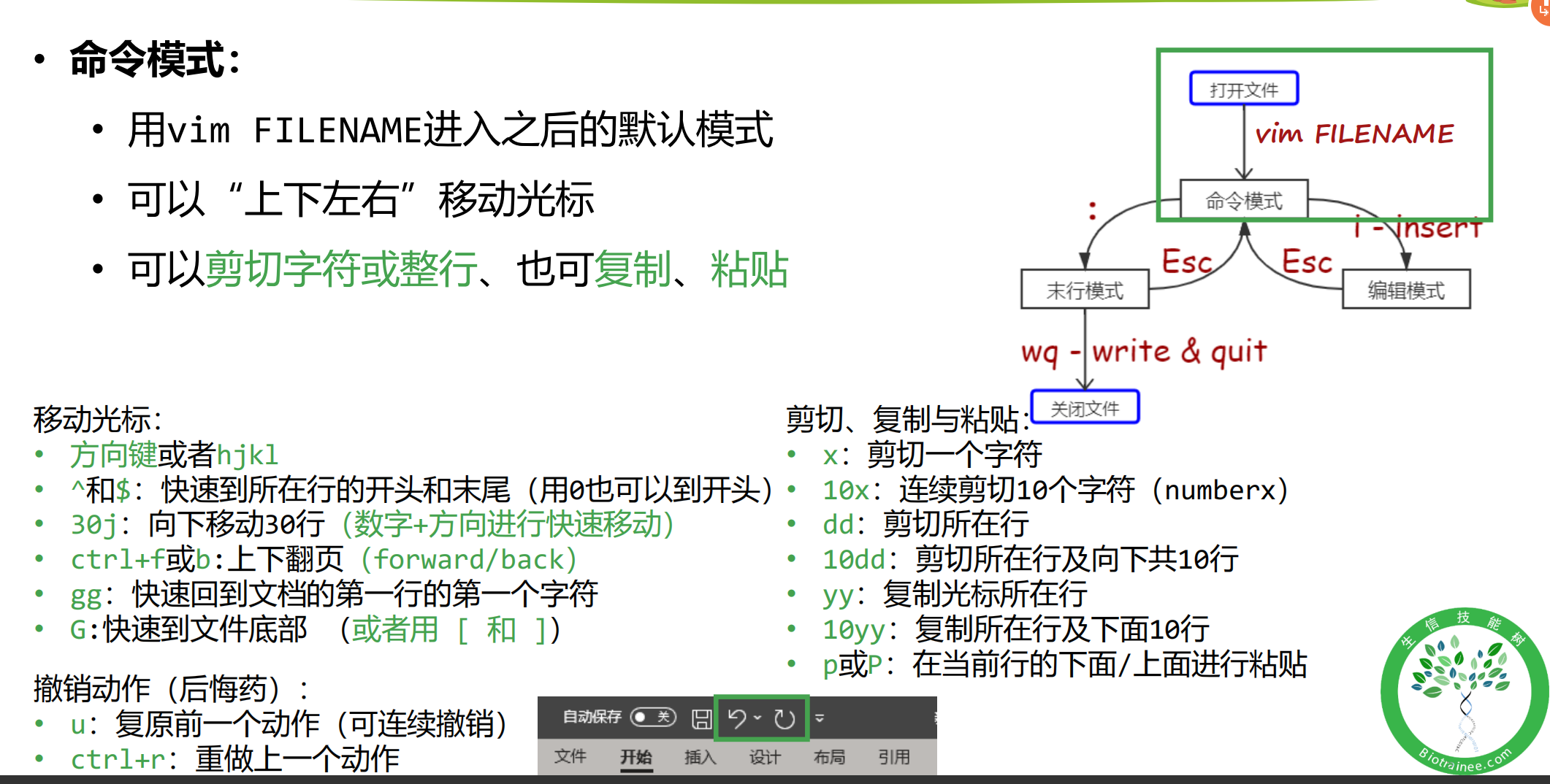
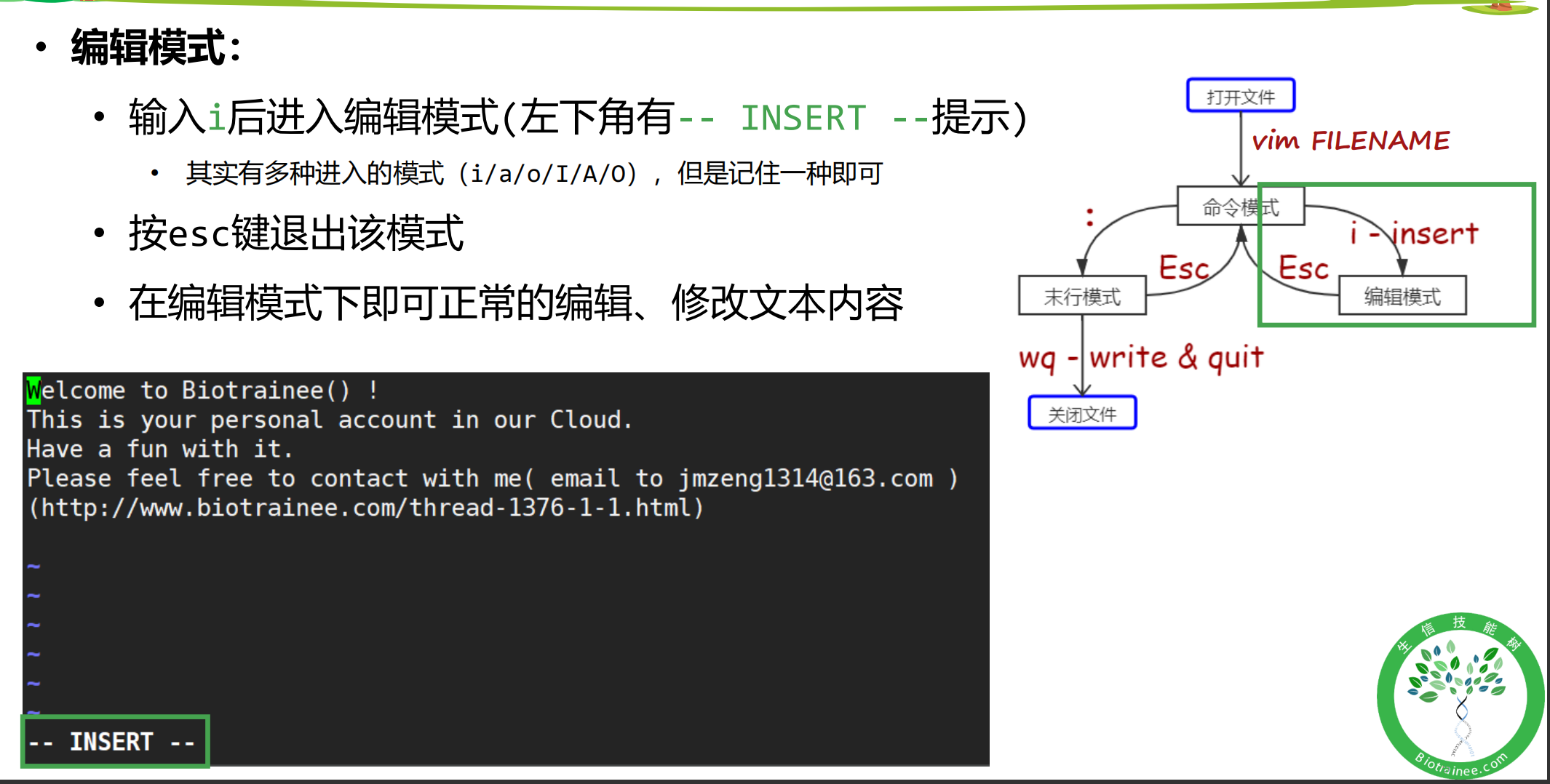
一、Vim编辑器
二、文件处理三架马车
1、grep

grep Biotrainee -r ./less Data/example.gtfless -S Data/example.gtf | grep 'gene'less -S Data/example.gtf | grep -w 'gene'less -S Data/example.gtf | grep -w -c 'gene'less -S Data/example.gtf | grep -v -w 'gene'cat >filegeneUTRstart_codonstop_codon^C(Ctrl C)less -S Data/example.gtf | grep -w -f 'gene' | less -S
补充:正则表达式
##正则表达式是对字符串操作的一种逻辑公式,就是用事先定义好的一些特定字符、
##及这些特定字符的组合,组成一个“规则字符串”,这个“规则字符串”用来表达对字符串的一种过滤逻辑。
^ 行首
$ 行尾
. 换行符之外的任意单个字符
? 匹配之前项0次或者一次
+ 匹配1次或者多次
* 匹配0次或者多次
{n} 匹配n次
{n,} 匹配至少n次
{m,n} 至少m,最多n
[] 匹配任意一个
[^] 排除字符
| 或者
2、sed
##流编辑器,一般用来对文本进行增删改查
##用法:sed [-options] 'script' file(s)
##常见参数:
# -n :禁止显示所有输入内容,只显示经过sed处理的行(常用)
# -e :直接在命令模式上进行 sed 的动作编辑,接要执行的一个或者多个命令
# -f :执行含有 sed 动作的文件
# -r :sed 的动作支持的扩展正则(默认基础正则)
# -i :直接修改读取的文件内容,不输出。
cat readme.txt | sed '1i Welcome to Biotrainee() '
cat readme.txt | sed '1a Welcome to Biotrainee() '
cat readme.txt | sed '1c Welcome to Biotrainee()
cat readme.txt | sed '/^$/d'
cat readme.txt | sed 's/is/IS/g'
cat readme.txt | sed '1 s/ee/EE/g'
cat readme.txt | sed '1~3 s/ee/EE/'
cat readme.txt | sed '/www/ s/ee/EE/'
cat readme.txt | sed 'y/abc/ABC/'
cat readme.txt | sed -n '/ee/p'
cat readme.txt | sed -n 's/ee/EE/p'
3、awk
## 也称gawk,编程语言,可对文本和数据进行处理
## 常见参数:-F,fields,设置字段分隔符;
## -v,var=value 定义awk程序中的一个变量及其默认值
## 用法:awk [options] '{script}' file
## 基础结构: ' {script} '
## 匹配结构: ' /pattern/{script} '
## 扩展结构: 'BEGIN{script} {script} END{script}'
# 基本用法
less -S Data/example.gtf
less -S Data/example.gtf | cut -f 9 | less -S
less -S Data/example.gtf | awk '{print $9}' | less -S
less -S Data/example.gtf | awk '{print $9,$10}' | less -S
less -S Data/example.gtf | awk -F '\t' '{print $9}' | less -S
# 扩展结构
less -S Data/example.gtf | awk '/UTR/{print $0}' | less -S
less -S Data/example.gtf | awk 'BEGIN{print "find UTR feature"} /UTR/{print $0} END{print "end"}'
less -S Data/example.gtf | awk 'BEGIN{FS="\t"} {print $9}' | less -S
less -S Data/example.gtf | awk 'BEGIN{FS="\t"} {print NR,$9}' | less -S
# 条件和循环
less -S Data/example.gtf | awk '{if($3=="gene") {print $0} }' | less -S
less -S Data/example.gtf | awk '{if($3=="gene") {print $1,$2,$3} }' | less -S
less -S Data/example.gtf | awk '{for(i=1;i<4;i++){print $i} }' | less -S
less -S Data/example.gtf | awk '{for(i=1;i<4;i++){print $i} }' | paste - - - | less -S
# 运算和字符处理
less -S Data/example.gtf | awk '/exon/{print $5-$4}' | less -S
less -S Data/example.gtf | awk '{print $5/$4}' | less -S
less -S Data/example.gtf | awk '{print int($5/$4) }' | less -S
less -S Data/example.gtf | awk '{print int($5/$4 + 0.5) }' | less -S
less -S Data/example.gtf | awk 'BEGIN{FS="\t";OFS="\t"} {gsub("gene","Gene",$3);print $0}' | less -S
三、生物信息学常见的数据格式
1、fasta
2、fastq
3、gff
4、gtf
四、软件安装
1.创建小环境,在小环境中安装软件
2.conda
3.mamba
4.理解路径的重要性
五、Linux系统环境
文件系统结构
/ 虚拟目录的根目录。通常不会在这里存储文件
/bin 二进制目录,存放许多用户级的GNU工具
/boot 启动目录,存放启动文件
/dev 设备目录,Linux在这里创建设备节点
/etc 系统配置文件目录
/home 主目录,Linux在这里创建用户目录
/lib 库目录,存放系统和应用程序的库文件
/media 媒体目录,可移动媒体设备的常用挂载点
/root root用户的主目录
/sbin 系统二进制目录,存放许多GNU管理员级工具
/run 运行目录,存放系统运作时的运行时数据
/tmp 临时目录,可以在该目录中创建和删除临时工作文件
/usr 用户二进制目录,大量用户级的GNU工具和数据文件都存储在这里
查看系统资源
查看CPU信息:lscpu
查看内存信息:free -h
查看硬盘信息:df -h
查看文件大小:du -h -d 1
查看文件大小:du -h -d 1
查看系统进程:top 或者 ps -ef 或者 jobs
1、变量
环境变量、状态变量、位置参数变量、自定义变量,调用变量时,要在变量前面加一个 $符号
环境变量:用于存储有关shell会话和工作环境的系统变量
状态变量:用于记录命令的运行结果
位置参数变量:用于用于向命令或程序脚本中传递信息
自定义变量:由用户自行定义的变量,可用于用户编写的脚本,多个命令间的值传递等
2、结构化语句
条件语句 if
#一个` if ` 就要对应一个 ` fi ` 。有三种结构:
# 1
if [ condition ]
then
commands
fi
# 2
if [ condition ]
then
commands
else
commands
fi
# 3
if [ condition ]
then
commands
else
if [ condition ]
then
commands
fi
fi
# (else if 可以缩写为 elif )
if [ condition ]
then
commands
elif [ condition ]
then
commands
fi
fi
循环语句
#for循环
for i in 1 2 3 4 5
do
echo ${i} "Welcome to Biotrainee() !"
done
for i in {1..10}
do
touch file${i}
done
list="CDS exon gene start_codon stop_codon transcript UTR"
for i in ${list}
do
echo "This feature is ${i}"
done
#while 循环
ls file* | while read id;
do
mv ${id} ${id}.txt ;
done
ls file* > config
cat config | while read id
do
mv ${id} ${id%.txt}
done
shell脚本
#test1.sh
$ vim test.sh
#!/bin/bash
echo "Welcome to Biotrainee() !"
$ bash test.sh
# 1标准输出 和 2标准误输出
$ bash test.sh 1>test.log 2>&1
# 可执行权限
$ ls -lh test.sh
$ chmod 764 test.sh
$ ls -lh test.sh
# 路径调用可执行文件
./test.sh
#test2.sh
$ cat test2.sh
#!/bin/bash
cat $1
$ bash test2.sh readme.txt
#test3.sh
$ cat test3.sh
#!/bin/bash
echo "Start"
sleep 100s
echo "End"
$ bash test3.sh
$ nohup bash test3.sh &
$ nohup bash test3.sh 1>test3.log 2>&1 &
$ top
$ ps -ef | grep test3
代码及图片均来自于生信技能树卖萌哥