Phylogenetic Analysis by Maximum Likelihood (PAML)
将最大似然法用于系统发育分析
PAML 是一个用最大似然法来对 DNA 和蛋白质序列进行系统发育分析的软件包,它可以用来估计参数和检验假设,以研究进化过程,目前是由杨子恒团队维护的。
杨子恒,1964 年出生于中国甘肃省,生物学家,英国皇家学会院士 ,英国伦敦大学学院RA Fisher统计遗传学讲座教授,中国科学院动物研究所计算与进化生物学中心主任。
PAML 软件包目前包含如下程序:baseml,basemlg,codeml,evolver,pamp,yn00,mcmctree,和chi2,最主要的是 baseml 和 codeml。
baseml是计算核酸的,codeml 是由两个旧程序合并构成,分别是codonml(对蛋白编码DNA序列执行密码子替代模型)和aaml(用来执行氨基酸序列模型)。
举个例子说明:利用codeml计算dN/dS
蛋白编码基因(protein coding sequence)的自然选择压力水平可以通过dN/dS(ω)值的大小来衡量,其中,dS代表同义替换率(synonymous rate),dN代表非同义替换率(non-synonymous rate)。 在没有受到选择压力时,同义替换率和非同义替换率相等,此时dN/dS = 1; 当受到负选择或净化选择压力时,自然选择会阻止氨基酸发生改变,同义替换率会大于非同义替换率,即dN/dS < 1; 当受到正选择压力时,氨基酸的置换率会受自然选择的青睐,即蛋白功能可能会发生适应性改变,此时dN/dS > 1。
以前学习过密码子表,好几个密码子会对应同一个氨基酸,他们之中的某一个字母的替换就是同义替,比如说TTT和TTC都编码苯丙氨酸,那么由T突变到C就是统一替换;比如说,TTT编码苯丙氨酸,CTT编码亮氨酸,那么由T突变到C就是非同义替换。
通过codeml内的最大似然估计法的模型的估计出祖先的序列,然后与现有的提供的个体的序列进行比较,得到一个 ω 值,这个 ω 值对应了dN/dS ,所以,这个软件的本质是跟统计相关的,在一定的概率下去估计。
如何做?
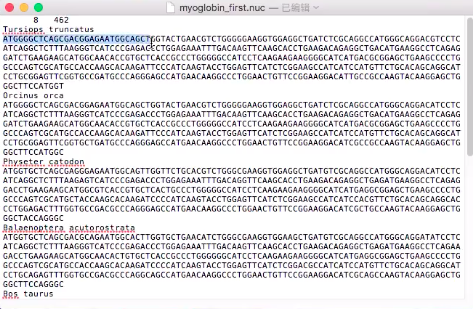
需要准备个体的序列文件,比如说8个物种,在一个文件里面
8 462Hsa_Human AAGGTCTTTGAAAGGTGTGAGTTGGCCAGAACT...Hla_gibbon AAGGTCTTTGAAAGGTGTGAGTTGGCCAGAACT...Cgu/Can_colobus AAGATCTTTGAAAGGTGTGAGTTGGCCAGAACT...Pne_langur AAGATCTTTGAAAGGTGTGAGTTGGCCAGAACT...Mmu_rhesus AAGATCTTTGAAAGGTGTGAGTTGGCCAGAACT...Ssc_squirrelM AAGGTCTTCGAAAGGTGTGAGTTGGCCAGAACT...Cja_marmoset AAGGTCTTTGAAAGGTGTGAGTTGGCCAGAACT...
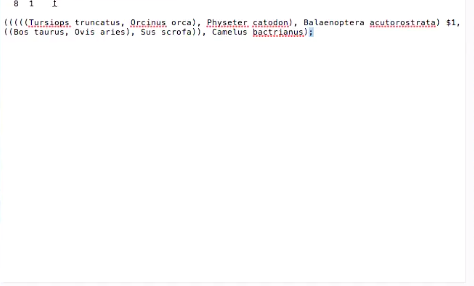
- 树文件,就是经常看见的像树状结构一样的
- Control File
这个文件里面是你要执行的参数设置,
输入输出参数:输入序列路径;输出序列路径等等
数据使用说明参数:
seqtype,设置输入的多序列比对数据的类型:1,密码子数据;2,氨基酸数据;3,输入数据虽然为密码子序列,但先转换为氨基酸序列后再进行分析。
解读结果
观看的教学视频是根据 likelihood ratio 计算的,计算这个的可信度。
参考链接:
Phylogenetic Analysis by Maximum Likelihood (PAML)
PAML中文手册
系统发育分析软件PAML的安装和简介
PAML选择压力分析
【PAML教程】利用codeml计算dN/dS

