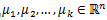- 实验目的
- 实验原理
- 1、 随机选取k个聚类质心点(cluster centroids)为
- 2、 重复下面过程直到收敛 {
- 实验环境
- 实验内容
- 实验步骤
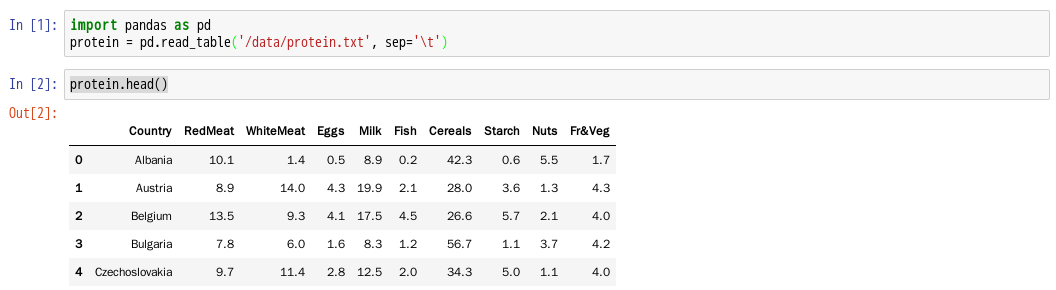
- 4.使用pandas的read_table方法读取protein.txt文件,以\t分隔并传入protein
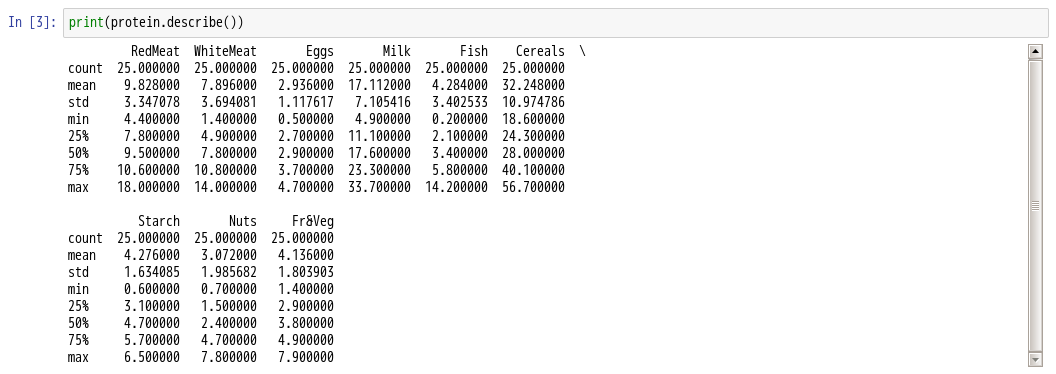
- 5.查看protein的描述性统计
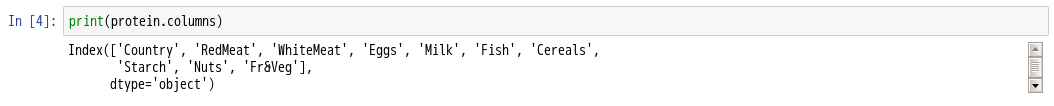
- 6.查看protein的列名
- 7.用.shape方法可以读取矩阵的形状
- 8.导入sklearn模块中的preprocessing函数
- 9.导入sklearn模块中的KMeans方法
- 10.导入Matplotlib模块
- 11.使用KMeans算法生成实例myKmeans
- 12.利用.fit()方法对sprotein_scaled进行模型拟合
- 13.打印输出myKmeans模型
- 14.使用.predict方法,用训练好的模型进行预测
- 15.编写print_kmcluster函数并输出结果
实验目的
实验原理
K-means算法是将样本聚类成k个簇(cluster),具体算法描述如下:
1、 随机选取k个聚类质心点(cluster centroids)为
2、 重复下面过程直到收敛 {
对于每一个样例i,计算其应该属于的类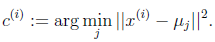
对于每一个类j,重新计算该类的质心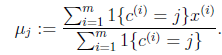
}
K是我们事先给定的聚类数,c(i)代表样例i与k个类中距离最近的那个类,c(i)的值是1到k中的一个。质心uj代表我们对属于同一个类的样本中心点的猜测,拿星团模型来解释就是要将所有的星星聚成k个星团,首先随机选取k个宇宙中的点(或者k个星星)作为k个星团的质心,然后第一步对于每一个星星计算其到k个质心中每一个的距离,然后选取距离最近的那个星团作为c(i),这样经过第一步每一个星星都有了所属的星团;第二步对于每一个星团,重新计算它的质心uj(对里面所有的星星坐标求平均)。重复迭代第一步和第二步直到质心不变或者变化很小。求点群中心的算法:
一般来说,求点群中心点的算法你可以使用各个点的X/Y坐标的平均值。不过,我这里想告诉大家另三个求中心点的的公式:
1)Minkowski Distance公式——λ可以随意取值,可以是负数,也可以是正数,或是无穷大。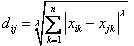
2)Euclidean Distance公式——也就是第一个公式λ=2的情况 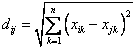
3)CityBlock Distance公式——也就是第一个公式λ=1的情况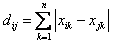
实验环境
- Linux Ubuntu 16.04
- Python 3.6
- Anaconda 4
- IPython Notebook
实验内容
学习KMeans算法,了解模型创建、使用模型及模型评价等操作实验步骤
1.打开终端模拟器,切换到/data目录下,使用wget命令下载实验数据- cd /data
- wget http://192.168.1.100:60000/allfiles/sklearn/protein.txt
2.开启jupyter notebook
- jupyter notebook —ip=’127.0.0.1’
4.使用pandas的read_table方法读取protein.txt文件,以\t分隔并传入protein
5.查看protein的描述性统计
6.查看protein的列名
7.用.shape方法可以读取矩阵的形状
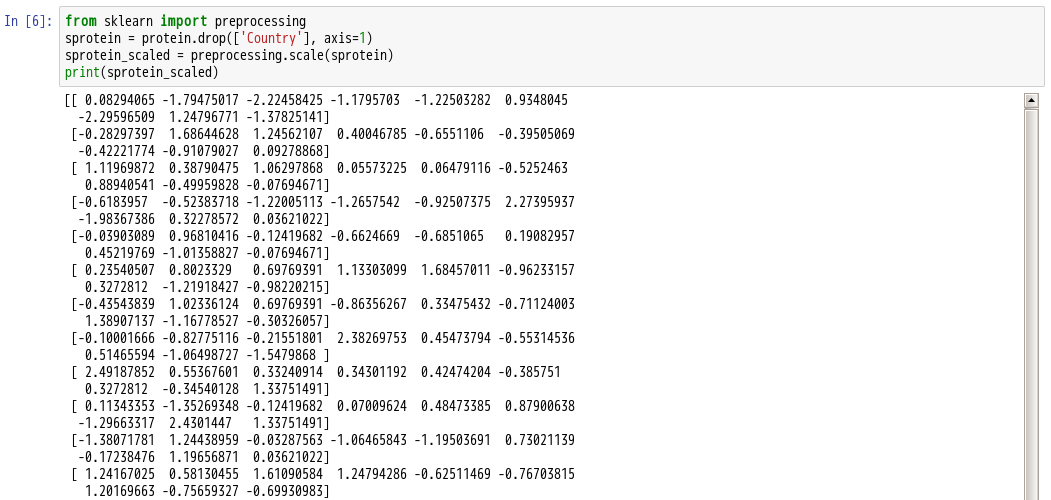
8.导入sklearn模块中的preprocessing函数
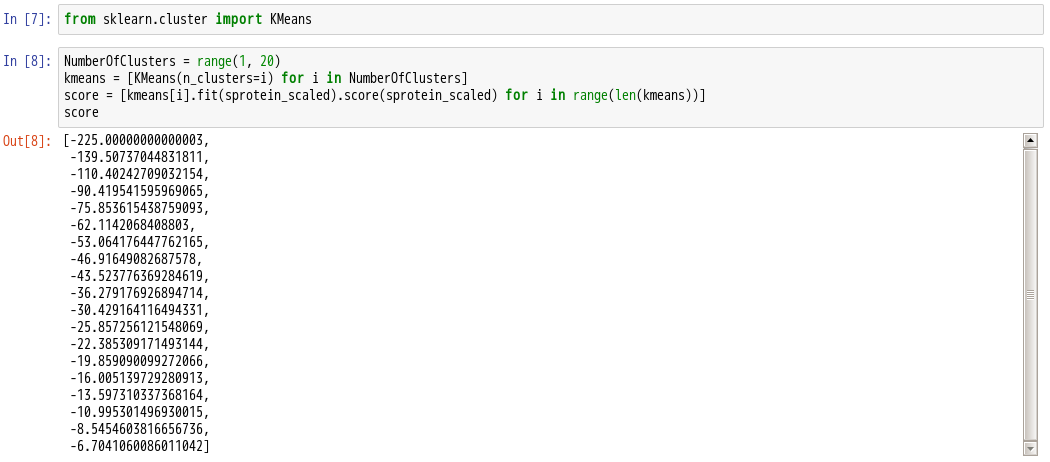
9.导入sklearn模块中的KMeans方法
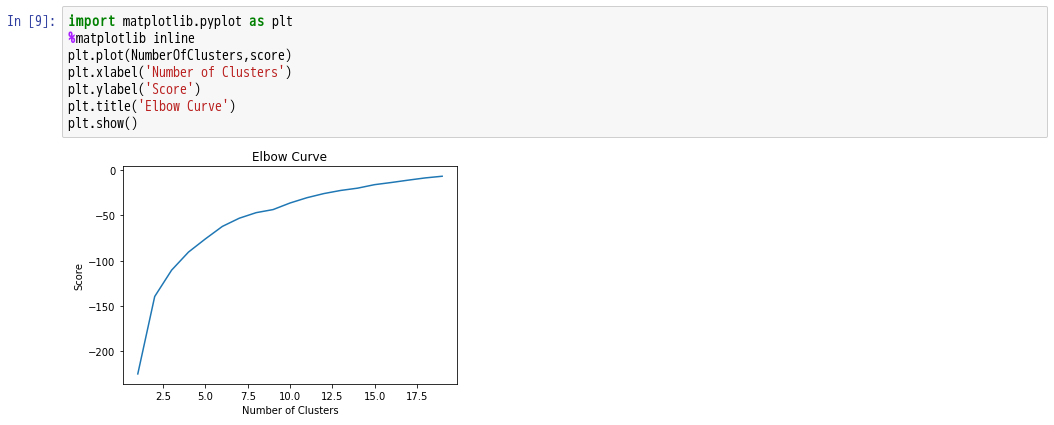
10.导入Matplotlib模块
11.使用KMeans算法生成实例myKmeans
myKmeans = KMeans(algorithm=”auto”,n_clusters=5,n_init=10,max_iter=200)
参数解释:
- algorithm:有“auto”, “full” or “elkan”三种选择,默认的”auto”则会根据数据值是否是稀疏的,来决定如何选择”full”和“elkan”,一般数据是稠密的,那么就是 “elkan”,否则就是”full”
- n_clusters=5:即k值,一般需要多试一些值以获得较好的聚类效果
- n_init:用不同的初始化质心运行算法的次数
- max_iter: 最大的迭代次数
12.利用.fit()方法对sprotein_scaled进行模型拟合
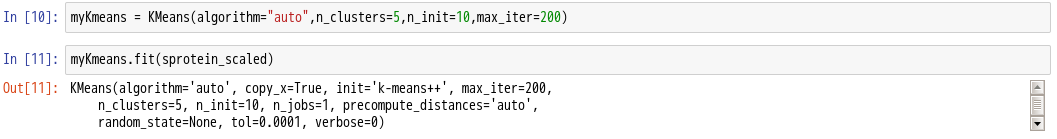
查看模型13.打印输出myKmeans模型

预测模型14.使用.predict方法,用训练好的模型进行预测

结果输出15.编写print_kmcluster函数并输出结果
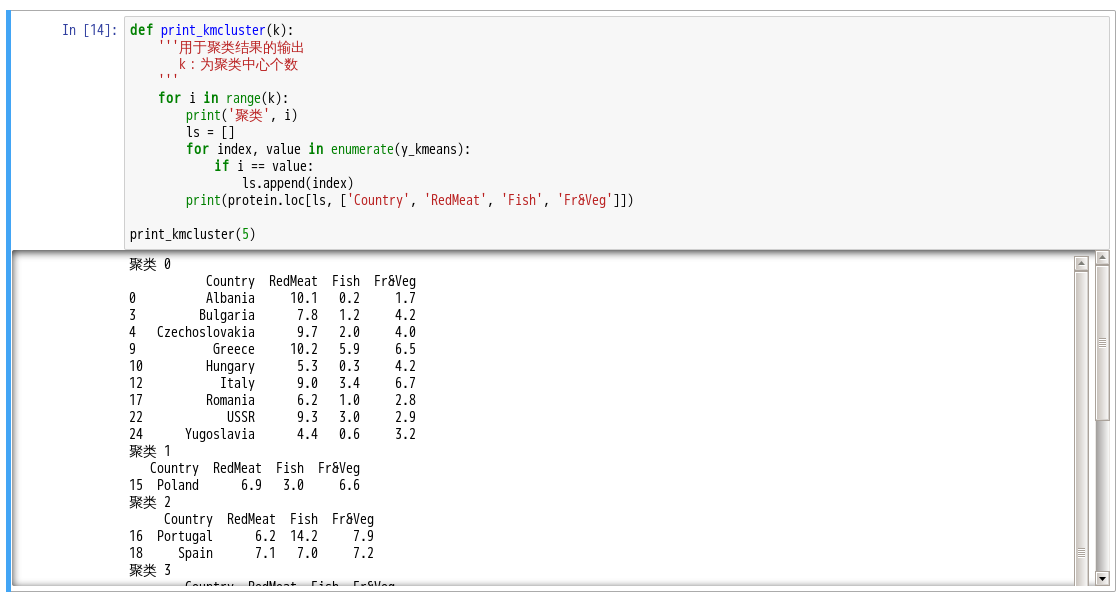
至此,实验结束!