自定义GSEA
https://zhuanlan.zhihu.com/p/663943812
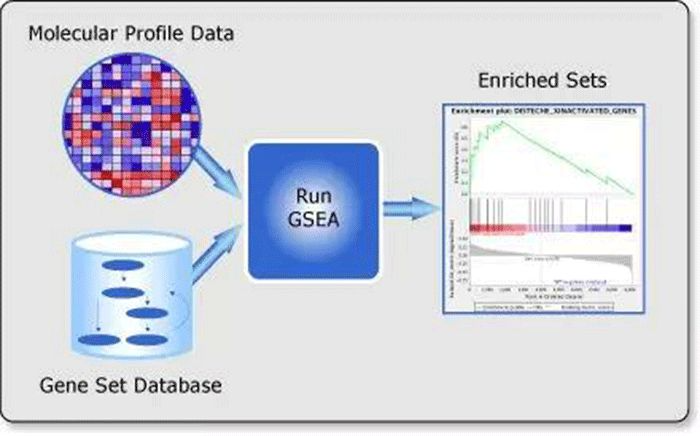
GSEA案例解析
介绍GSEA分析之前,我们先看一篇Cell文章(https://sci-hub.tw/10.1016/j.cell.2016.11.033)的一个插图 (SCI-HUB客户端(文献神器V4.0)——下载文献如此简单)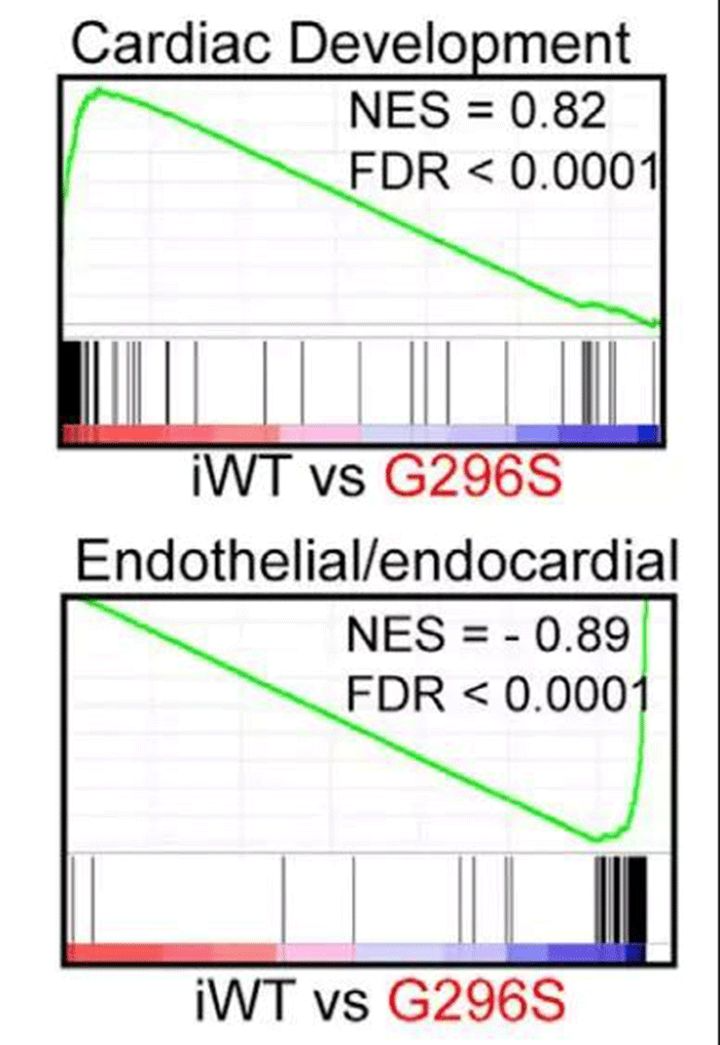
而对于参与内皮或内膜发育的基因集,在iwt组中表达更低,在G296S组中表达更高。
作者根据这个图和其它证据推测iwt组的心脏发育更加完善,而G296S组更倾向于心脏内皮或内膜的发育,即GATA基因的这种突变可能导致心脏内皮或内膜的过度发育而导致心脏相关疾病的产生。
那么GSEA分析是什么?
参考GSEA官网主页的描述:Gene Set Enrichment Analysis (GSEA) is a computational method that determines whether an a priori defined set of genes shows statistically significant, concordant differences between two biological states (e.g. phenotypes). 在上述Cell文章中,作者更加关心参与心脏发育的基因集 (即a priori defined set of genes)与两个状态(突变体和野生型,状态的度量方式是基因表达)的关系,因此利用GSEA对其进行分析后发现,参与心脏发育 (收缩力、钙调控和新陈代谢)的基因集的表达模式更接近于iwt组的表型,而不是G296S组; 而参与心脏内皮或内膜发育的这些基因的表达模式更接近于G296S组的表型而不是iwt组的表型。这就是GSEA分析所适用的主要场景之一。它能帮助生物学家在两种不同的生物学状态 (biological states)中,判断某一组有特定意义的基因集合的表达模式更接近于其中哪一种。因此GSEA是一种非常常见且实用的分析方法,可以将数个基因组成的基因集与整个转录组、修饰组等做出简单而清晰的关联分析。
除了对特定gene set的分析,反过来GSEA也可以用于发现两组样本从表达或其它度量水平分别与哪些特定生物学意义的基因集有显著关联,或者发现哪些基因集的表达模式或其他模式更接近于表型A、哪些更接近于表型B。这些特定的基因集合可以从GO、KEGG、Reactome、hallmark或MSigDB等基因集中获取,其中MSigDB数据库整合了上述所有基因集。研究者也可自定义gene set (即新发现的基因集或其它感兴趣的基因的集合)。 GSEA分析似乎与GO分析类似但又有所不同。GO分析更加依赖差异基因,实则是对一部分基因的分析 (忽略差异不显著的基因),而GSEA是从全体基因的表达矩阵中找出具有协同差异 (concordant differences)的基因集,故能兼顾差异较小的基因。因此二者的应用场景略有区别。另外GO富集是定性的分析,GSEA考虑到了表达或其它度量水平的值的影响。另外,对于时间序列数据或样品有定量属性时,GSEA的优势会更明显,不需要每个分组分别进行富集,直接对整体进行处理。可以类比于之前的WGCNA分析。 ### GSEA定义 Gene Set Enrichment Analysis (基因集富集分析)用来评估一个预先定义的基因集的基因在与表型相关度排序的基因表中的分布趋势,从而判断其对表型的贡献。其输入数据包含两部分,一是已知功能的基因集 (可以是GO注释、MsigDB的注释或其它符合格式的基因集定义),一是表达矩阵 (也可以是排序好的列表),软件会对基因根据其与表型的关联度(可以理解为表达值的变化)从大到小排序,然后判断基因集内每条注释下的基因是否富集于表型相关度排序后基因表的上部或下部,从而判断此基因集内基因的协同变化对表型变化的影响。 (The gene sets are defined based on prior biological knowledge, e.g., published information about biochemical pathways or coexpression in previous experiments. The goal of GSEA is to determine whether members of a gene set S tend to occur toward the top (or bottom) of the list L, in which case the gene set is correlated with the phenotypic class distinction.) 这与之前讲述的GO富集分析不同。GO富集分析是先筛选差异基因,再判断差异基因在哪些注释的通路存在富集;这涉及到阈值的设定,存在一定主观性并且只能用于表达变化较大的基因,即我们定义的显著差异基因。而GSEA则不局限于差异基因,从基因集的富集角度出发,理论上更容易囊括细微但协调性的变化对生物通路的影响,尤其是差异倍数不太大的基因集。GSEA原理
给定一个排序的基因表L和一个预先定义的基因集S (比如编码某个代谢通路的产物的基因, 基因组上物理位置相近的基因,或同一GO注释下的基因),GSEA的目的是判断S里面的成员s在L里面是随机分布还是主要聚集在L的顶部或底部。这些基因排序的依据是其在不同表型状态下的表达差异,若研究的基因集S的成员显著聚集在L的顶部或底部,则说明此基因集成员对表型的差异有贡献,也是我们关注的基因集。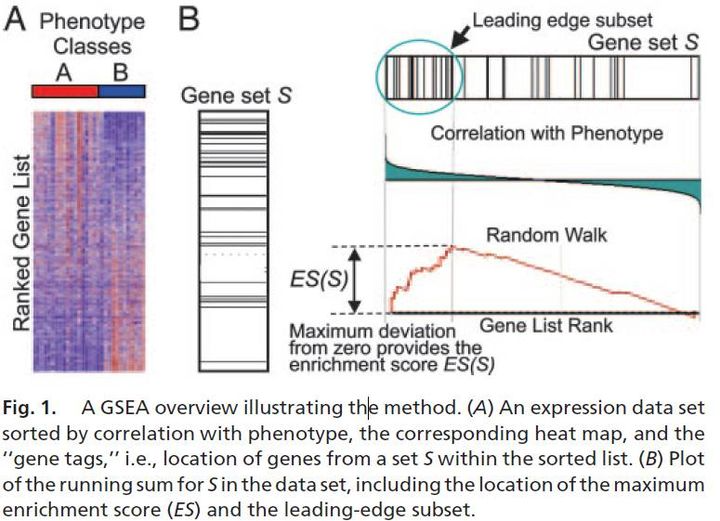
GSEA计算中几个关键概念:
- 计算富集得分** **(ES, enrichment score). ES反应基因集成员s在排序列表L的两端富集的程度。计算方式是,从基因集L的第一个基因开始,计算一个累计统计值。当遇到一个落在s里面的基因,则增加统计值。遇到一个不在s里面的基因,则降低统计值。
每一步统计值增加或减少的幅度与基因的表达变化程度(更严格的是与基因和表型的关联度,可能是fold-change,也可能是pearson corelation值,后面有介绍几种不同的计算方式)是相关的,可以是线性相关,也可以是指数相关 (具体见后面参数选择)。富集得分ES最后定义为最大的峰值。正值ES表示基因集在列表的顶部富集,负值ES表示基因集在列表的底部富集。 - 评估富集得分(ES)的显著性。通过基于表型而不改变基因之间关系的排列检验 (permutation test)计算观察到的富集得分(ES)出现的可能性。若样品量少,也可基于基因集做排列检验 (permutation test),计算p-value。
- 多重假设检验校正。首先对每个基因子集s计算得到的ES根据基因集的大小进行标准化得到Normalized Enrichment Score (NES)。随后针对NES计算假阳性率。(计算NES也有另外一种方法,是计算出的ES除以排列检验得到的所有ES的平均值)
- Leading-edge subset,对富集得分贡献最大的基因成员。

