- 文件位置: /data/doctor_offline/structured/noreview/
躁郁样
躁狂
行为及情绪异常
心境高涨
情绪起伏大
技术狂躁症
攻击行为
易激惹
思维奔逸
控制不住的联想
精神运动性兴奋
- csv文件的内容是该疾病对应的症状, 每种症状占一行.
- 文件位置: /data/doctor_offline/structured/noreview/躁狂症.csv
- 进行命名实体审核:
- 进行命名实体审核的工作我们这里使用AI模型实现, 包括训练数据集, 模型训练和使用的整个过程, 因此这里内容以独立一章的形成呈现给大家, 具体参见第五章: 命名实体审核任务.
- 代码位置: 在/data/doctor_offline/structured/reviewed/目录下执行.
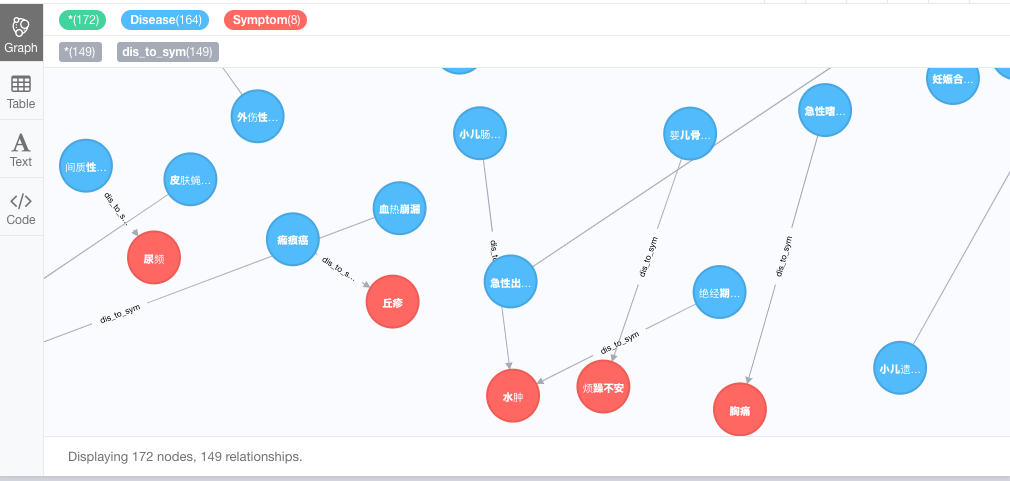
- 将命名实体写入图数据库的原因:
- 写入的数据供在线部分进行查询,根据用户输入症状来匹配对应疾病.
# 引入相关包
import os
import fileinput
from neo4j import GraphDatabase
from config import NEO4J_CONFIG
driver = GraphDatabase.driver( **NEO4J_CONFIG)
def _load_data(path):
"""
description: 将path目录下的csv文件以指定格式加载到内存
:param path: 审核后的疾病对应症状的csv文件
:return: 返回疾病字典,存储各个疾病以及与之对应的症状的字典
{疾病1: [症状1, 症状2, ...], 疾病2: [症状1, 症状2, ...]
"""
# 获得疾病csv列表
disease_csv_list = os.listdir(path)
# 将后缀.csv去掉, 获得疾病列表
disease_list = list(map(lambda x: x.split(".")[0], disease_csv_list))
# 初始化一个症状列表, 它里面是每种疾病对应的症状列表
symptom_list = []
# 遍历疾病csv列表
for disease_csv in disease_csv_list:
# 将疾病csv中的每个症状取出存入symptom列表中
symptom = list(map(lambda x: x.strip(),
fileinput.FileInput(os.path.join(path, disease_csv))))
# 过滤掉所有长度异常的症状名
symptom = list(filter(lambda x: 0<len(x)<100, symptom))
symptom_list.append(symptom)
# 返回指定格式的数据
return dict(zip(disease_list, symptom_list))
def write(path):
"""
description: 将csv数据写入到neo4j, 并形成图谱
:param path: 数据文件路径
"""
# 使用_load_data从持久化文件中加载数据
disease_symptom_dict = _load_data(path)
# 开启一个neo4j的session
with driver.session() as session:
for key, value in disease_symptom_dict.items():
cypher = "MERGE (a:Disease{name:%r}) RETURN a" %key
session.run(cypher)
for v in value:
cypher = "MERGE (b:Symptom{name:%r}) RETURN b" %v
session.run(cypher)
cypher = "MATCH (a:Disease{name:%r}) MATCH (b:Symptom{name:%r}) \
WITH a,b MERGE(a)-[r:dis_to_sym]-(b)" %(key, v)
session.run(cypher)
cypher = "CREATE INDEX ON:Disease(name)"
session.run(cypher)
cypher = "CREATE INDEX ON:Symptom(name)"
session.run(cypher)
# 输入参数path为csv数据所在路径
path = "/data/doctor_offline/structured/reviewed/"
write(path)