take “GSE53757” for example
不可以无脑运行!!! 注意注释的地方!
需要用到的R包
library(GEOquery)library(stringr)
(一)GEOquery下载芯片
gse_number = "GSE53757" # 这里改成需要下载的GSE编号eSet <- getGEO(gse_number,destdir = './import/GEO/',getGPL = F)eSet = eSet[[1]]
(二)临床数据整理
GEO数据库里,好像很少带有生存信息的,所以主要是为了分组
pd <- pData(eSet) # pd是包含所有临床信息的数据框group = ifelse(str_detect(pd$`tissue:ch1`, "normal"), "normal", "tumor") # 改!字符串匹配生成分组
(三)表达矩阵整理
主要是把探针ID转变成gene symbol,一共有四种方法,我只用到了第一种,其他几种,用到的时候再补充
exp <- exprs(eSet)exp = log2(exp+1)gpl_number <- eSet@annotation # 查看GPL,这个芯片是GPL570
3.1 BiocManager
在下面这个网址里查找gpl_number,得到第三列Bioconductor里的r包名称(箭头处)
网址:http://www.bio-info-trainee.com/1399.html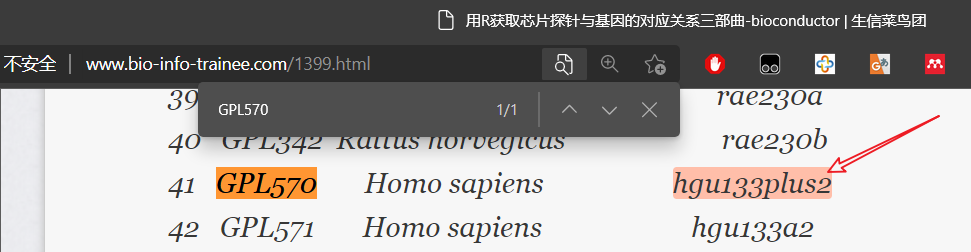
3.2 下载soft文件
在geo界面有一个表格
第一排的soft文件,下载,解压,从不已叹号开头的那一行往后,就是芯片注释,可以另存为csv方便读取,然后r语言替换行名就ok

