数据准备
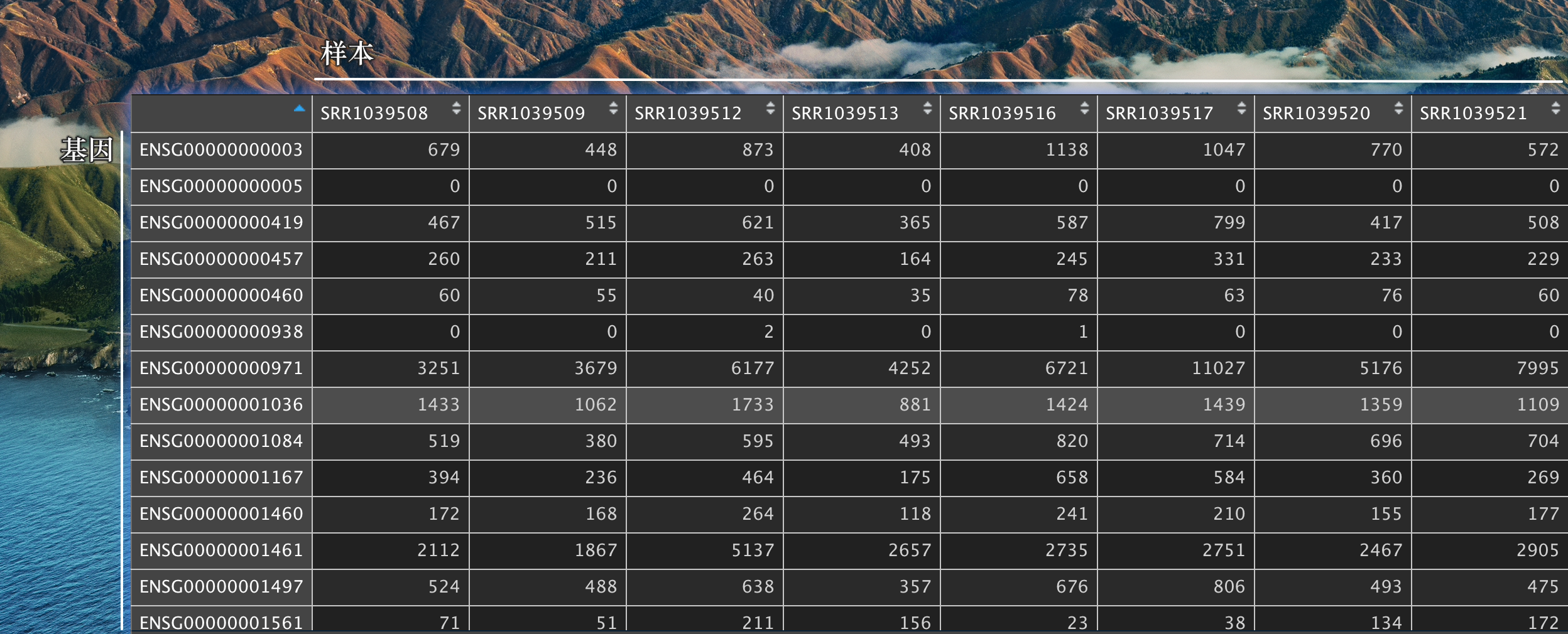
表达矩阵
一个表达矩阵,列为样本,行为基因
列名为样本名,行名为基因名,只能是以 SYMBOL 命名。
如果是Human, Mouse,Rat 之一,ENSEMBL 也是接受的。
如果不是,我们也提供了方便的工具函数用于ID转换,请参考工具-ID转换
我们使用airway 做为示例数据,数据结构是 data.frame
library(airway)data("airway")row_counts <- as.data.frame(assay(airway))
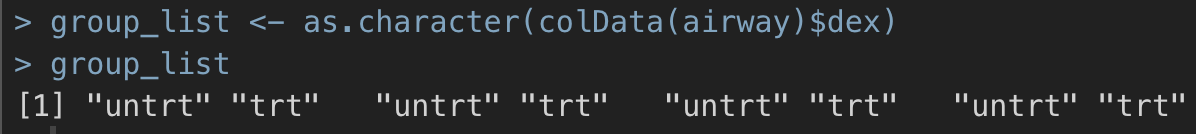
分组信息
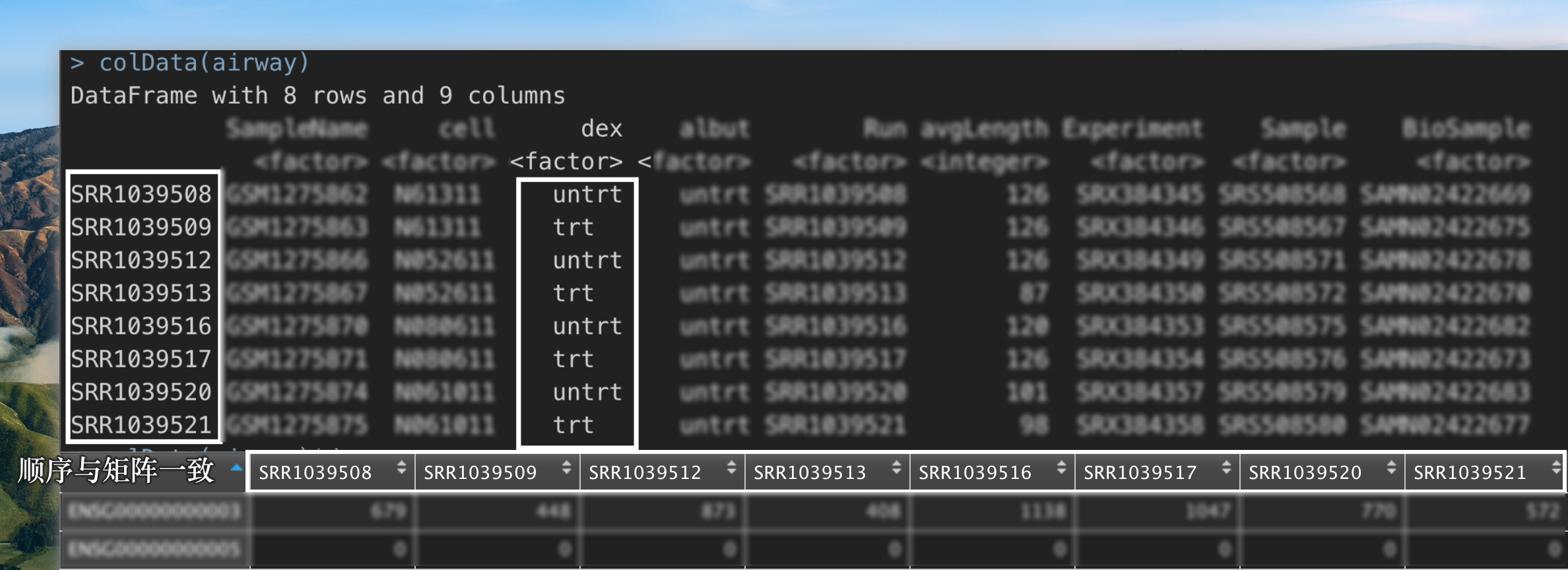
一个样本两分组的信息,必须是与矩阵列名顺序一致。
在airway 数据集中,我们可以得到这样一个分组信息,数据类型是 character,数据结构是 vector
group_list <- as.character(colData(airway)$dex)
数据过滤
可以看到一些基因在所有样本都是0,这种就要过滤掉。默认的参数就可以满足这个条件。filterMethod是用来过滤表达矩阵的
默认设置的是 "rowSums(expMatrix > 0) >= ncol(expMatrix)/2"
当然也可以根据自己的需求做一些其他修改。
物种设置
species参数是指定物种名称的。
不同物种对应着数据库里相应的数据。涉及的数据库请参考 数据库
三大物种
如果是 Human, Mouse,Rat 之一,只需要 species 参数就可以了。
airway 数据集的物种是人,那么就只需要把 species设置为 Human,并且结合上面准备好的数据。
还有需要注意的是 caseGroup 参数,也就是指明分组信息中实验组是哪一个。
data_i <- Create_DEGContainer(species = "Human",dataType = "Counts",idType = "ENSEMBL",expMatrix = row_counts,groupInfo = group_list,caseGroup = "trt")
其他物种
如果是其他物种,需要看情况而定,比如猪(pig),就需要更多参数设置。
OrgDb信息获取地址
https://bioconductor.org/packages/release/BiocViews.html#___OrgDb
如果没查到可以自己构建注释数据,通过eggnog一类的平台。
http://eggnog-mapper.embl.de/
organism 信息获取地址
https://www.genome.jp/kegg/catalog/org_list.html
如果没查到可以自己构建注释数据,通过eggnog一类的平台。
http://eggnog-mapper.embl.de/
https://www.genome.jp/tools/kofamkoala
msi_species信息获取代码msigdbr::msigdbr_species()
如果没有就别设置这个了。
这里是示例代码:
data_i <- Create_DEGContainer(species = "pig",dataType = "Counts",idType = "SYMBOL",expMatrix = row_counts,groupInfo = group_list,caseGroup = "NBW",OrgDb="org.Ss.eg.db",organism="ssc",msi_species="Sus scrofa")
或者是其他自己注释的数据。
其中 GOTERM2GENE GOTERM2NAME KEGGTERM2GENE KEGGTERM2NAME 对应不同的注释数据。
如何整理这些数据,可以参考 工具-注释数据整理
这里是示例代码:
data_i <- Create_DEGContainer(expMatrix = row_counts,groupInfo = group_list,caseGroup = "C",idType = "SYMBOL",species = "unknow",GOTERM2GENE = GO_anno,GOTERM2NAME = GO_description,KEGGTERM2GENE = KEGG_anno,KEGGTERM2NAME = KEGG_description)
当然KEGG 和 GO 是独立的,也就意味着你可以只自定义其中一个
比如:
data_i <- Create_DEGContainer(expMatrix = row_counts,groupInfo = group_list,caseGroup = "C",idType = "SYMBOL",species = "pig",GOTERM2GENE = GO_anno,GOTERM2NAME = GO_description,organism="ssc",msi_species="Sus scrofa)
当然实在没有相关数据也没关系,直接 不设置 即可。