批量绘制单基因相关性图
介绍
基因的表达调控是一个复杂的、动态的作用网络,具有
多向性、可逆性、多维性等特点,在庞大的基因表达中,可能某些协同(正协同、负协同)表达的基因共同参与了某个通路或功能的调控。
在转录组表达数据种寻找这些协同表达的基因有助于发现和挖掘更多的靶基因。我们可以计算两个基因之间的表达相关性来预测基因参与的分子功能或者潜在的调控作用。
在获得样本的转录组数据后,计算基因间的相关性,批量绘图,更加快速的寻找和筛选靶基因。
演练
我们绘制类似于下面这种类型的相关性散点图:
开始操作:
首先加载需要用到的 R 包:
# 批量绘制单基因相关性图# 加载R包library(tidyverse)library(ggplot2)library(ggpubr)library(cowplot)library(ggExtra)
读取表达量数据,一般是 normalized 之后的:
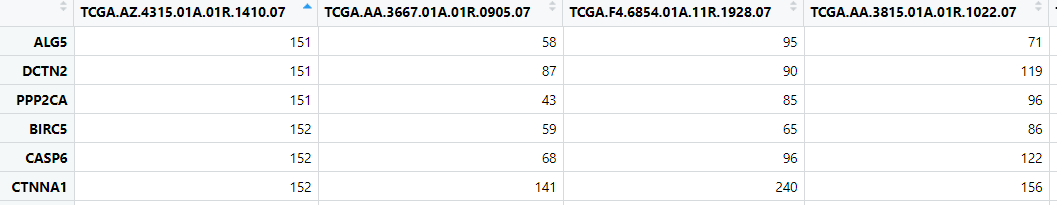
每行代表一个基因,每列代表一个样本。
# 设置工作路径setwd('C:/Users/admin/Desktop/')# 读取表达量矩阵expst <- read.delim('batch_cor.txt',header = T,row.names = 1)# 取对数new_exp <- log2(expst + 1)# 转置t_exp <- t(new_exp)# 转化为data.framet_exp <- as.data.frame(t_exp)# 选择目的基因target_gene <- 'MYC'
假如我们想看 MYC 基因和其它基因的相关性,接下来计算相关性系数:
# 计算每个基因与目的基因的相关性系数及p值cor_list <- list()for (i in colnames(t_exp)) {# 提取目的基因表达值tar <- t_exp[,target_gene]# 相关性检验cor_res <- cor(x = tar,y = t_exp[,i],method = 'pearson')# pvaluecor_pval <- cor.test(x = tar,y = t_exp[,i])$p.value# 合并结果final_res <- data.frame(tar_genename = target_gene,gene_name = i,cor_results = cor_res,cor_pvalue = cor_pval)# 储存cor_list[[i]] <- final_res}# 合并结果gene_corres <- do.call('rbind',cor_list)# 查看结果head(gene_corres,4)tar_genename gene_name cor_results cor_pvalueTMSB10 MYC TMSB10 -0.057883781 1.422938e-01ACTB MYC ACTB -0.007909501 8.412195e-01FTL MYC FTL 0.338104705 1.097893e-18S100A6 MYC S100A6 -0.030446676 4.405127e-01
筛选相关性系数较高或较低的基因:
# 筛选相关性系数> 0.7 或< -0.7 且p值小于0.05的基因high_cor <- gene_corres %>% filter(abs(cor_results) >= 0.3 ,cor_pvalue < 0.05 )# 保存基因名high_corgene <- high_cor$gene_name# 查看符合条件的基因数量length(high_corgene)[1] 252
可以看到筛选到 252 个符合条件的基因,接下来就是批量绘图的重头戏了:
# 批量绘图plotlst <- list()for (i in high_corgene) {# 绘图p <- ggplot(t_exp,aes_string(x = target_gene,y = i)) +geom_point(size = 2,color = '#EC0101',alpha = 0.5) +theme_bw() +# 主题细节调整theme(axis.title = element_text(size = 16),axis.text = element_text(size = 14),axis.ticks.length = unit(0.25,'cm'),axis.ticks = element_line(size = 1),panel.border = element_rect(size = 1.5),panel.grid = element_blank()) +# 添加回归线geom_smooth(method = 'lm',se = T,color = '#F9B208',size = 1.5,fill = '#FEA82F') +# 添加相关性系数及p值stat_cor(method = "pearson",digits = 3,size=6)# 添加边际柱形密度图p1 <- ggMarginal(p,type = "densigram",xparams = list(binwidth = 0.1, fill = "#B3E283",size = .7),yparams = list(binwidth = 0.1, fill = "#8AB6D6",size = .7))# 保存在list里plotlst[[i]] <- p1}# 拼图,4个一行allplot <- plot_grid(plotlist = plotlst,ncol = 4 ,align = "hv")
这里所有的图以 list 类型储存在 plotlst 里,一般不建议在绘图面板展示,图太多了直接会卡,我们可以保存下来:
# 计算高度hei = length(plotlst)/4*5# 保存图片,时间比较久ggsave(allplot,filename = 'all-corrplot.pdf',limitsize = FALSE,width = 20,height = hei)
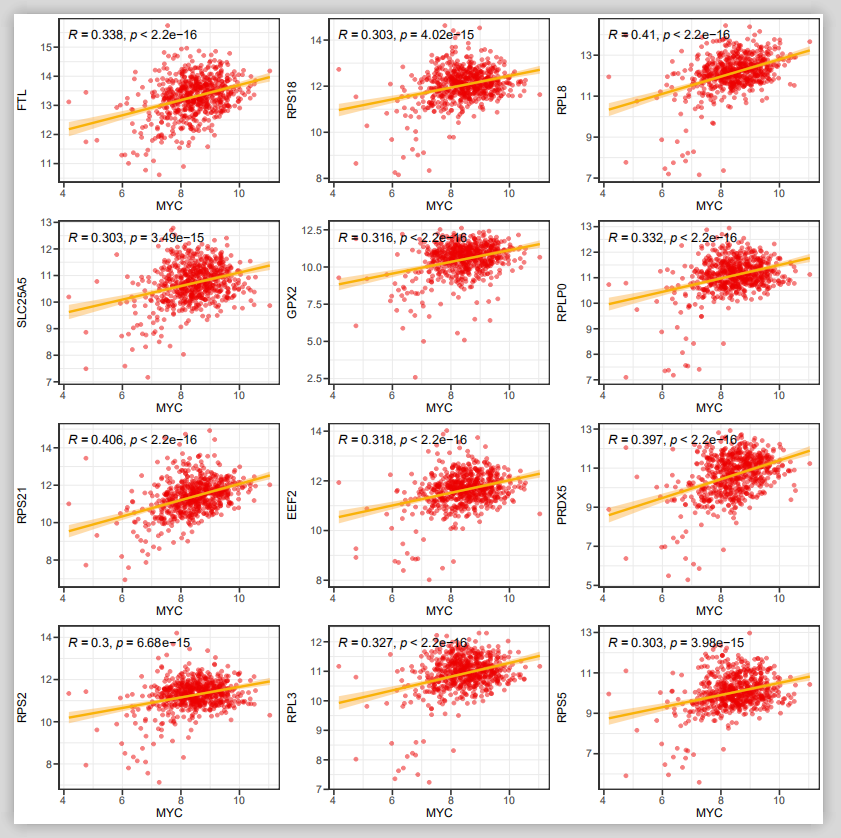
看看前两行结果:
nice!不错。
如果全部结果保存在一个文件里太大,同时运行的时间也比较久的话,我们可以每个 pdf 文件存成一定数量的图,结果多生成一些 pdf 文件,这样也可以减少一些时间,假如我们可以没 12 个图保存为一个文件 pdf:
# 改进,每12个保存为一个pdf文件for (n in seq(12,252,12)) {print(n)# 切分起始位置nstart <- n - 11# 1、切分plotlsttmp_list <- plotlst[nstart:n]# 2、拼图temp_plot <- plot_grid(plotlist = tmp_list,ncol = 4 ,align = "hv")# 3、保存图片ggsave(temp_plot,filename = paste(nstart,n,'corrplot.pdf',sep = '_'),width = 20,height = 20)}[1] 12[1] 24[1] 36...
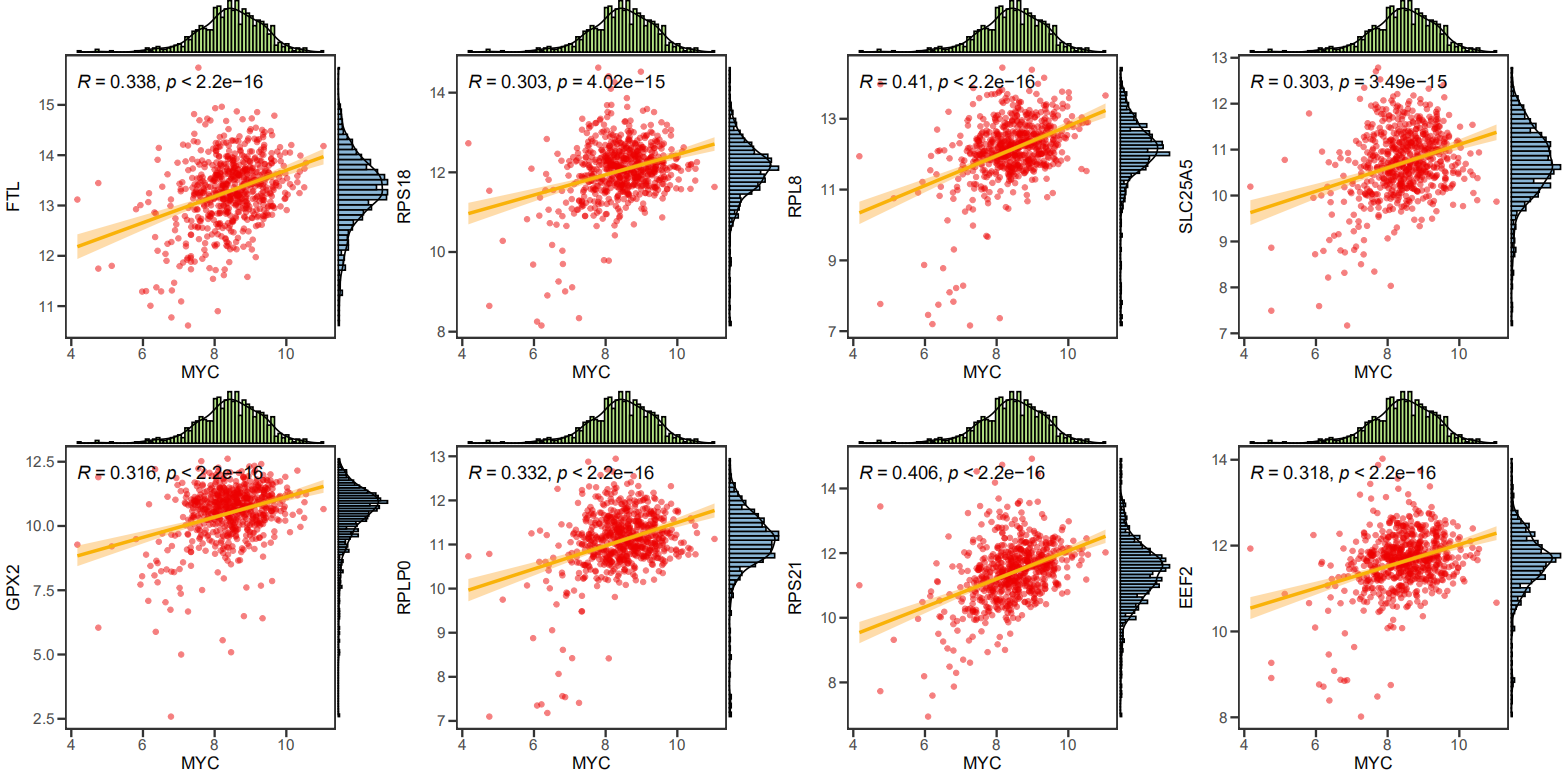
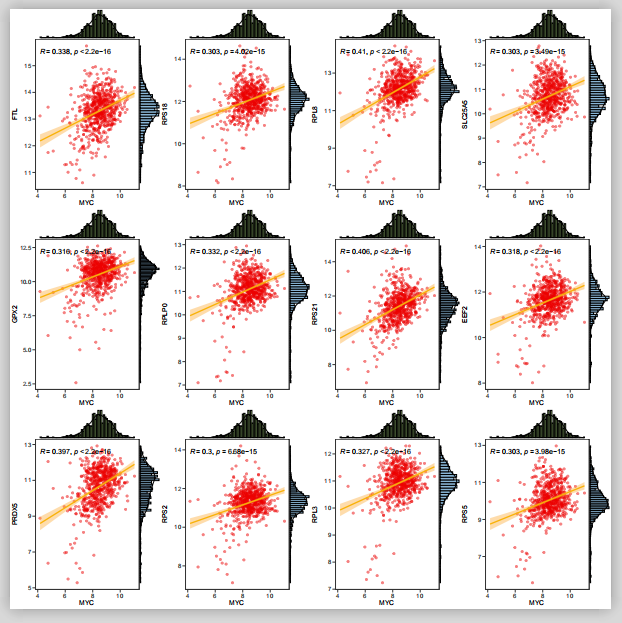
这样就很快画完啦!产生了 21 个 pdf 文件,打开一个看看结果:
图小了柱形图就看着太密:
尝试把柱形图去掉试试: