批量读取文件一文搞定
提高效率,解放双手
对于新手小白来说,使用 R 来 读取文件 是 第一个要过的门槛 ,当初我也是在这门槛绊倒过数次,不过多多练习,总结经验肯定会越来越熟练的。
当你有一两个或者两三个文件需要读取时,想必会一个个读进来就完事了,最多复制几行代码,改一下文件名咯。但是如果有成百上千个呢?那不是 废手废眼睛?
昨天群里有小伙伴问怎么批量读取文件,然后把文件一个个命名保存到环境变量里。写了一个批量读取的代码发在了群里,今天总结一下批量读取的多种方式。
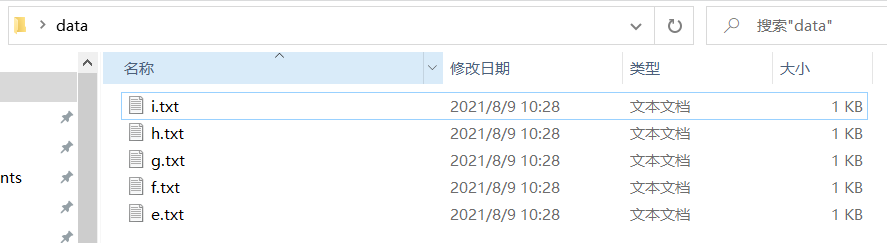
我在桌面建了 dada 文件夹,里面有 5 个测试 txt 文件:
1、批量读取保存到环境变量
这里先实现批量读取再赋值保存到环境变量的操作。
我觉得如果每个文件内容不一样,这样是可以的,但是文件如果太多,都保存到环境变量容易发生 变量名重名 之类的错误:
# 加载R包library(dplyr)# 设置工作路径setwd('C:\\Users\\admin\\Desktop\\data')# 查看文件dalst <- list.files(pattern = '*.txt')dalst[1] "e.txt" "f.txt" "g.txt" "h.txt" "i.txt"
使用基础 for 循环读取:
# for 循环for(i in 1:length(dalst)){# 名称向量name = c("a", "b", "c", "d","e")# 读取并赋值assign(name[i],read.table(dalst[i],header = T))}
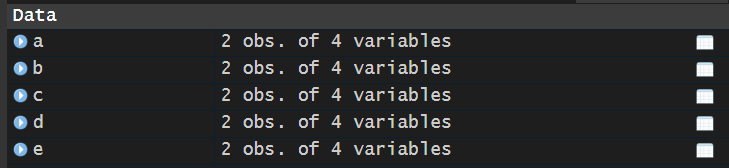
可以看到读取进来并且命好名保存到环境变量里了。
使用 lapply 版:
# lapply 版dalst <- list.files(pattern = '*.txt') %>% lapply(.,read.table,header =T)for (i in 1:length(dalst)) {# 名称向量name <- c("a", "b", "c", "d","e")# 赋值assign(name[i],dalst[[i]])}
2、批量读取保存到 list
为避免储存太多环境变量或者发生错误,建议保存到 list 里,需要用的时候直接取出来即可。
dalst <- list.files(pattern = '*.txt') %>% lapply(.,read.table,header =T)dalst[[1]]a ay b by1 -1.5 -1.3 1.4 1.82 -1.6 -1.6 1.6 1.5[[2]]a ay b by1 -1.5 -1.3 1.4 1.82 -1.6 -1.6 1.6 1.5...
用 lapply 读取返回的直接是一个 list 的类型,但是每个 list 的名字是 1、2、3、4 的格式,我们只需要把名字改成对应的即可:
# 命名listnames(dalst) <- c("a", "b", "c", "d","e")dalst$aa ay b by1 -1.5 -1.3 1.4 1.82 -1.6 -1.6 1.6 1.5$ba ay b by1 -1.5 -1.3 1.4 1.82 -1.6 -1.6 1.6 1.5...
mapply 版:
# mapply 版# 文件名dalst <- list.files(pattern = '*.txt')# 名称向量name <- c("a", "b", "c", "d","e")res <- mapply(function(n,d){n <- read.table(d,header = T)},name,dalst,SIMPLIFY = F)res$aa ay b by1 -1.5 -1.3 1.4 1.82 -1.6 -1.6 1.6 1.5$ba ay b by1 -1.5 -1.3 1.4 1.82 -1.6 -1.6 1.6 1.5...
3、批量读取合并保存到数据框
如果每个数据的 列名一样 ,只是 记录的内容不一样 ,我们更希望批量读取进来并且 合并到一个表格里面 ,这也是批量读取最常用的方式,并且结果可读性更好:
do.call 合并数据:
# do.calllist.files(pattern = '*.txt') %>% lapply(.,read.table,header =T) %>%do.call(rbind,.)a ay b by1 -1.5 -1.3 1.4 1.82 -1.6 -1.6 1.6 1.53 -1.5 -1.3 1.4 1.8...
Reduce 合并数据:
# Reducelist.files(pattern = '*.txt') %>% lapply(.,read.table,header =T) %>%Reduce(rbind,.)a ay b by1 -1.5 -1.3 1.4 1.82 -1.6 -1.6 1.6 1.53 -1.5 -1.3 1.4 1.8...
purrr 包 map_dfr 函数直接内部合并,不需要再合并,简化操作:
# map 函数library(purrr)list.files(pattern = '*.txt') %>% map_dfr(.,read.table,header =T)a ay b by1 -1.5 -1.3 1.4 1.82 -1.6 -1.6 1.6 1.53 -1.5 -1.3 1.4 1.8...
plyr 包和 map_dfr 有异曲同工之妙,也是非常的方便:
# plyr 包library(plyr)list.files(pattern = '*.txt') %>% ldply(.,read.table,header =T)a ay b by1 -1.5 -1.3 1.4 1.82 -1.6 -1.6 1.6 1.53 -1.5 -1.3 1.4 1.8...
现在批量读取再也不用麻麻担心你不会了。

