主要梳理的是10X Visium的流程相关知识。
空间转录组可测量完整组织切片中的总mRNA,并绘制出现基因活性的位置的图。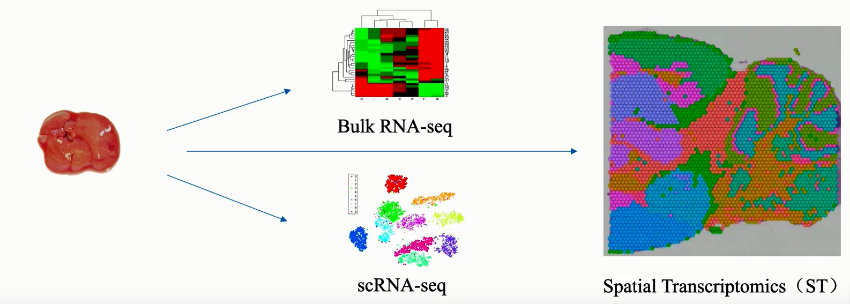
在癌症的研究中,当免疫细胞浸润到肿瘤组织中和未浸润到时,对于免疫抑制类药物的反应不同。空间转录组分析能够解决相关问题。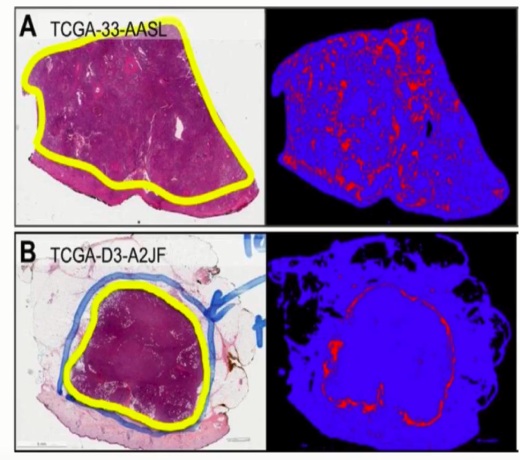
实验原理与流程:
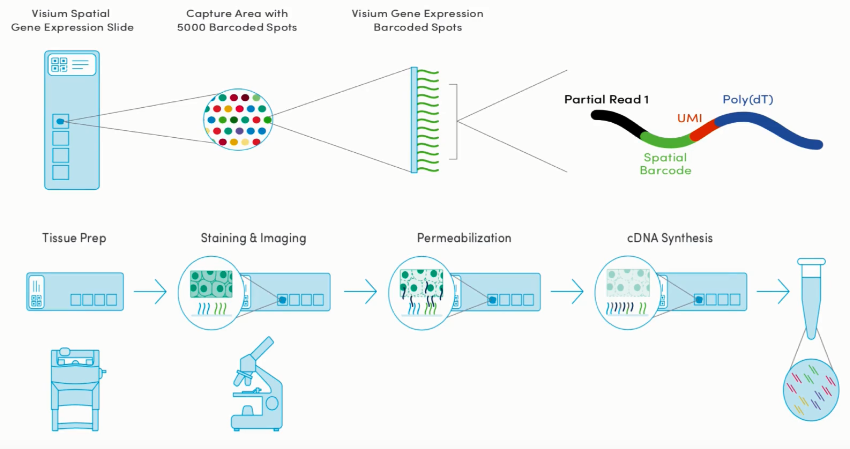
在玻片上原位合成序列,设置的序列上有对应的空间信息。一个玻片上大概有5000个Spots。一般一个Spots中会有1-10个细胞,但是视具体切片组织而言。
测序结果处理

数据格式: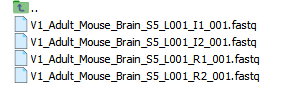
空间转录组表达矩阵的获取可以利用Space ranger进行处理。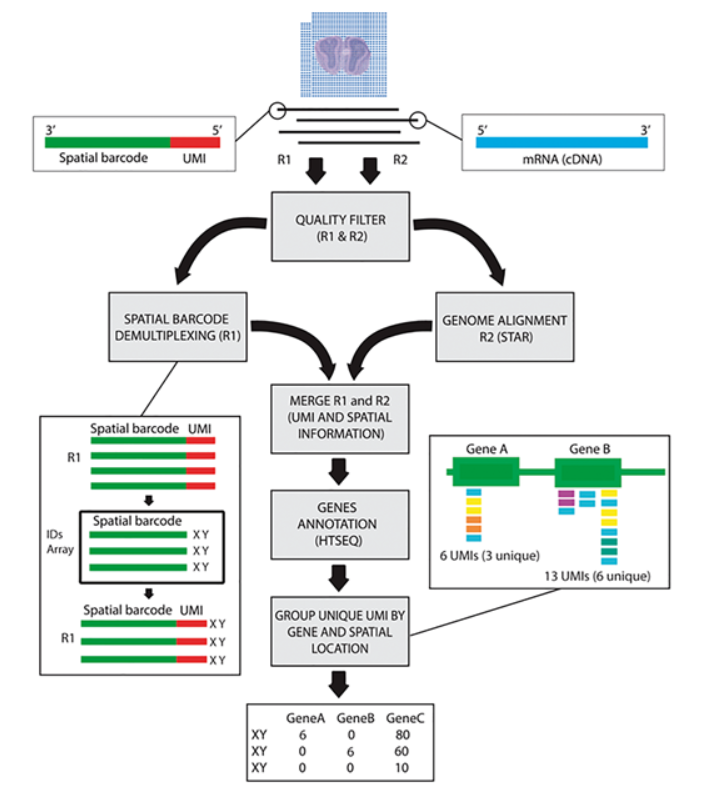
SpaceRanger的结果:
├── analysis|---├── clustering│---├── diffexp│---├── pca│---├── tsne│---└── umap├── cloupe.cloupe├── filtered_feature_bc_matrix ##需要的表达矩阵结果│---├── barcodes.tsv.gz│---├── features.tsv.gz│---└── matrix.mtx.gz├── filtered_feature_bc_matrix.h5 ##需要的表达矩阵结果├── metrics_summary.csv├── molecule_info.h5├── possorted_genome_bam.bam├── possorted_genome_bam.bam.bai├── raw_feature_bc_matrix│---├── barcodes.tsv.gz│---├── features.tsv.gz│---└── matrix.mtx.gz├── raw_feature_bc_matrix.h5├── spatial # 空间信息全在这 :这些文件是用户提供的原始全分辨率brightfield图像的下采样版本。下采样是通过box滤波实现的,它对全分辨率图像中像素块的RGB值进行平均,得到下采样图像中一个像素点的RGB值。│---├── aligned_fiducials.jpg 这个图像的尺寸是tissue_hires_image.png。由基准对齐算法发现的基准点用红色高亮显示。此文件对于验证基准对齐是否成功非常有用。│---├── detected_tissue_image.jpg│---├── scalefactors_json.json│---├── tissue_hires_image.png 图像的最大尺寸为2,000像素│---├── tissue_lowres_image.png 图像的最大尺寸为600像素。│---└── tissue_positions_list.csv└── web_summary.html
ST的分析思路
空间转录组的常规的分析思路:
因为每个Spots中的细胞量较少,所以讲每个Spot看作是一个细胞进行分析。然后参考ScRNA-seq的思路进行分析。
利用每个Spot的分子特征对Spots进行聚类,聚类成多个亚群。因为组织相同区域的表达谱具有一定的相似性,所以相同组织区域的spot更容易被聚成同一个亚群。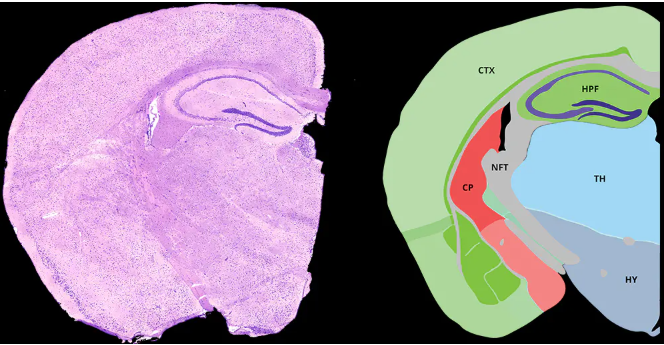
STX1A在皮质,Prkcd在丘脑,HPCA在海马,Prnch在下丘脑。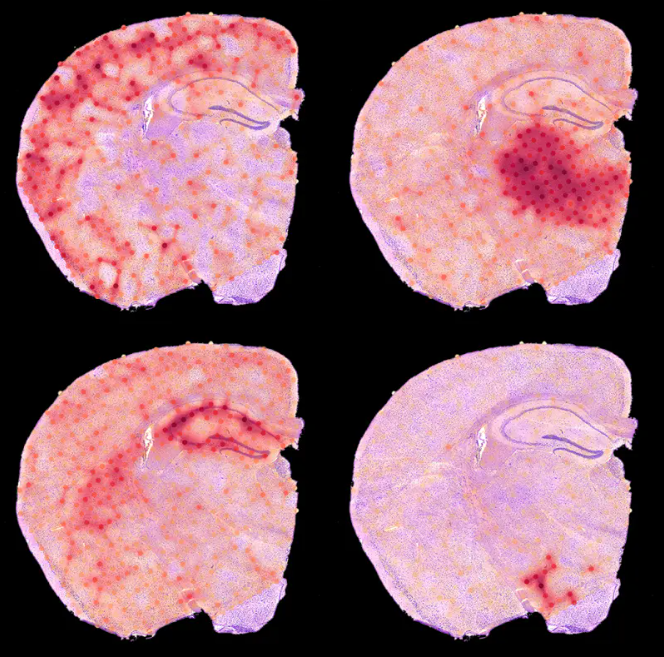
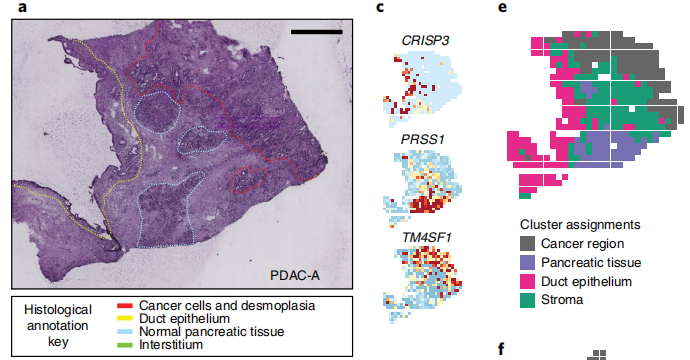
以分子特征聚类的Spot亚群和解剖学组织区域划分具有较高的重合度。因此对于spot亚群进行分子特征分析可以近似的看成是不同组织区域的分组特征分析。
(1)亚群鉴定
与scRNA-seq数据类似,基于分子特征对Spots亚群进行聚类(本质还是数学聚类)。基于切片的HE染色结果与Spot位置进行对比,对识别出的类进行注释。
(2)亚群特征基因分析
因为Spots亚群与解剖区域有重合,为了了解解剖区域的分子特征,可以直接研究对应亚群的特征基因。和ScRNA-seq不一样的是,单细胞一般会关注细胞间的差异,ST-seq分析应该考虑切片的背景信息,研究的是组织不同区域间的差异他们的空间异质性。对于异质性高的组织一般应该侧重与同切片分析。对于心脏之类的正常组织样本异质性不强,可以混合多切片进行分析。
Seurat分析流程
1.数据读取
读取SpaceRanger处理后的数据。
2.数据过滤
和单细胞数据处理一样,需要对数据进行过滤。通过UMI,nGene,线粒体比例对数据过滤。
问题:过滤后可能导致切片上有部分区域无数据;不过滤数据质量会较低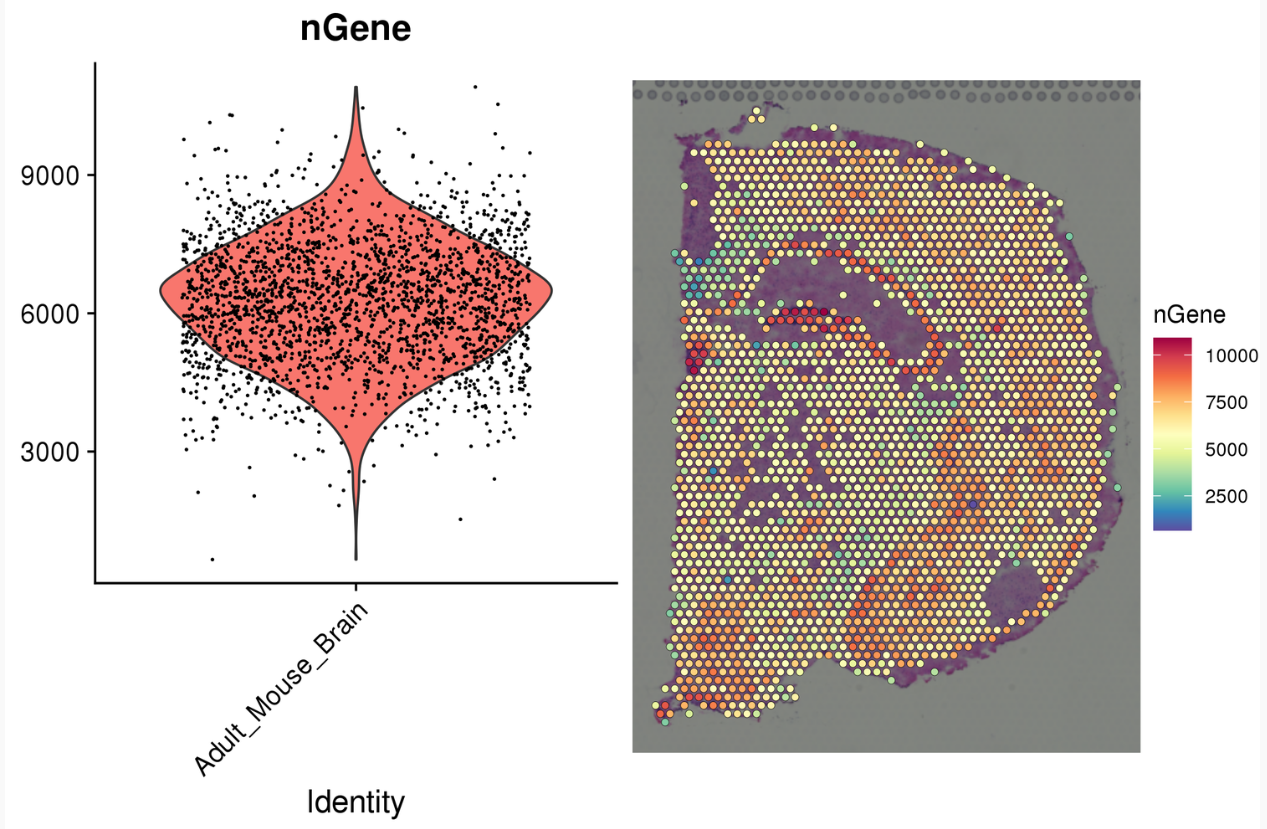
3.数据预处理
需要对数据进行标准化处理,从而减少测序深度带来的影响
同时,因为组织中细胞的密度差异原因,使得数据存在异质性,所以需要进行有效的Normalization,但是因为组织特异性的原因,常规的标准化方法(LogNormalize)不适用。
Seurat推荐使用SCTransform()进行标准化处理
4.Spots聚类
和单细胞的流程相似,利用特征基因对所有Spot进行聚类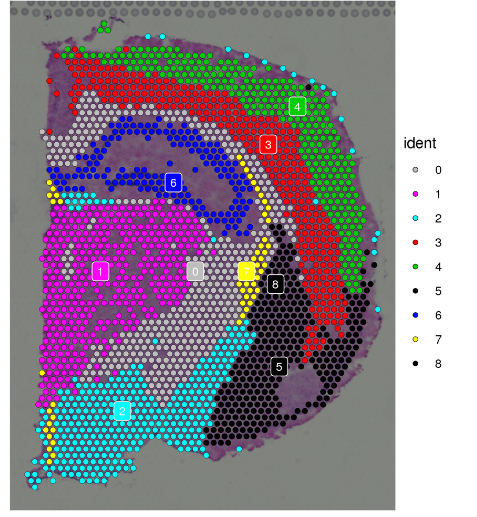
5.Marker基因识别
第一种是基于组织内预先注释的解剖区域来进行差异表达(对于有切片组织特征),可以和单细胞的流程相似。利用FindMarkers去识别差异marker。
第二种是在没有预先注释的情况下,寻找显示空间模式的特征。利用FindSpatiallyVariables函数进行实现。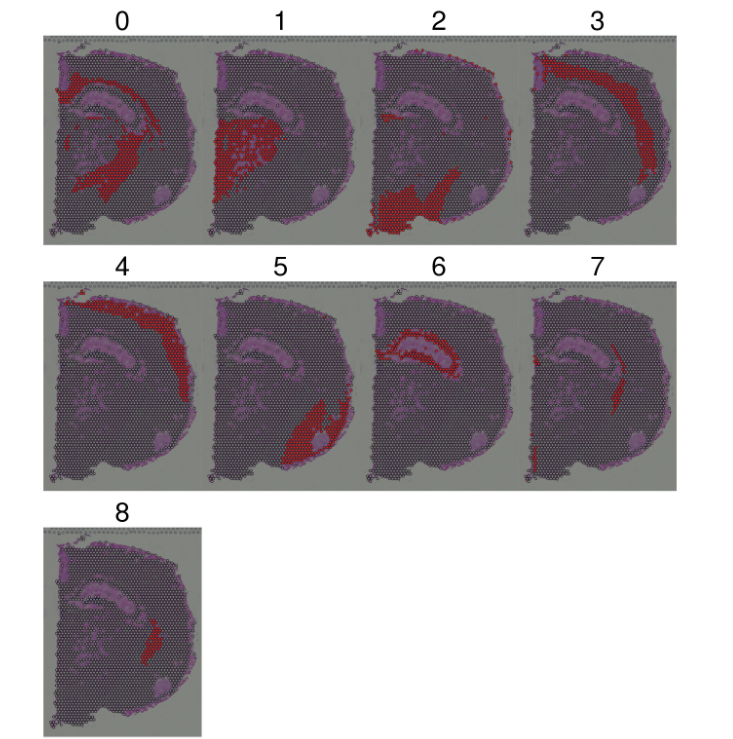
6.Marker基因的常规分析
GO分析
KEGG分析
PPI分析
TFBS分析
多样本合并分析
对于多个切片组织,可以使用Seurat中的merge进行合并。然后对Spot进行聚类分析等基础分析。
也可以使用Splotch进行分析:
1.表达量归一化,综合使用每个Spot位置信息,UMI等信息,将表达量归一化为表达量值/所有切片测序深度中位数值。 会排除spots测序深度的影响,排除测序误差的影响,降低数据复杂程度。
2.共表达分析,对于基因进行聚类,将基因分为多个模块
3.模块功能鉴定,对于识别出的模块进行功能富集分析,探索模块基因的潜在功能。
问题
1.现在的常规分析思路,是将单spot看作单个细胞,但是单个spot中一般是多个细胞。
2.常规分析中使用了基因表达信息,但是对于空间位置信息未充分使用。
3.有些基因在整个切片上都是高表达,有些基因只在某块区域表达,如何利用空间位置信息去充分挖掘特征基因信息。

