摘要:在构建CT图像深度学习模型时,往往要对原始的CT图像DICOM文件进行预处理,本文描述了使用Python语言对DICOM文件进行批量处理的一些步骤。
一、基本概念介绍
- CT值
CT值是定义的一种用来定量衡量组织对X光吸收率的计量单位,可用来测定人体组织或器官的密度,单位为HU。
我们读取DICOM格式的CT图像中CT值的取值范围取决于扫描仪 (scanner)的位数,有一半左右的csanner为12bits,因此当截距为-1024时,范围可达到-1024~+3072
- 窗宽(WW)与窗位(WL)
CT能识别人体内2000个不同灰阶的密度差别,而如下图所示,人的眼睛只能分辨16 个灰阶度,因此人眼在CT图像上能分辨的CT值为125 Hu ( 2000 / 16 )。进行分段观察才能凸显出CT 的优点。观察的CT值范围为窗宽(WW) ,观察的中心CT值即为窗位或窗中心(WL)。
窗宽和窗位如何选定呢?窗位应该接近需要观察的CT 值,窗宽要能反映该组织或病变的CT 值变化范围。通常查看不同部位的CT图像,有与之相对应的固定窗宽和窗位值。
RadiAnt DICOM Viewer是一款常用的DICOM格式文件查看软件,其对几种常见部位的窗宽和窗位值设定如下:
比如对腹部CT影像进行查看,则将窗位设置为60,窗宽设定为400。
PS:
- 在Kaggle上看到一篇有关CT DICOM文件窗位、窗宽探索的Notebook,可以供参考学习https://www.kaggle.com/redwankarimsony/rsna-str-pe-gradient-sigmoid-windowing
- 在Radiopaedia上看到对Windowing的介绍,可供学习,极具参考价值:https://radiopaedia.org/articles/windowing-ct
- DICOM格式
DICOM是Digital Imaging and Communications in Medicine的缩写,为医学图像的相关信息的国际标准,它定义了质量能满足临床需要的可用于数据交换的医学图像格式。
DICOM文件由头信息(tag)以及数据两部分组成。数据部分存储的是原始像素值(pixel value),本身是没有意义的,需要转换为以HU为单位的CT值,转换公式如在所示:
CTvalue是第i个像素的CT值,pixelValue是第i个像素的灰度值,rescaleSlope与rescaleIntercept是Tag中的两个值,可以从DICOM的头信息中获取。
RadiAnt DICOM Viewer对于浏览查看患者的一系列DICOM文件比较适合,但将CT图像输入到模型前的一些预处理,使用python进行批量处理会更高效,也会降低由不同人使用软件预处理时造成的人为偏差。接下来两部分会介绍使用python中的pydicom进行DICOM文件的分析处理。
二、相关库的安装
- pydicom (一个用于处理DICOM格式文件的Python包)
使用conda进行python库的管理,相关配置可参见Windows10系统使用tensorflowGPU中的描述。
conda install -c conda-forge pydicom
在读取DICOM医疗图像时,如果不涉及到压缩格式,一般pydicom就能处理并得到pixel_array。但有时我们会遇到有压缩格式的dcm图像,于是需要结合其他package来处理。
- gdcm (用于读取压缩的DICOM格式文件)
conda install -c conda-forge gdcm
三、DICOM数据读取和分析
公共数据库
3D-IRCADb 01是一个公共数据库,包含了10位男性和10位女性的CT图像,其中75%的样本患有肝细胞癌(HCC),并且包含相对应的肝脏mask文件,适合用于训练腹部CT肝脏自动分割模型。数据库中CT存储格式为DICOM。
import pydicom as dicomimport numpy as nptest_path ='./data/3Dircadb1.1/PATIENT_DICOM/image_70' #DICOM文件地址image_ds = dicom.read_file(test_path) # 读取DICOM文件,或dicom.dcmread()print(image_ds.dir())#查看全部属性
['AccessionNumber', 'AcquisitionDate', 'AcquisitionTime', 'BitsAllocated', 'BitsStored', 'Columns', 'HighBit', 'ImageOrientationPatient', 'ImagePositionPatient', 'ImageType', 'InstanceNumber', 'InstitutionName', 'Modality', 'PatientBirthDate', 'PatientID', 'PatientName', 'PatientSex', 'PhotometricInterpretation', 'PixelData', 'PixelRepresentation', 'PixelSpacing', 'ReferringPhysicianName', 'RescaleIntercept', 'RescaleSlope', 'Rows', 'SOPClassUID', 'SOPInstanceUID', 'SamplesPerPixel', 'SeriesInstanceUID', 'SeriesNumber', 'SliceThickness', 'SpacingBetweenSlices', 'StudyDate', 'StudyID', 'StudyInstanceUID', 'StudyTime', 'WindowCenter', 'WindowCenterWidthExplanation', 'WindowWidth']
print(image_ds) #查看全部字典信息
(0008, 0008) Image Type CS: ['ORIGINAL', 'PRIMARY'](0008, 0016) SOP Class UID UI: CT Image Storage(0008, 0018) SOP Instance UID UI: 1.2.826.0.1.3680043.2.1125.3960029311677393949124574691280653592(0008, 0020) Study Date DA: '20090527'(0008, 0022) Acquisition Date DA: '20090527'(0008, 0030) Study Time TM: '125005'(0008, 0032) Acquisition Time TM: '125005'(0008, 0050) Accession Number SH: ''(0008, 0060) Modality CS: 'CT'(0008, 0080) Institution Name LO: 'IRCAD'(0008, 0090) Referring Physician's Name PN: ''(0010, 0010) Patient's Name PN: 'liver_01^patient'(0010, 0020) Patient ID LO: ''(0010, 0030) Patient's Birth Date DA: '20090527'(0010, 0040) Patient's Sex CS: 'M'(0018, 0050) Slice Thickness DS: "1.600000023841858"(0018, 0088) Spacing Between Slices DS: "1.600000023841858"(0020, 000d) Study Instance UID UI: 1.2.826.0.1.3680043.2.1125.6907248010745639067628287040727898539(0020, 000e) Series Instance UID UI: 1.2.826.0.1.3680043.2.1125.9647753419956608505832502991212815128(0020, 0010) Study ID SH: ''(0020, 0011) Series Number IS: None(0020, 0013) Instance Number IS: "70"(0020, 0032) Image Position (Patient) DS: [0, 0, 112.00000166893](0020, 0037) Image Orientation (Patient) DS: [1, 0, 0, 0, 1, 0](0028, 0002) Samples per Pixel US: 1(0028, 0004) Photometric Interpretation CS: 'MONOCHROME2'(0028, 0010) Rows US: 512(0028, 0011) Columns US: 512(0028, 0030) Pixel Spacing DS: [0.569999992847443, 0.569999992847443](0028, 0100) Bits Allocated US: 16(0028, 0101) Bits Stored US: 16(0028, 0102) High Bit US: 15(0028, 0103) Pixel Representation US: 1(0028, 1050) Window Center DS: "0.0"(0028, 1051) Window Width DS: "0.0"(0028, 1052) Rescale Intercept DS: "0.0"(0028, 1053) Rescale Slope DS: "1.0"(0028, 1055) Window Center & Width Explanation LO: ''(7fe0, 0010) Pixel Data OW: Array of 524288 elements
# tag中和pixel value转换相关的两个值
rescaleIntercept = np.float(image_ds.RescaleIntercept)
rescaleSlope = np.float(image_ds.RescaleSlope)
print ("rescaleIntercept:%.3f & rescaleSlope:%.3f" %(rescaleIntercept,rescaleSlope))
rescaleIntercept:0.000 & rescaleSlope:1.000
以上是对DICOM文件中tag信息的提取,接下来是对pixel数据的提取。
pixel_img = image_ds.pixel_array # 读取pixel value
ct_img = pixel_img*rescaleSlope + rescaleIntercept #转换为ct value
# 定义WW/WL窗口转换函数
def setDicomWinWidthWinCenter(img_data, winwidth, wincenter, rows, cols):
img_temp = img_data
#img_temp.flags.writeable = True
min = (2 * wincenter - winwidth) / 2.0 + 0.5
max = (2 * wincenter + winwidth) / 2.0 + 0.5
dFactor = 255.0 / (max - min)
for i in np.arange(rows):
for j in np.arange(cols):
img_temp[i, j] = int((img_temp[i, j]-min)*dFactor)
min_index = img_temp < 0
img_temp[min_index] = 0
max_index = img_temp > 255
img_temp[max_index] = 255
return img_temp
#对于腹部CT,设置窗宽为400,窗位为60
abdomen_img = setDicomWinWidthWinCenter(ct_img,400,60,512,512)
- 从医院收集到的CT图像
从医院收集到的CT图像同样为DICOM格式,但与公共数据集有所不同,这些数据是压缩过的DICOM格式,如果不导入gdcm库,会出现报错,找不到依赖库GDCM。
import pydicom as dicom
import gdcm
import numpy as np
ourdata = './门脉.dcm'
ds = dicom.dcmread(ourdata)
rescaleIntercept = np.float(ds.RescaleIntercept)
rescaleSlope = np.float(ds.RescaleSlope)
print ("rescaleIntercept:%.3f & rescaleSlope:%.3f" %(rescaleIntercept,rescaleSlope))
rescaleIntercept:-1024.000 & rescaleSlope:1.000
在本例中,Rescale Intercept为1024,和公共数据集中的0有所不同。
pixel = ds.pixel_array # 读取pixel value
ct = pixel*rescaleSlope + rescaleIntercept #转换为ct value
abdomen = setDicomWinWidthWinCenter(ct,400,60,512,512)
将使用固定窗位和窗宽的CT图像保存为jpg格式的文件。
import matplotlib.pyplot as plt
plt.imsave('abdomen_门脉.jpg', abdomen, format='jpg', cmap='gray')
输出的图像如下图所示: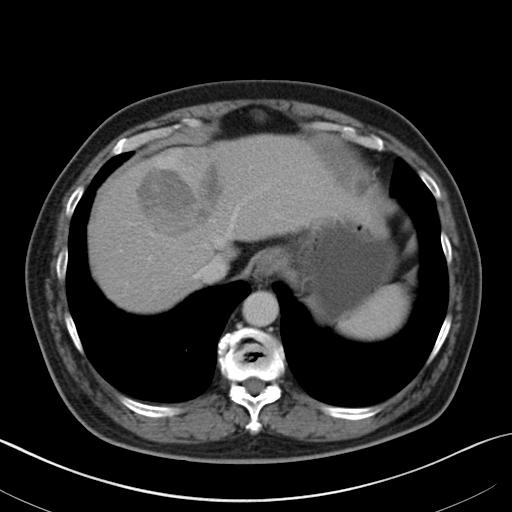
之后可以通过写python脚本,对大量的原始CT的DICOM文件进行批量预处理。
- RTSTRUCT类型DICOM文件的读取与解析

