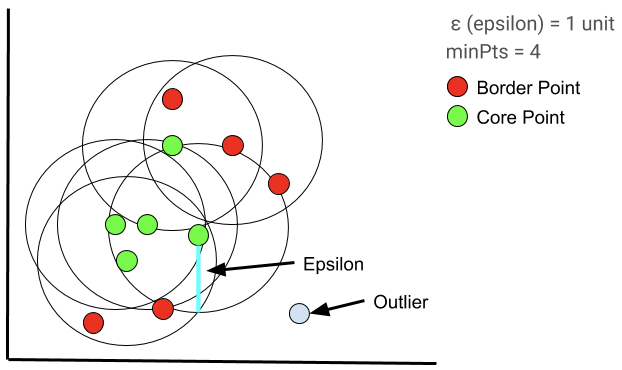
DBSCAN:Density-Based Spatial Clustering of Applications with Noise,具有噪声的基于密度的聚类方法。
DBSCAN 是一种基于密度的聚类算法,这类密度聚类算法一般假定类别可以通过样本分布的紧密程度决定。同一类别的样本,他们之间是紧密相连的,也就是说,在该类别任意样本周围不远处一定有同类别的样本存在。
通过将紧密相连的样本划为一类,这样就得到了一个聚类类别。通过将所有各组紧密相连的样本划为各个不同的类别,则我们就得到了最终的所有聚类类别结果。
前置概念
假设样本集为 ,定义下面几个概念:
-邻域:对于
,其
-邻域包含样本集
中与
的距离不大于
的样本(包括
自己),即
,这个子样本集的个数记为
- 核心点(core point):对于任
,如果其
-邻域至少包含
个样本,即
,则
是一个核心点。
- 边界点(border point):对于任
,若其不是一个核心点,但位于一个或多个核心点的
-邻域内,则
是一个边界点。
离群点(outlier):对于任
,若其既不是核心点也不是边界点,则它是一个离群点。
密度直达(directly density-reachable):若
位于
的
-邻域中,且
是一个核心点,则称
由
密度直达。注意:除非
也是核心点,否则此时不能说
由
密度直达,密度直达不具有对称性。
- 密度可达(density-reachable):对于
和
,若存在样本序列
,其中
,
,且
由
密度直达,则称
由
密度可达。此时序列中的传递样本
均为核心点,因为只有核心点才能使其他样本密度直达。注意:密度可达也不满足对称性,这个可以由密度直达的不对称性得出。
- 密度相连(density-connected):对于
和
,若存在
使得
和
均由
密度可达,则称
和
密度相连。注意:密度相连是满足对称性的。
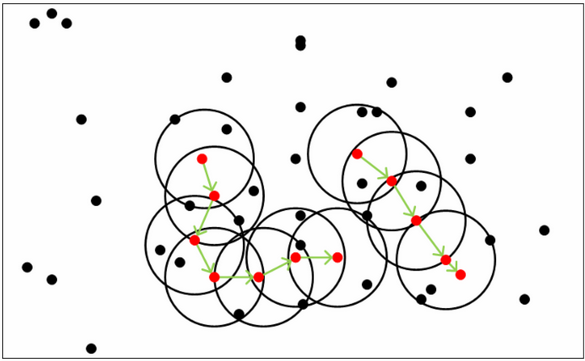
以下图为例,图中。红色的点都是核心点,因为其
-邻域至少有 5 个样本,黑色的样本是非核心点。所有核心点密度直达的样本在以红色核心点为中心的超球体内,如果不在超球体内,则不能密度直达。图中用绿色箭头连起来的核心点组成了密度可达的样本序列,在这些密度可达的样本序列的
-邻域内所有的样本相互都是密度相连的。
算法原理
DBSCAN 的原理描述起来很简单:任选一个没有类别的核心点作为种子,找到该核心点能够密度可达的样本集合,即为一个聚类簇。接着继续选择另一个没有类别的核心点去寻找密度可达的样本集合,这样就得到另一个聚类簇。一直运行到所有核心点都有类别为止。
算法的输入:
- 样本集
:::info 距离的度量一般使用欧式距离。 :::
:::warning
某些非核心点可能同时位于两个(或多个)核心点的-邻域内,但这两个核心点并不密度直达(也就是说不属于同一个聚类簇),那么如何界定该非核心点属于哪个聚类簇呢?
一般来说,此时 DBSCAN 采用先来后到的方法,该非核心点会被分配给先进行聚类的那个簇。
因此,DBSCAN 并不是完全稳定的算法。
:::
代码实现
导包:
import numpy as npimport matplotlib.pyplot as pltfrom sklearn.datasets import make_circles, make_moons
生成测试数据:
X1, y1 = make_circles(n_samples=500, factor=0.4, noise=0.1)X2, y2 = make_moons(n_samples=300, noise=0.1)X2 = X2 + np.array([2.2, 1])X = np.vstack((X1, X2))
画图观察数据:
plt.scatter(X[:, 0], X[:, 1])plt.show()

实现 DBSCAN:
### 输入参数eps = 0.15min_pts = 5### 辅助变量、结果变量cluster_no = np.full(X.shape[0], -1) # X 中每个样本所属的簇的编号。先全部初始化为 -1,-1 代表不属于任何簇clusters_count = 0 # 簇的个数core_points_and_neighbors = {} # 所有核心点及其邻域内的点(索引)。key 为核心点(索引),value 是一个列表,包含该核心点的邻域内的所有点(索引)all_core_points = [] # 所有核心点(索引)not_visited_core_points = [] # 所有未访问过的核心点(索引)visited_points = [] # 已访问过的点(索引)### 找出所有核心点及其邻域内的点for i, o in enumerate(X):distance_square = np.sum((o - X) ** 2, axis=1) # 算出点 o 与所有点的距离的平方neighbors = np.where(distance_square <= eps ** 2)[0] # 点 o 的邻域内的所有点(索引),注意:这其中包括点 o 自己if len(neighbors) >= min_pts:core_points_and_neighbors[i] = neighborsall_core_points = list(core_points_and_neighbors.keys()) # 所有核心点(索引)not_visited_core_points = all_core_points.copy() # 所有未访问过的核心点(索引)### 每轮循环随机找一个未访问过的核心点,以该点为中心进行广度优先搜索,### 找出该点能密度可达的所有点,这些点就组成了一个簇。### 当所有核心点都被访问过一次后,跳出循环。while len(not_visited_core_points) > 0:### 创建一个队列,用于广度优先搜索。随机选取一个未访问过的核心点放入队列中core_point = np.random.choice(not_visited_core_points) # 随机选取一个核心点(索引)queue = [core_point] # 初始化队列not_visited_core_points.remove(core_point)### 利用队列进行广度优先搜索。### 每轮循环从队首取出一个点,如果该点是核心点,将其邻域内的所有未访问过的点加入队尾,### 直到队列为空,搜索结束,这些搜索到的所有点组成一个簇。while len(queue) > 0:point = queue.pop(0) # 取出队首的点(索引)### 不管是不是核心点,执行下面两行代码cluster_no[point] = clusters_count # 设置簇号visited_points.append(point) # 添加到已访问列表### 如果该点是核心点if point in all_core_points:if point in not_visited_core_points:not_visited_core_points.remove(point) # 从“未访问过的核心点”列表中删除queue.extend([neighbor for neighbor in core_points_and_neighbors[point] if neighbor not in visited_points and neighbor not in queue]) # 将该点的邻域内所有未访问过的点加入队列,队列中已有的不重复加入clusters_count += 1 # 簇的个数加一
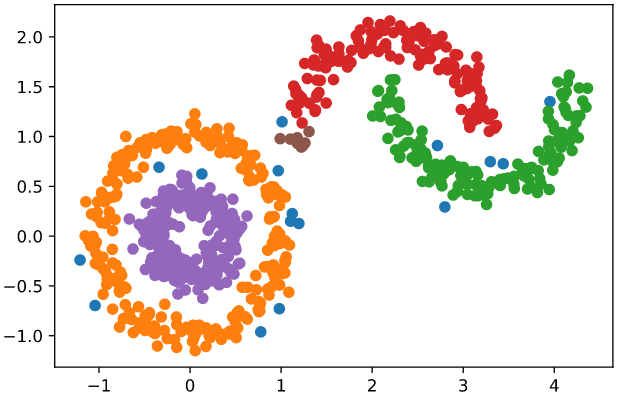
可视化展示聚类结果:
for i in range(-1, clusters_count):points = X[cluster_no == i]plt.scatter(points[:, 0], points[:, 1])plt.show()
 :::info
图中蓝色的点是离群点,不属于任何簇。
:::
:::info
图中蓝色的点是离群点,不属于任何簇。
:::
超参数的选择
TODO
如何评价结果好坏
TODO
算法优缺点
优点:
- 可以对任意形状的稠密数据集进行聚类,相对的,K-Means 之类的聚类算法一般只适用于凸数据集。
- 可以在聚类的同时发现异常点,对数据集中的异常点不敏感。
- 聚类结果基本稳定,相对的,K-Means 之类的聚类算法初始值对聚类结果有很大影响。
缺点:
- 有 2 个超参数,因此调参比 K-Means 更复杂,因为要对 2 个超参数联合调参。
- 如果样本集的密度不均匀、聚类间距差相差很大时,聚类质量较差,这时用 DBSCAN 聚类一般不适合。
- 样本集较大时,花费时间较长。

