对于10X空间转录组数据而言,可以使用Space Ranger对数据进行处理。
软件下载
https://support.10xgenomics.com/spatial-gene-expression/software/downloads/latest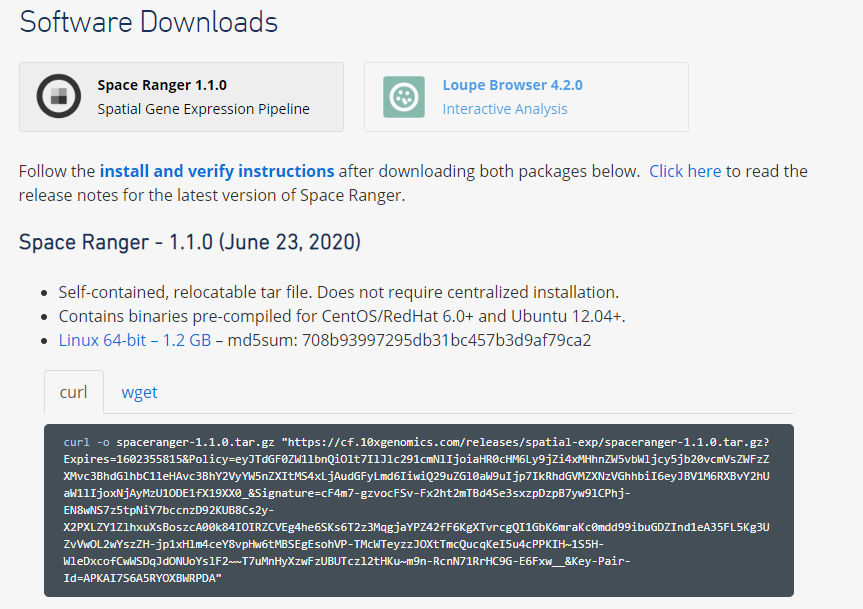
下载,解压后即可使用。
基因组下载
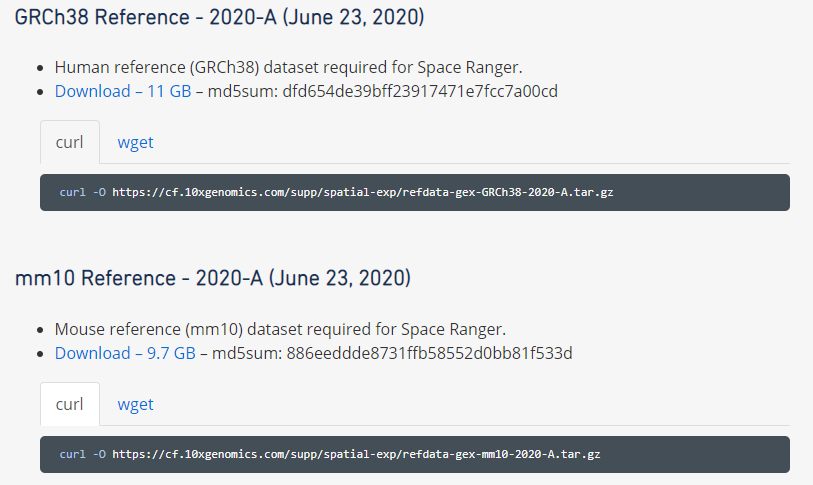
应该也可以使用常规提供的参考基因组(等待测试)
软件安装
解压Space Ranger文件
tar -xzvf spaceranger-1.1.0.tar.gz
加入到环境变量
export PATH=/opt/spaceranger-1.1.0:$PATH
fastq序列文件:
10x建议测序read2设置90个碱基的长度。reads序列长度达到70bp以后,mapping率就不太变化了,有些样本在长度大于90bp以后mapping率反而下降了。
10x给出这样的建议是考虑到建库后插入片段太短的话,太长的序列里面会包含部分polyA甚至UMI序列反而影响比对效果。
因为国内目前基本上是测2x150bp的,如果建库片段过短比对率较低的话建议将read2剪切成90bp来分析效果应该会好一点。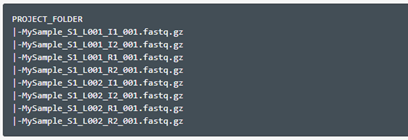
图像文件:
Space Ranger支持两种样式的显微镜图像:用苏木精和曙红(H&E)染色的明场图像,必须是24位彩色TIFF,16位灰度TIFF或JPEG。Space Ranger要求任一方向上的图像至少为2000像素。
图像边框的四个角位置是固定的,比如说三角形在左下角,如果扫描出来的图片四个角的位置变了需要先手动调整过来再使用。
自动对齐:$ cd /home/jdoe/runs$ spaceranger count --id=sample345 \ ###结果输出文件夹名称--transcriptome=/opt/refdata/GRCh38-3.0.0 \ ####基因组目录--fastqs=/home/jdoe/runs/HAWT7ADXX/outs/fastq_path \ ###fastq文件路径--sample=mysample \ ###原始样本名--image=/home/jdoe/runs/images/sample345.tif \ ##图片文件--slide=V19J01-123 \ ##使用的10x芯片型号--area=A1 ##样本所在芯片的区域(四通道芯片位置从上到下分别为A1/B1/C1/D1)--slidefile=V19J01-123.gpr ###当无法联网时,下载对应的gpr文件离线进行分析
手动对齐:
$ cd /home/jdoe/runs$ spaceranger count --id=sample345 \--transcriptome=/opt/refdata/GRCh38-3.0.0 \--fastqs=/home/jdoe/runs/HAWT7ADXX/outs/fastq_path \--sample=mysample \--image=/home/jdoe/runs/images/sample345.tif \--slide=V19J01-123 \--area=A1 \--loupe-alignment=sample345.json ##图片手动对齐生成的json文件,如果是自动对齐不要需要此参数
另外,spaceranger默认使用系统上可用的所有内核来执行管。所以可以使用–localcores选项来限制使用内核的数量。同理,可以使用-localmem来限制使用内存的大小。
结果输出:
- Outputs of spatial pipeline: /opt/sample345/outs/spatial
- Filtered feature-barcode matrices MEX: /opt/sample345/outs/filtered_feature_bc_matrix
- Filtered feature-barcode matrices HDF5:

