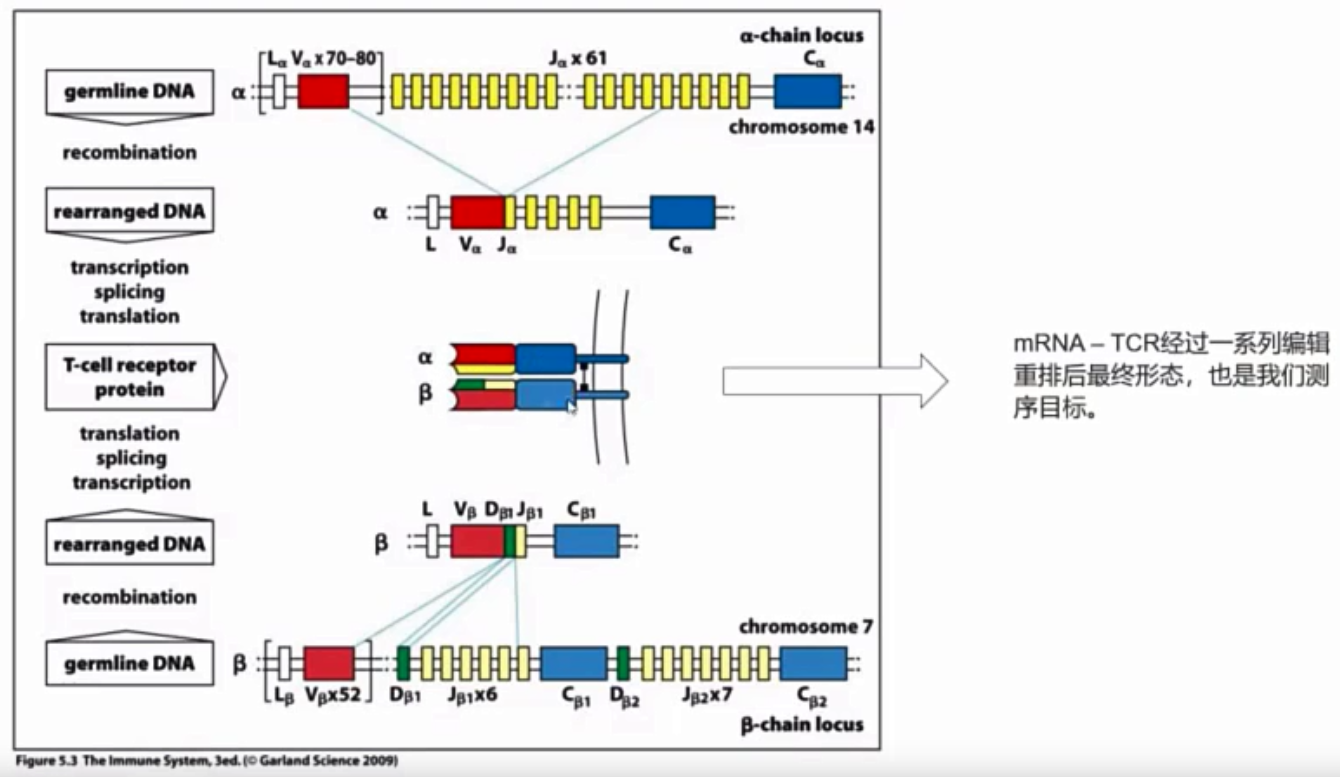
VDJ
参考:https://www.jianshu.com/p/a87f2601831e
VDJ基因是编码生成BCR和TCR的基因。BCR和TCR,是B(T) Cell Receptor的缩写,是一种长在B或T细胞表面的免疫球蛋白,其作用是识别抗原,接收抗原刺激。其中,BCR由重链和轻链组成,TCR由α和β链组成。
每一个BCR,包含两条重链和两条轻链。其中重链由一个可变区(V区)和三个恒定区(C区)组成,轻链则包含一个可变区与一个恒定区。重链的恒定区,决定了BCR的免疫原性,即决定了它属于5类免疫球蛋白(IgA、IgG、IgM、IgD、IgE)中的哪一种。而轻链的恒定区,则决定了搭配的轻链种类(k或入型)。而可变区(V区)则决定了它的特异性,即决定了它能和什么样的抗原结合。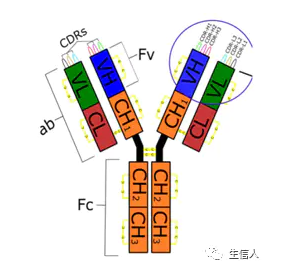
cellranger
使用cellranger进行定量
cellranger vdjcellranger count
cellranger vdj --id=自己给样本的命名\--reference==/pathway/ref 参考基因组所在的路径\--fastqs=/pathway/fastqs 对应fastq所在位置\--sample=sample fastq前面的编号,方便识别--localcores=20 设置计算的核心数--localmen=100 设置计算的内存--denovo 不添加denovo参数的时候,序列会以局部比对的方式,比对到10X官网上下载的参考序列上(就是reference),反之,则是先对reads进行组装,然后再比对。--inner-enrichment-primers 是对扩增引物的指定。适用于除了人和鼠之外,对其他物种进行V(D)J测序时,可能需要自行设定引物序列。
结果:
Outputs:- Run summary HTML: /outs/web_summary.html- Run summary CSV: /outs/metrics_summary.csv- Clonotype info: /outs/clonotypes.csv (需要)- Filtered contig sequences FASTA: /outs/filtered_contig.fasta- Filtered contig sequences FASTQ: /outs/filtered_contig.fastq- Filtered contigs (CSV): /outs/filtered_contig_annotations.csv(需要)- All-contig FASTA: /outs/all_contig.fasta- All-contig FASTA index: /outs/all_contig.fasta.fai- All-contig FASTQ: /outs/all_contig.fastq- Read-contig alignments: /outs/all_contig.bam- Read-contig alignment index: /outs/all_contig.bam.bai- All contig annotations (JSON): /outs/all_contig_annotations.json- All contig annotations (BED): /outs/all_contig_annotations.bed- All contig annotations (CSV): /outs/all_contig_annotations.csv- Barcodes that are declared to be targetted cells: /outs/cell_barcodes.json- Clonotype consensus FASTA: /outs/consensus.fasta- Clonotype consensus FASTA index: /outs/consensus.fasta.fai- Clonotype consensus FASTQ: /outs/consensus.fastq- Contig-consensus alignments: /outs/consensus.bam- Contig-consensus alignment index: /outs/consensus.bam.bai- Clonotype consensus annotations (JSON): /outs/consensus_annotations.json- Clonotype consensus annotations (CSV): /outs/consensus_annotations.csv(需要)- Concatenated reference sequences: /outs/concat_ref.fasta- Concatenated reference index: /outs/concat_ref.fasta.fai- Contig-reference alignments: /outs/concat_ref.bam- Contig-reference alignment index: /outs/concat_ref.bam.bai- Loupe V(D)J Browser file: /outs/vloupe.vloupe

clonotypes.csv
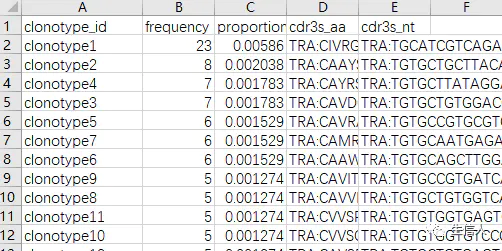
clonotype_id:是这个样本内部依次排下去的序号,不同样本之间不通用!!!
frequency:这个表示这个克隆型在样本中的多少个cell中被检测到,侧面部分反映了这个BCR的丰度
proportion:clonotype表达的细胞数占样本细胞总数的比例
CDR3区域的氨基酸构成。
CDR3区域的核苷酸组合。
CDR3是V(D)J基因编码的核心区域,通常会包含V基因的一部分,然后D基因,还有J基因的一部分,因此是BCR或者TCR上最具有代表性的,最具有辨识度的一段区域
不同的annotation.csv文件。侧重不同层面对数据进行注释和解读。
Contig 注释结果文件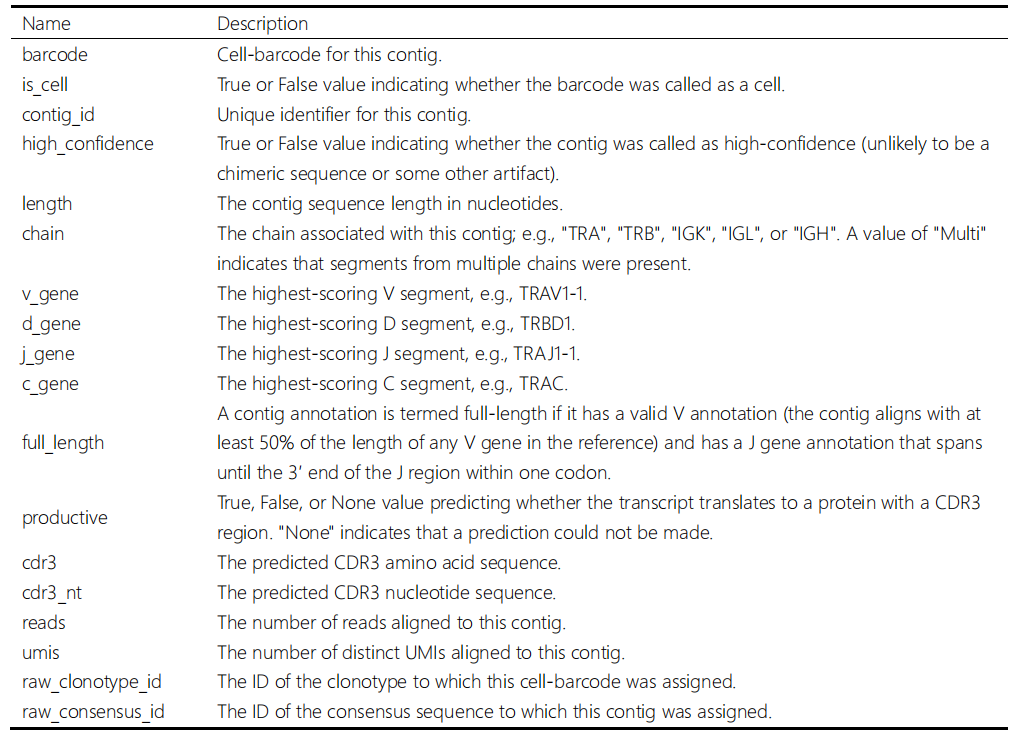
官方说明:https://support.10xgenomics.com/single-cell-vdj/software/pipelines/latest/output/overview
单细胞数据分析
数据读取
数据质控
数据标准化
数据筛选
数据归一化
主成分选择
细胞聚类
细胞降维
marker基因分析及可视化
细胞群的差异基因
TCR/BCR分析
数据读取
数据质控
数据清洗
克隆型统计
不同V(D)J基因统计
不同链CDR3统计
常规分析:
1.CDR3长度统计
immunarch::repExplore(immdata$data,"lens",.col = "nt") %>% vis()scRepertoire::lengthContig(combined, cloneCall="nt", chains = "combined")
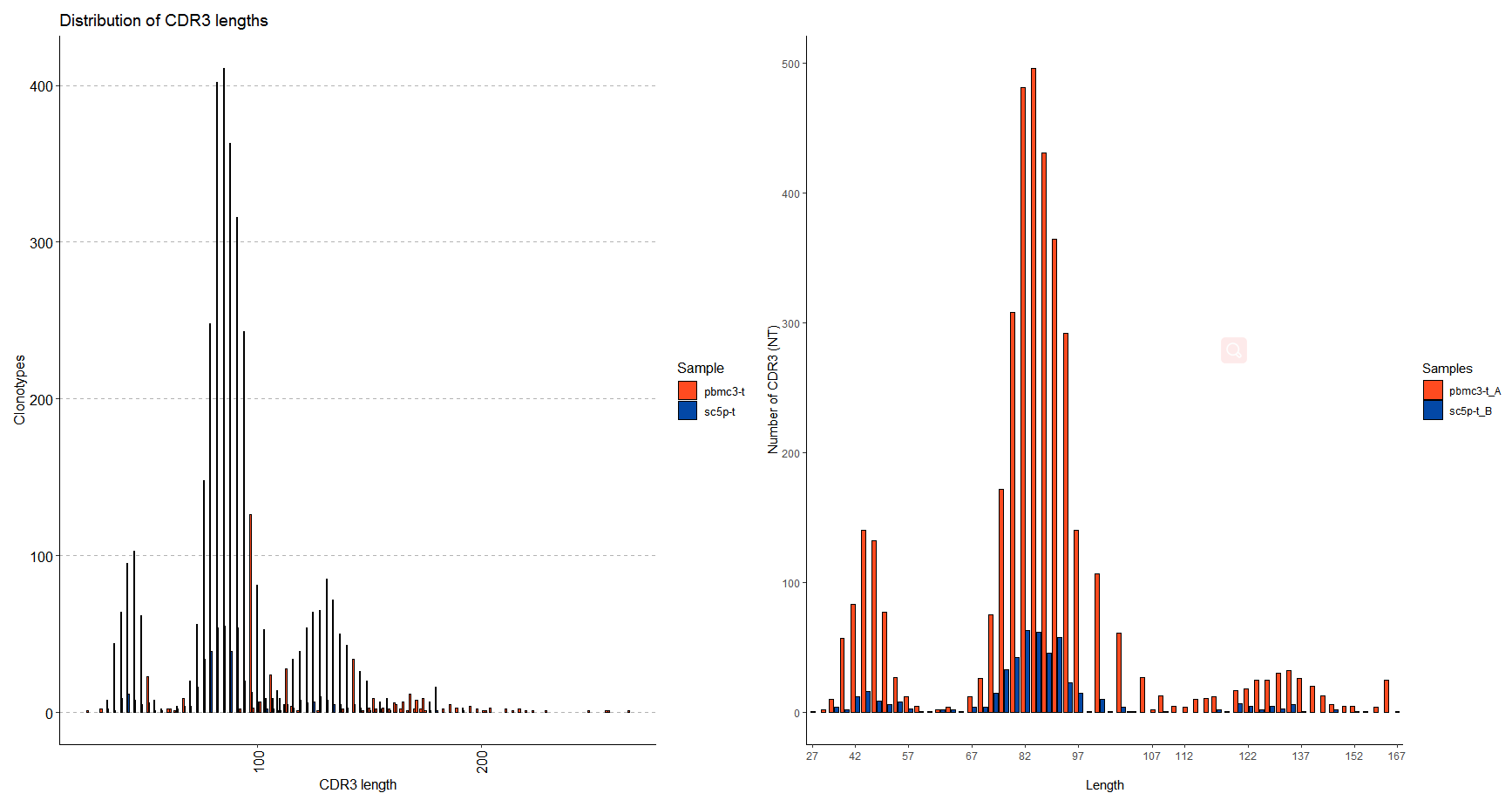
“nt” for nucleotide sequences,
“aa” for amino acid sequences,
“v” for V gene segments,
“j” for J gene segments.
CDR3 gene (gene)
CDR3 nucleotide (nt)
CDR3 amino acid (aa)
CDR3 gene+nucleotide (gene+nt)

