本章概要
本章讲述线性模型(linear model),相关内容包括:
- 线性回归(linear regression)
序关系(order)、均方差(square error)最小化、欧式距离(Euclidean distance)、最小二乘法(least square method)、参数估计(parameter estimation)、多元线性回归(multivariate linear regression)、广义线性回归(generalized linear model)、对数线性回归(log-linear regression);
- 对数几率回归(逻辑回归)(logistic regression)
分类、Sigmoid函数、对数几率(log odds / logit)、极大似然法(maximum likelihood method);
- 线性判别分析(linear discriminant analysis - LDA)
类内散度(within-class scatter)、类间散度(between-class scatter);
- 多分类学习(multi-classifier)
拆解法,一对一(One vs One - OvO)、一对其余(OvR)、多对多(MvM)、纠错输出码(ECOC)、编码矩阵(coding matrix)、二元码、多标记学习(multi-label learning);
- 类别不平衡(class-imbalance)
再缩放(rescaling)、欠采样(undersampling)、过采样(oversampling)、阈值移动(threshold-moving);
习题解答
3.1 线性回归模型偏置项
偏置项b在数值上代表了自变量取0时,因变量的取值;
1.当讨论变量x对结果y的影响,不用考虑b;
2.可以用变量归一化(max-min或z-score)来消除偏置。
3.2 证明对数似然函数是凸函数(参数存在最优解)
直接给出证明结果如下图:
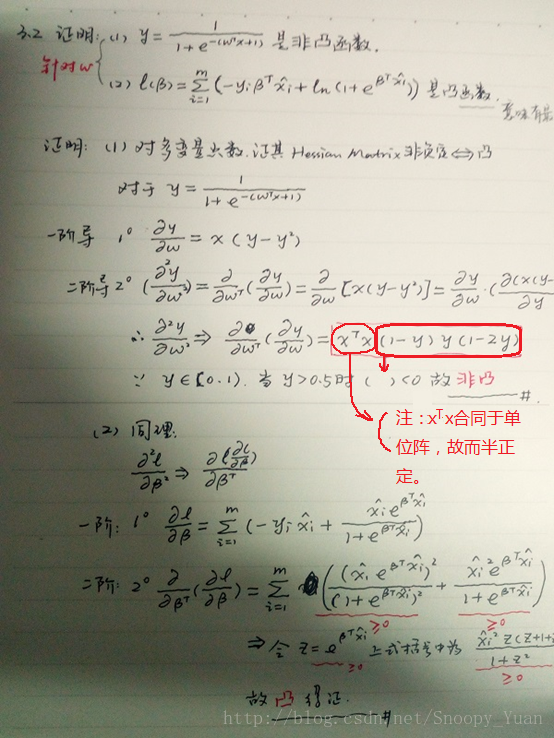
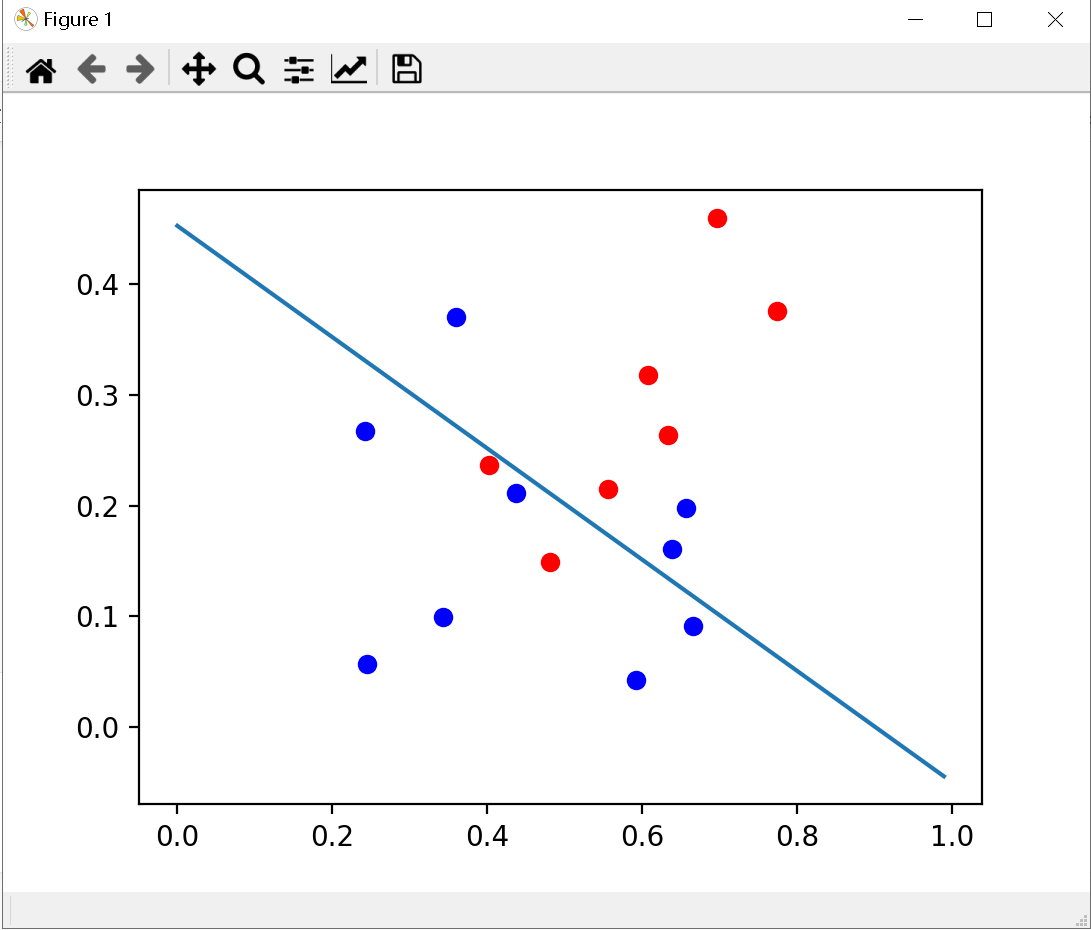
3.3 编程实现对率回归
所使用的数据集如下: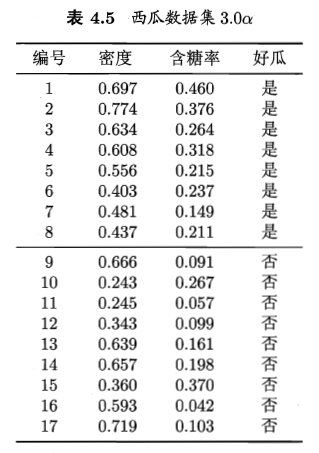
首先,我们要构造跃阶函数,也就是sigmoid函数,书中给的函数是:<br />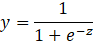<br />由这个跃阶函数来处理二分类问题。
但是,这个跃阶函数的效果不是太好,亲自试验过,发现效果不是一般的差。一般都是用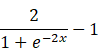
这个公式来计算,也就是上图注释掉的代码。
import numpy as np
import matplotlib.pyplot as plt
def sigmoid(inX):
# return 1.0/(1+np.exp(-inX))
return 2*1.0/(1+np.exp(-2*inX))-1
# 训练
def gradAscend(data,labels):
m = data.shape[0] # 行
n = data.shape[1] # 列
step = 0.001
time = 1000
wights = np.ones((n,1))
for i in range(time):
h = sigmoid(data*wights)
error = labels-h
wights = wights+step*data.T*error
return wights
def createDataSet():
group = np.matrix([[0.697,0.460,1],
[0.774,0.376,1],
[0.634,0.264,1],
[0.608,0.318,1],
[0.556,0.215,1],
[0.403,0.237,1],
[0.481,0.149,1],
[0.437,0.211,1],
[0.666,0.091,1],
[0.243,0.267,1],
[0.245,0.057,1],
[0.343,0.099,1],
[0.639,0.161,1],
[0.657,0.198,1],
[0.360,0.370,1],
[0.593,0.042,1],
[0.719,0.103,1]])
labels = np.matrix([[1],[1],[1],[1],[1],[1],[1],[1],[0],[0],[0],[0],[0],[0],[0],[0],[0]])
return group, labels
group, labels = createDataSet()
w = gradAscend(group,labels)
data = np.array(group)
X1 = []
Y1 = []
X2 = []
Y2 = []
for i in range(7):
X1.append(data[i,0])
Y1.append(data[i,1])
for i in range(7,16):
X2.append(data[i,0])
Y2.append(data[i,1])
plt.scatter(X1, Y1, c='red')
plt.scatter(X2, Y2, c='blue')
x1 = np.arange(0, 1, step=0.01)
plt.plot(x1, (0.5 - x1 * w[0,0] - w[2,0]) / w[1,0])
plt.show()
print(w)
3.4 比较k折交叉验证法与留一法

本题采用UCI中的 Iris Data Set 和 Blood Transfusion Service Center Data Set,基于sklearn完成练习。
关于数据集的介绍:
IRIS数据集简介 - 百度百科;通过花朵的性状数据(花萼大小、花瓣大小…)来推测花卉的类别。变量属性X=4种,类别标签y公有3种,这里我们选取其中两类数据来拟合对率回归(逻辑斯蒂回归)。
Blood Transfusion Service Center Data Set - UCI;通过献血行为(上次献血时间、总献血cc量…)的历史数据,来推测某人是否会在某一时段献血。变量属性X=4种,类别y={0,1}。该数据集相对iris要大一些。
1. 数据导入、可视化、预分析:
iris数据集十分常用,sklearn的数据包已包含该数据集,我们可以直接载入,也可以下载后导入。对于transfusion数据集,我们从UCI官网上下载导入即可。
采用seaborn库可以实现基于matplotlib的非常漂亮的可视化呈现效果,下图是采用seaborn.pairplot()绘制的iris数据集各变量关系组合图,从图中可以看出,类别区分十分明显,分类器应该比较容易实现: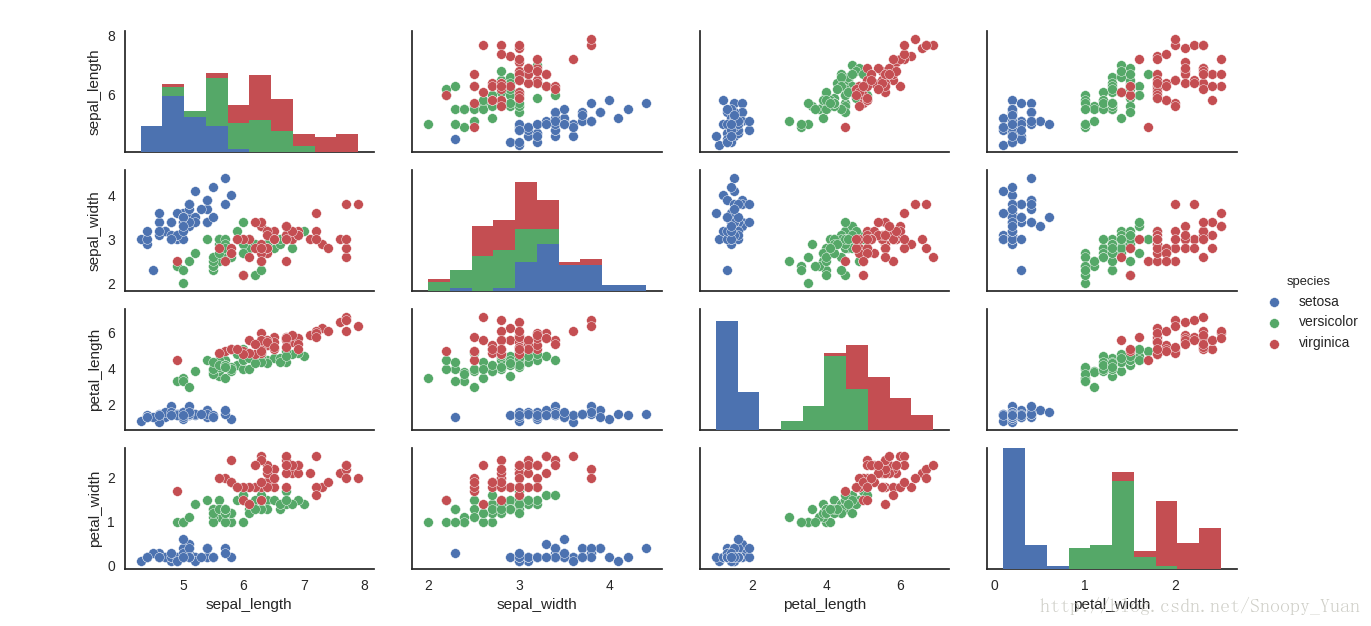
相关样例代码:
import numpy as np
import seaborn as sns
sns.set(style="white", color_codes=True)
iris = sns.load_dataset("iris")
iris.plot(kind="scatter", x="sepal_length", y="sepal_width")
sns.pairplot(iris,hue='species')
sns.plt.show()
2. 基于sklearn进行拟合与交叉验证:
这里我们选择iris中的两类数据对应的样本进行分析。k-折交叉验证可直接根据sklearn.model_selection.cross_val_predict()得到精度、F1值等度量(该函数要求1<k<n-1)。留一法稍微复杂一点,这里采用loop实现。
面向iris数据集的样例代码:
import numpy as np
import seaborn as sns
from sklearn.linear_model import LogisticRegression
from sklearn import metrics
from sklearn.model_selection import cross_val_predict, LeaveOneOut
sns.set(style="white", color_codes=True)
iris = sns.load_dataset("iris")
X = iris.values[50:150, 0:4]
y = iris.values[50:150, 4]
iris.plot(kind="scatter", x="sepal_length", y="sepal_width")
sns.pairplot(iris, hue='species')
sns.plt.show()
# 2-nd logistic regression using sklearn
# log-regression lib model
log_model = LogisticRegression()
m = np.shape(X)[0]
# 10-folds CV
y_pred = cross_val_predict(log_model, X, y, cv=10)
print(metrics.accuracy_score(y, y_pred))
# LOOCV
from sklearn.model_selection import LeaveOneOut
loo = LeaveOneOut()
accuracy = 0
for train, test in loo.split(X):
log_model.fit(X[train], y[train]) # fitting
y_p = log_model.predict(X[test])
if y_p == y[test]: accuracy += 1
print(accuracy / np.shape(X)[0])
得出了精度(预测准确度)结果如下:
0.97
0.96
可以看到,两种方法的模型精度都十分高,这也得益于iris数据集类间散度较大。
面向transfusion数据集的样例代码
from sklearn.linear_model import LogisticRegression
from sklearn import metrics
from sklearn.model_selection import cross_val_predict, LeaveOneOut
import numpy as np
dataset_transfusion = np.loadtxt('transfusion.data', delimiter=",", skiprows=1)
X2 = dataset_transfusion[:, 0:4]
y2 = dataset_transfusion[:, 4]
# log-regression lib model
log_model = LogisticRegression()
m = np.shape(X2)[0]
# 10-folds CV
y2_pred = cross_val_predict(log_model, X2, y2, cv=10)
print(metrics.accuracy_score(y2, y2_pred))
# LOOCV
loo = LeaveOneOut()
accuracy = 0;
for train, test in loo.split(X2):
log_model.fit(X2[train], y2[train]) # fitting
y2_p = log_model.predict(X2[test])
if y2_p == y2[test]: accuracy += 1
print(accuracy / np.shape(X2)[0])
结果:
0.76
0.77
也可以看到,两种交叉验证的结果相近,但是由于此数据集的类分性不如iris明显,所得结果也要差一些。同时由程序运行可以看出,LOOCV的运行时间相对较长,这一点随着数据量的增大而愈发明显。
所以,一般情况下选择K-折交叉验证即可满足精度要求,同时运算量相对小。