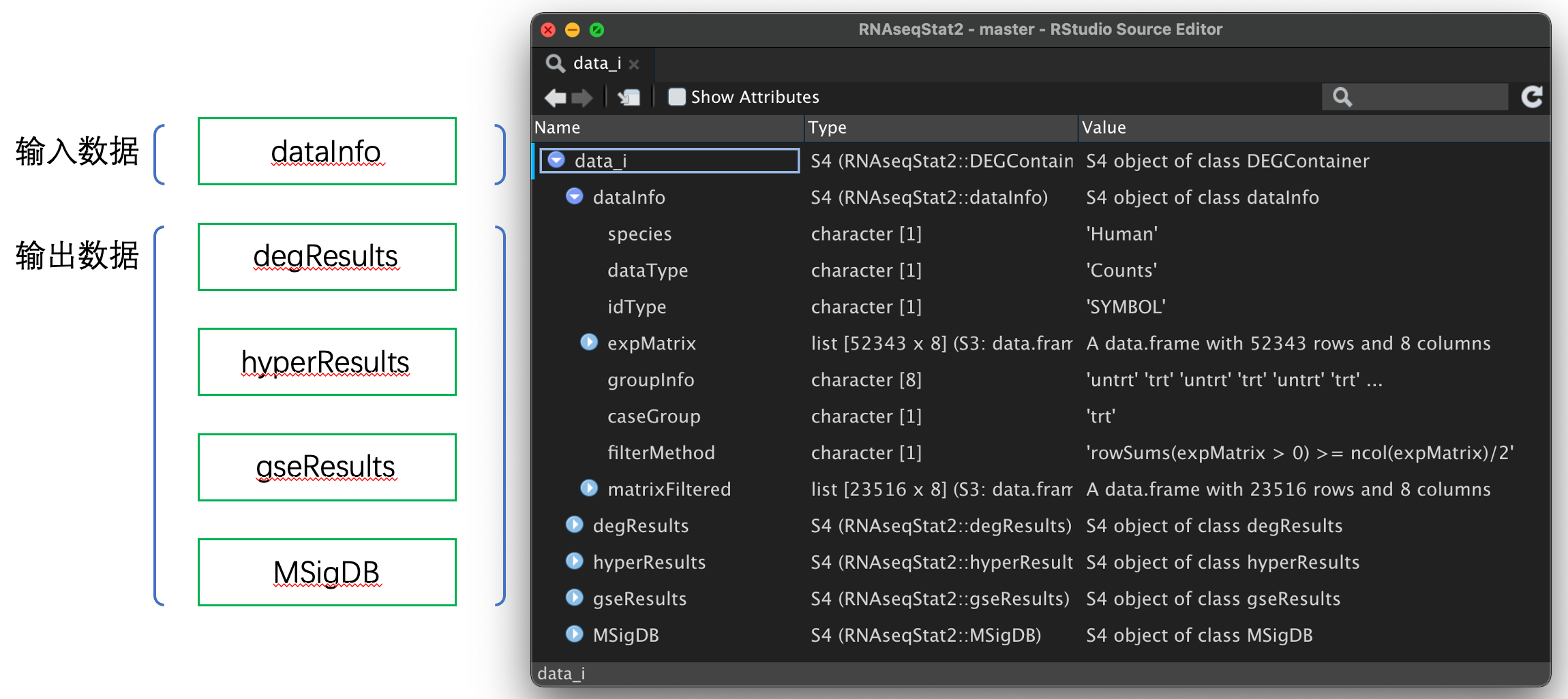
一个 DEGContainer 对象就像乐高积木一样,可以被拆解成分散的模块。
目前对于这样的子模块并没有做很多函数的支持,主要是拆解数据方便查看。
所以不推荐在运算完成之前这样做。
拆分代码
dtinfo <- dataInfo(data_i)degres <- degResults(data_i)hyperres <- hyperResults(data_i)gseres <- gseResults(data_i)msigdb <- MSigDB(data_i)
五大模块
dataInfo
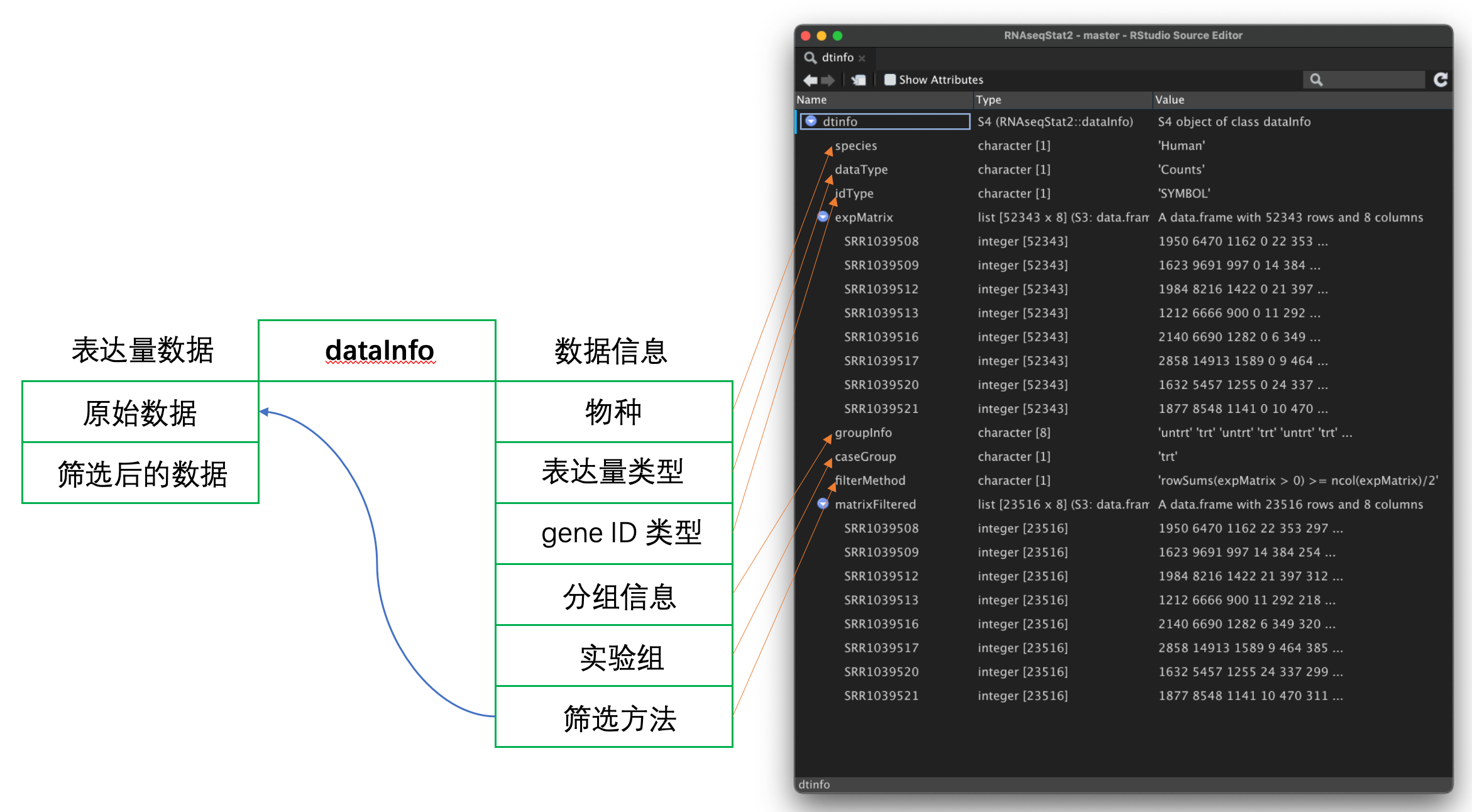
- expMatrix 筛选前的表达量矩阵
- species 物种名 比如 Human Mouse Rat 或者其他
- dataType 数据类型 Counts 还是 Array,如果是 Counts 意味着值为整数类型
- idType ID 类型 请使用 SYMBOL
- groupInfo 分组信息 每个样本对应哪个分组
- caseGroup 实验组 哪个分组是实验组
- filterMethod 筛选方法,可以是一个函数,可以是数字,可以是任何能对data.frame 的基因起到筛选作用的东西
filterMethod 筛选后的表达矩阵,也是之后用来的分析的表达矩阵。
degResults
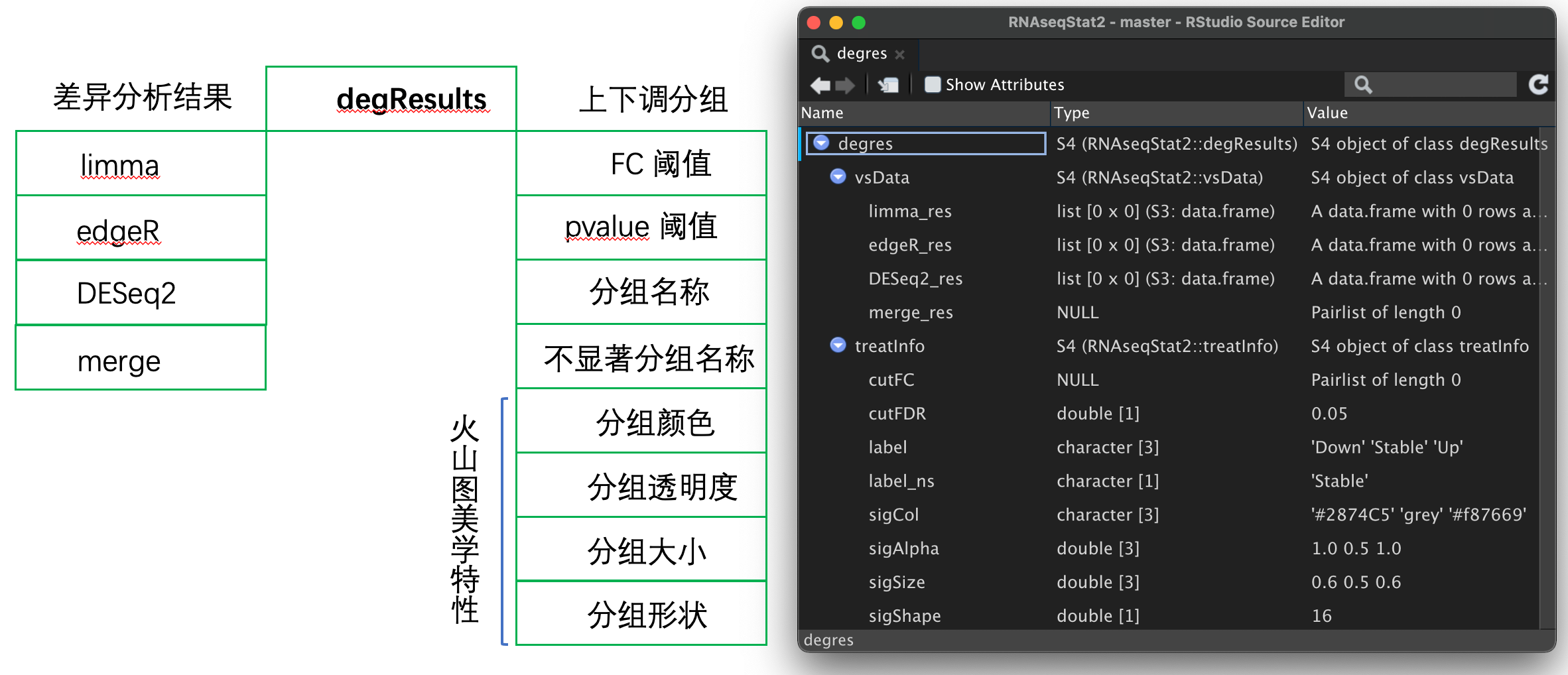
vsData 三种方法差异分析的结果,以及这三种方法的交集
**limma_res**limma 的差异分析结果**edgeR_res**edgeR 的差异分析结果**DESeq2_res**DESeq2_res 的差异分析结果**merge_res**当存在上述三种方法的两种及以上分析结果时,取的交集
treatInfo 处理差异分析结果的参数
**cutFC**为logFC设置的阈值,默认为NULL,计算过程中会自动指定一个合适的阈值,并更新到此处。也可以指定一个合适的值,但通常在差异分析运算之前,无法确定这个值。**cutFDR**为 pvalue 设置的阈值,默认0.05**label**上下调分组和不显著分组的名字,顺序应该是 下调 不显著 上调**label_ns**不显著分组的名字,应是label中的一个**sigCol****sigAlpha****sigSize****sigShape**火山图的散点 颜色 透明度 大小 形状,如果是一个大家都一样,如果是三个分别对应 下调 不显著 上调hyperResults
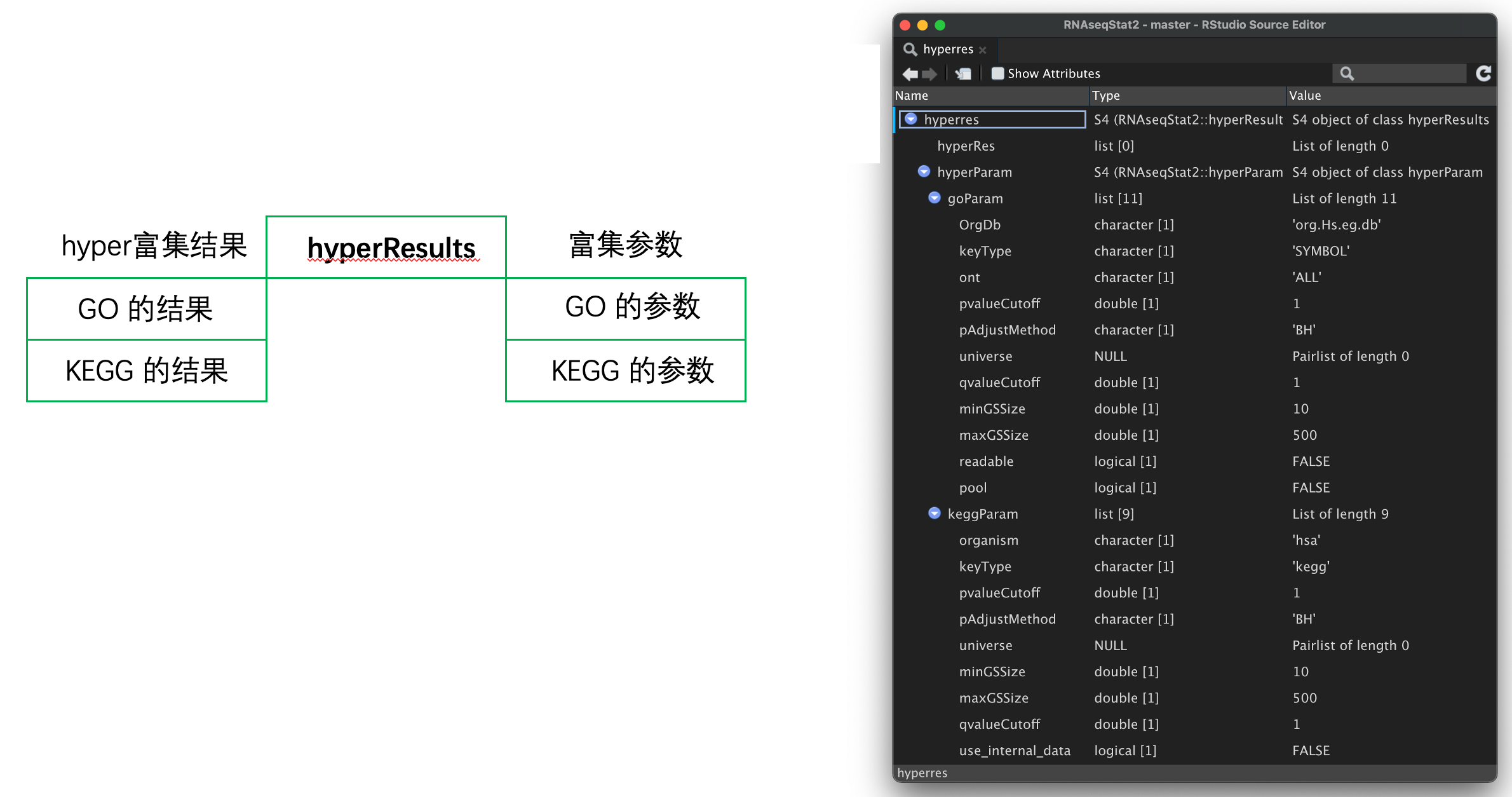
**hyperRes**hyper 分析的结果**hyperParam****goParam**hyper GO 的参数**keggParam**hyper KEGG 的参数
gseResults
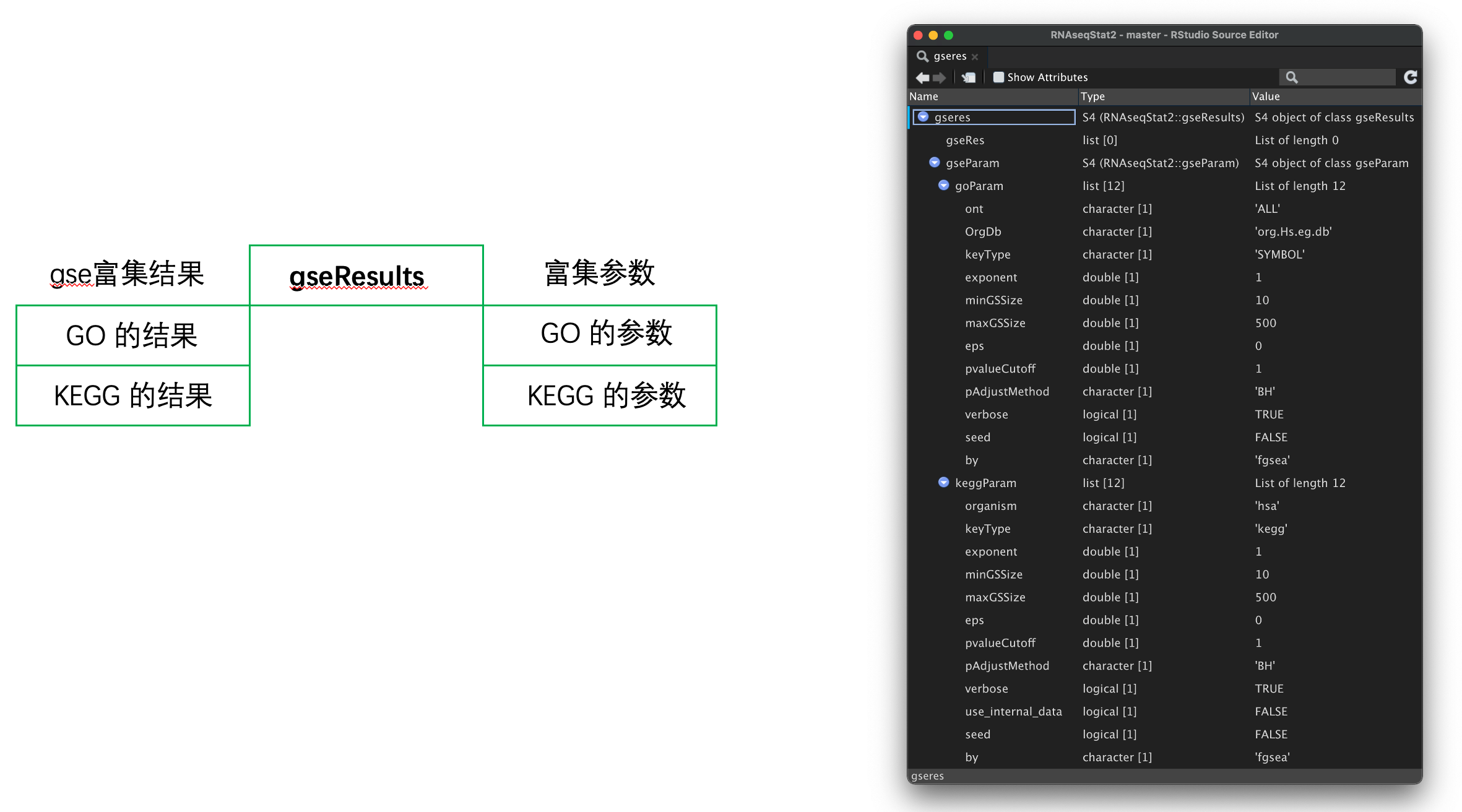
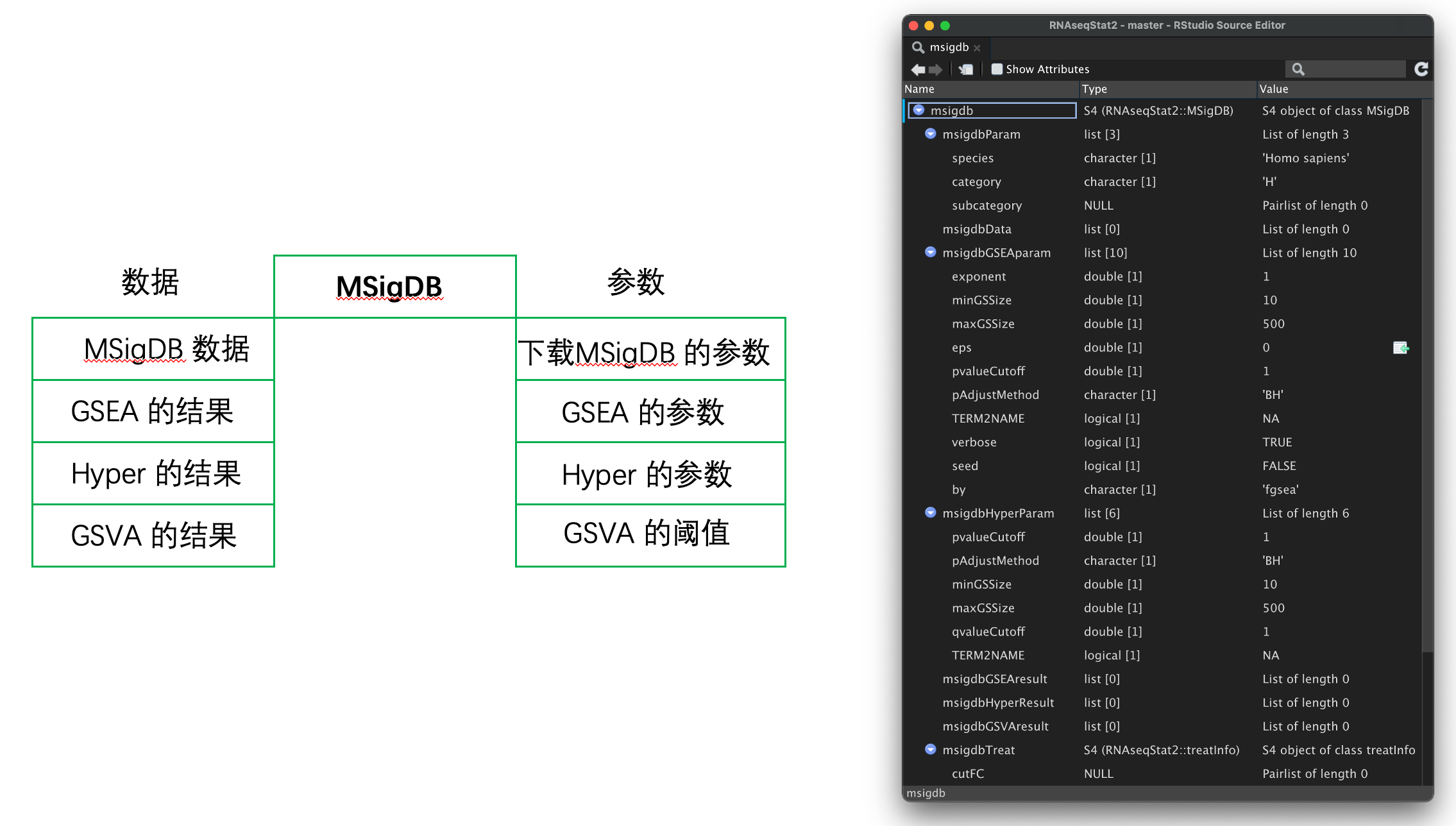
**gseRes**gse 分析的结果**gseParam**gse 分析的参数msigdbParam 配置 MSigDB 数据库的参数
- species 物种
- category MSigDB 分类中的一个,比如H,C1等
- subcategory 更细的分类
msigdbData 根据上面 MSigDB 数据库的参数下载的MSigDB 数据库
富集分析
msigdbGSEAparam 使用MSigDB 数据库进行 GSE 分析的参数
- msigdbGSEAparam 使用MSigDB 数据库进行 Hyper 分析的参数
- msigdbGSEAresult 使用MSigDB 数据库进行 GSE 分析的结果
msigdbHyperResult 使用MSigDB 数据库进行 Hyper 分析的结果
GSVA
msigdbGSVAresult 使用MSigDB 数据库进行 GSVA 分析的结果
- msigdbTreat 筛选 GSVA 分析结果的阈值
**cutFC**为logFC设置的阈值,默认为NULL,计算过程中会自动指定一个合适的阈值,并更新到此处。也可以指定一个合适的值,但通常在差异分析运算之前,无法确定这个值。**cutFDR**为 pvalue 设置的阈值,默认0.05**label**上下调分组和不显著分组的名字,顺序应该是 下调 不显著 上调**label_ns**不显著分组的名字,应是label中的一个**sigCol****sigAlpha****sigSize****sigShape**火山图的散点 颜色 透明度 大小 形状,如果是一个大家都一样,如果是三个分别对应 下调 不显著 上调对象操作
更改内容
以更改pvalue的阈值为例
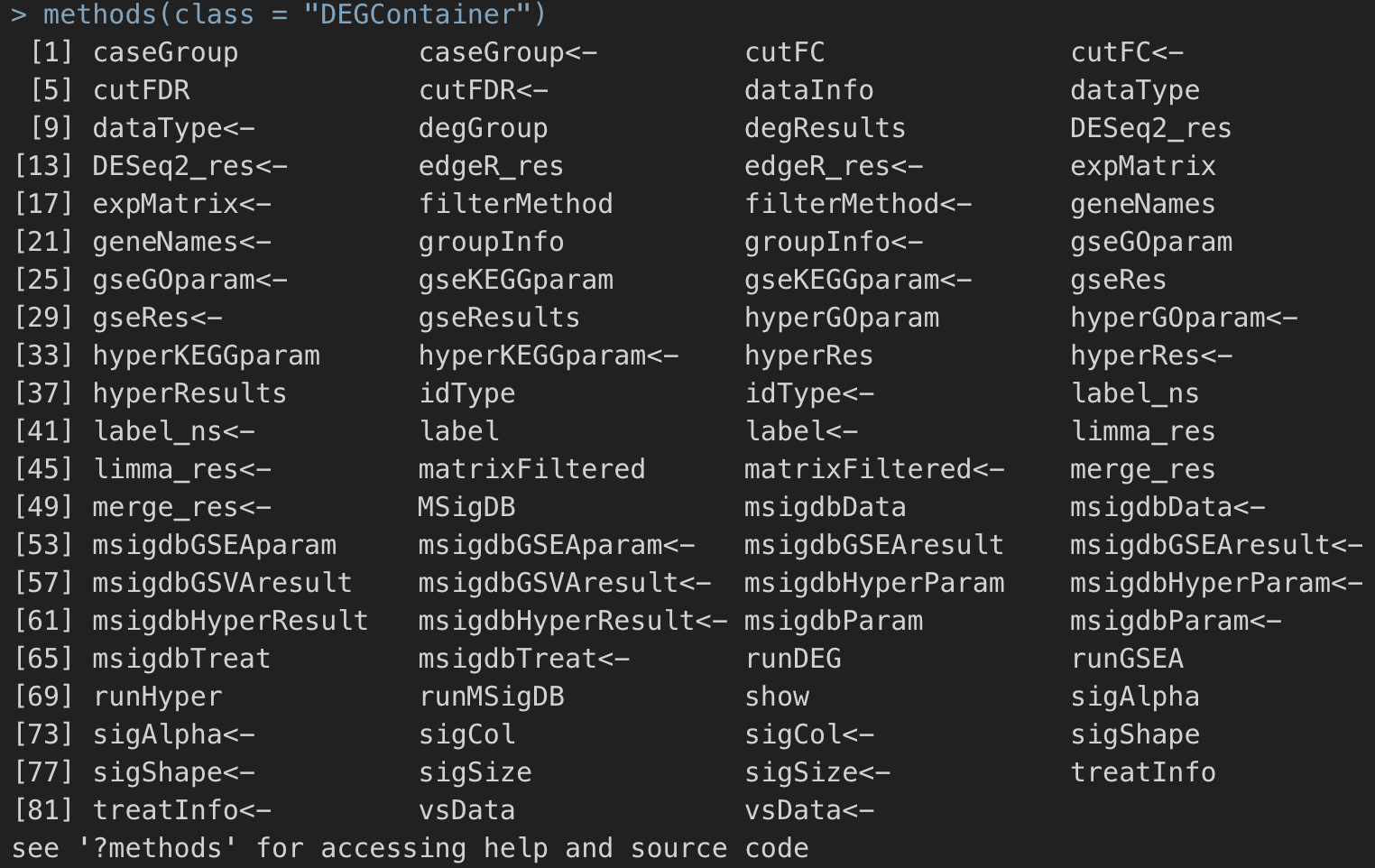
获取所有操作函数
methods(class = "DEGContainer")