任务说明
- 学习主题:作者关联(数据建模任务),对论文作者关系进行建模,统计最常出现的作者关系;
- 学习内容:构建作者关系图,挖掘作者关系
-
数据处理步骤
将作者列表进行处理,并完成统计。具体步骤如下:
将论文第一作者与其他作者(论文非第一作者)构建图;
-
社交网络分析
图是复杂网络研究中的一个重要概念。Graph是用点和线来刻画离散事物集合中的每对事物间以某种方式相联系的数学模型。Graph在现实世界中随处可见,如交通运输图、旅游图、流程图等。利用图可以描述现实生活中的许多事物,如用点可以表示交叉口,点之间的连线表示路径,这样就可以轻而易举的描绘出一个交通运输网络。
图类型
无向图,忽略了两节点间边的方向。
- 指有向图,考虑了边的有向性。
多重无向图,即两个结点之间的边数多于一条,又允许顶点通过同一条边和自己关联。
图统计指标
度:是指和该节点相关联的边的条数,又称关联度。对于有向图,节点的入度 是指进入该节点的边的条数;节点的出度是指从该节点出发的边的条数;
- 迪杰斯特拉路径:.从一个源点到其它各点的最短路径,可使用迪杰斯特拉算法来求最短路径;
- 连通图:在一个无向图 G 中,若从顶点i到顶点j有路径相连,则称i和j是连通的。如果 G 是有向图,那么连接i和j的路径中所有的边都必须同向。如果图中任意两点都是连通的,那么图被称作连通图。如果此图是有向图,则称为强连通图。
对于其他图算法,可以在networkx和igraph两个库中找到。
具体代码以及讲解
首先读取我们想要的数据:
# 导入所需的packageimport seaborn as sns #用于画图from bs4 import BeautifulSoup #用于爬取arxiv的数据import re #用于正则表达式,匹配字符串的模式import requests #用于网络连接,发送网络请求,使用域名获取对应信息import json #读取数据,我们的数据为json格式的import pandas as pd #数据处理,数据分析import matplotlib.pyplot as plt #画图工具
def readArxivFile(path, columns=['id', 'submitter', 'authors', 'title', 'comments', 'journal-ref', 'doi','report-no', 'categories', 'license', 'abstract', 'versions','update_date', 'authors_parsed'], count=None):'''定义读取文件的函数path: 文件路径columns: 需要选择的列count: 读取行数'''data = []with open(path, 'r') as f:for idx, line in enumerate(f):if idx == count:breakd = json.loads(line)d = {col : d[col] for col in columns}data.append(d)data = pd.DataFrame(data)return datadata = readArxivFile('arxiv-metadata-oai-snapshot.json',['id', 'authors_parsed'],200000)
创建作者链接的无向图:
import networkx as nx# 创建无向图G = nx.Graph()# 只用五篇论文进行构建for row in data.iloc[:500].itertuples(): # 将DataFrame迭代为元祖authors = row[2] # 将authors_parsed 取出authors = [' '.join(x[:-1]) for x in authors]# 第一个作者 与 其他作者链接for author in authors[1:]:G.add_edge(authors[0],author) # 添加节点2,3并链接23节点
- iterrows(): 将DataFrame迭代为(insex, Series)对。
- itertuples(): 将DataFrame迭代为元祖。
- iteritems(): 将DataFrame迭代为(列名, Series)对
# 将作者关系图进行绘制:plt.figure(figsize=(30,10))nx.draw(G, with_labels=True)

这里也可以用draw_networkx()。
参考链接
[‘Balázs C.’, ‘Nadolsky P. M.’, ‘Huston J.’, ‘The ATLAS Collaboration ‘, ‘Ryan P.’, ‘Liu Jian’, ‘Gray Benjamin’, ‘Lee Ho Nyung’, ‘Rabe Karin M.’, ‘Cooper Valentino R.’, ‘Johnston Karen’, ‘Kleis Jesper’, ‘Ziambaras Eleni’]try:print(nx.dijkstra_path(G, 'Balázs C.', 'Ziambaras Eleni'))# nx.dijkstra_path(G, source, target, weight='weight')# 也就是从 'Balázs C.'到 'Ziambaras Eleni'的最短路径except:print('No path')
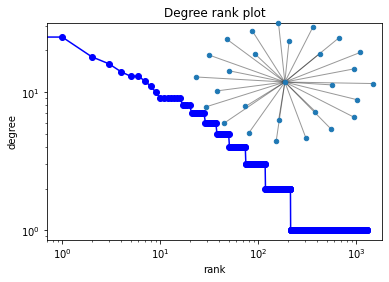
``` 如果我们500片论文构建图,则可以得到更加完整作者关系,并选择最大联通子图进行绘制,折线图为子图节点度值。python degree_sequence = sorted([d for n, d in G.degree()], reverse=True) # 这里的G.degree()可以理解为字典 dmax = max(degree_sequence)
plt.loglog(degree_sequence, “b-“, marker=”o”)
loglog 函数画出logy和logx的曲线,对数坐标可以清楚地看到较小值时的变化。
plt.title(“Degree rank plot”) plt.ylabel(“degree”) plt.xlabel(“rank”)
draw graph in inset
plt.axes([0.45, 0.45, 0.45, 0.45]) Gcc = G.subgraph(sorted(nx.connected_components(G), key=len, reverse=True)[0])
nx.connected_components(G)返回列表形式的G的全连通分量,每个分量一个子列表
sorted里面根据长度len排序,也就是说sorted之后返回的是按子列表长度升序排序的列表,再取出这个最长的列表
Graph.subgraph(nodes)
参数nodes指定子图的节点,返回原图上的子图
pos = nx.spring_layout(Gcc) # 这里是用Fruchterman-Reingold算法排列节点(样子类似多中心放射状)
plt.axis(“off”) # 不画图的axis,可以理解为去掉画布
nx.draw_networkx_nodes(Gcc, pos, node_size=20) # nx.draw_networkx_nodes仅绘制图G的节点
nx.draw_networkx_edges(Gcc, pos, alpha=0.4) # nx.draw_networkx_nodes仅绘制图G的边
plt.show()
```
networkX用法整理
networkx简单教程

