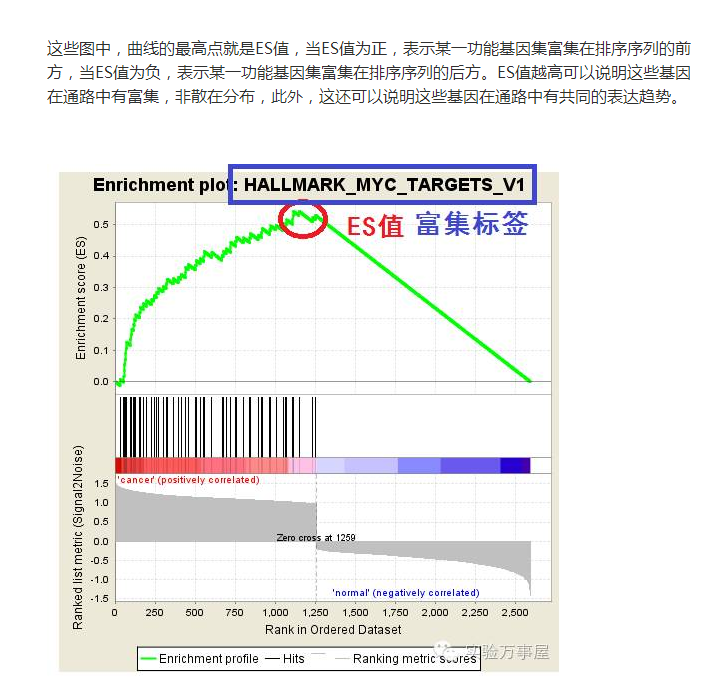
用GSEA来做基因集富集分析
原理:http://www.jianshu.com/p/199b44974480
Posted on 2015年12月30日
how to use GSEA?
这个有点类似于pathway(GO,KEGG等)的富集分析,区别在于gene set(矫正好的基于文献的数据库)的概念更广泛一点,包括了
how to download GSEA ?
软件下载地址:http://software.broadinstitute.org/gsea/downloads.jsp
教程:http://software.broadinstitute.org/gsea/doc/desktop_tutorial.jsp
需要自己安装好java环境!
what’s the input for the GSEA?
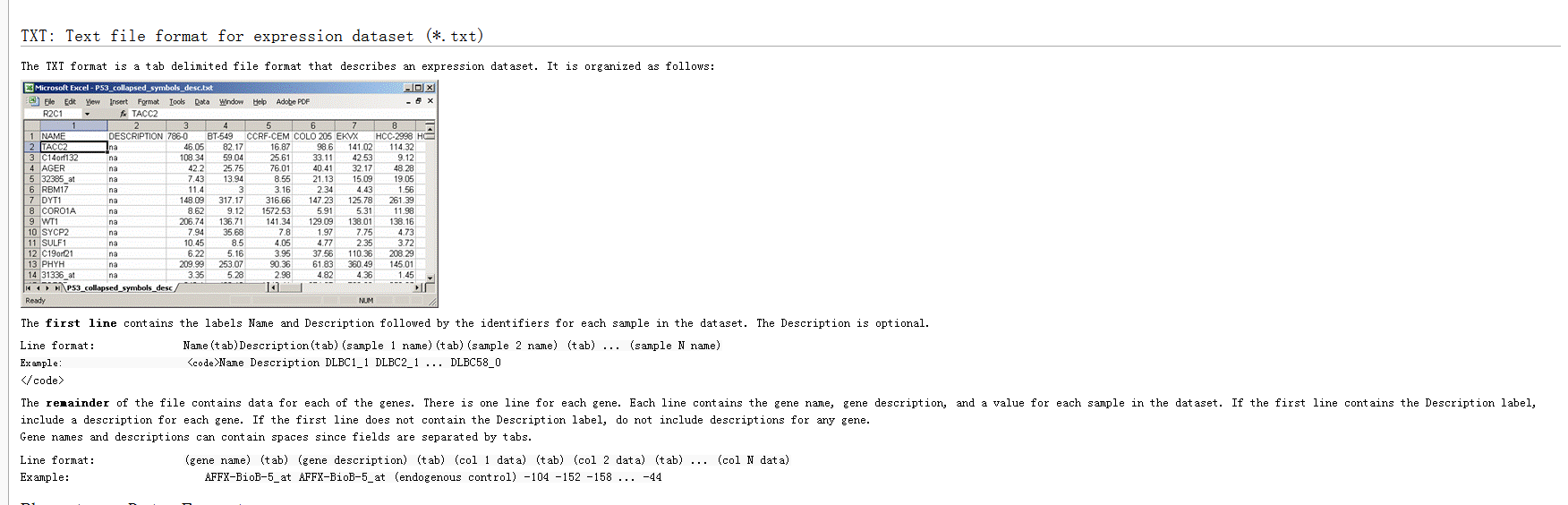
说明书上写的输入数据是:GSEA supported data files are simply tab delimited ASCII text files, which have special file extensions that identify them. For example, expression data usually has the extension .gct, phenotypes .cls, gene sets .gmt, and chip annotations .chip. Click the More on file formats help button to view detailed descriptions of all the data file formats.
并且提供了测试数据:http://software.broadinstitute.org/gsea/datasets.jsp
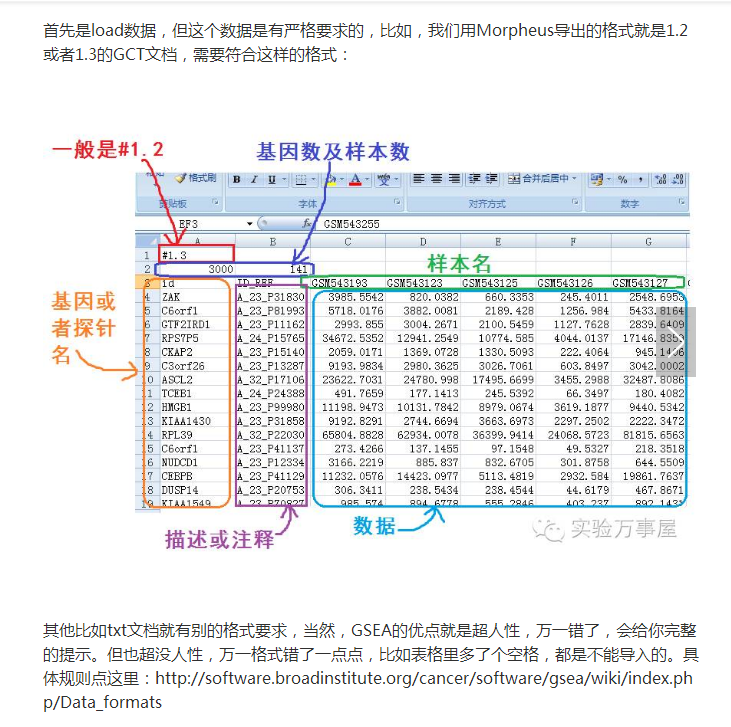
实际上没那么复杂,一个表达矩阵即可!然后做一个分组说明的cls文件即可。
主要是自己看说明书,做出要求的数据格式:http://www.broadinstitute.org/cancer/software/gsea/wiki/index.php/Data_formats
表达矩阵我这里下载GSE1009数据集做测试吧!
http://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=gse1009
ftp://ftp.ncbi.nlm.nih.gov/geo/series/GSE1nnn/GSE1009/matrix/GSE1009_series_matrix.txt.gz
cls的样本说明文件,就随便搞一搞吧,下面这个是例子:
6 2 1
# good bad
good good good bad bad bad
文件如下,六个样本,根据探针来的表达数据,分组前后各三个一组。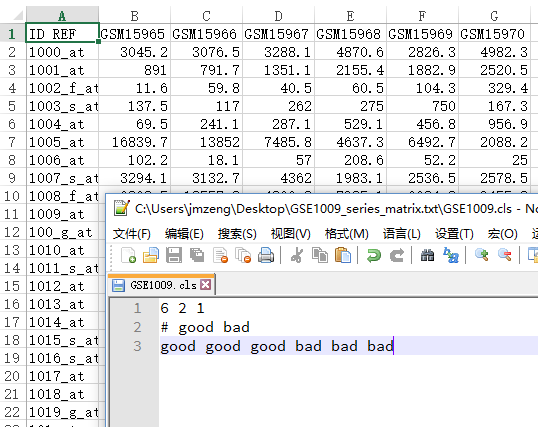
现在开始运行GSEA!
start to run the GSEA !
首先载入数据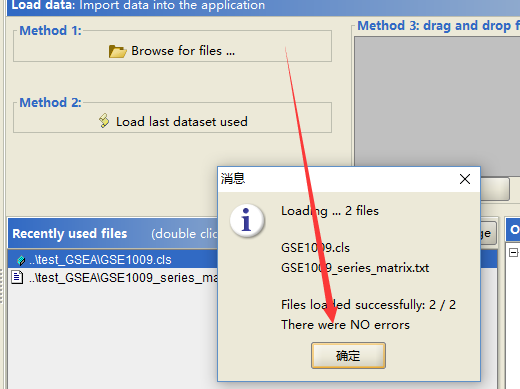
确定无误,就开始运行,运行需要设置一定的参数!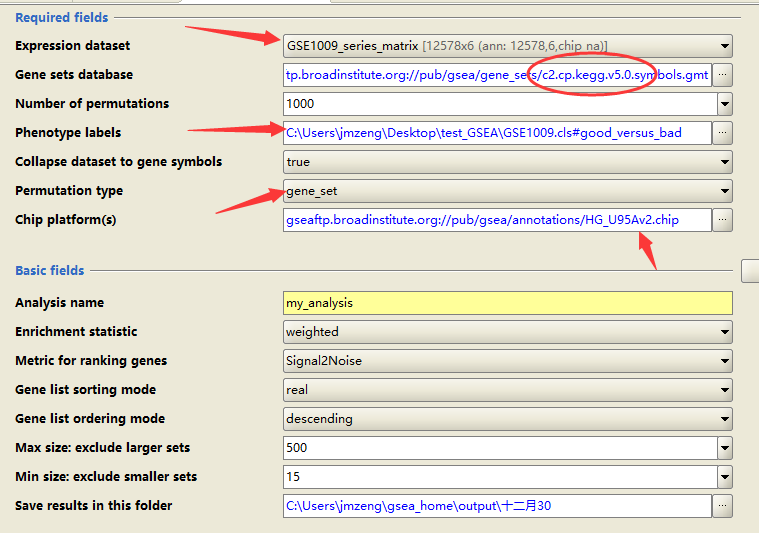
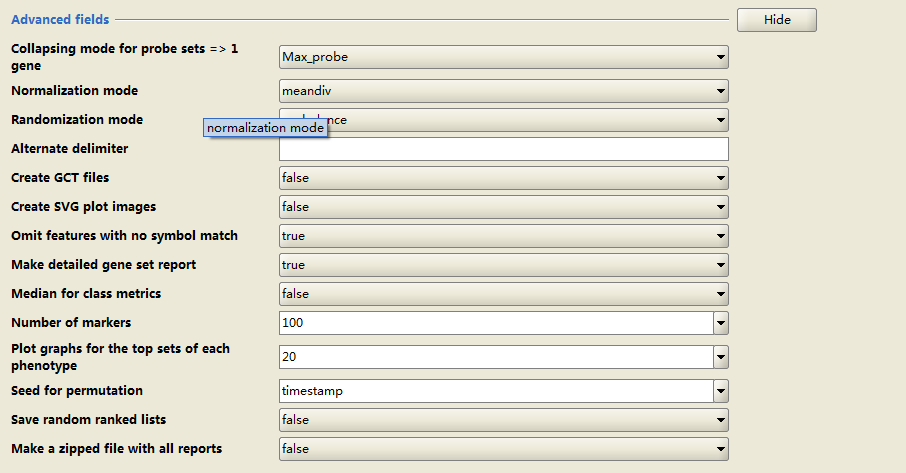
number of permutations : 选择随机的次数
permutation type : 进行基因功能团的随机 或者 进行样本之间的随机
metric for ranking genes : 排序时使用的方法
gene list sorting mode : 排序时使用真实值 或者 绝对值
gene list ordering mode : 上升 或者 下降
max size : 富集到最大数
min size : 富集到最小数(如果是自定义的背景集合,若要得到所有结果则 调为0)
save results : 保存结果的路径