前言:Python强大的脚本功能为生物信息学数据处理提供了巨大的帮助,本文介绍python在序列处理中的一点点小知识,希望能对初学生信者在序列上的处理上有一定帮助。
一、文件打开
Open函数
如打开f盘下的zzj.txt 文档:f=open(r’ f:\zzj.txt’)
这行代码非常简单,也很常用,我们通常将我们要处理的序列信息保存到txt文件里。
二、读取数据
1.读取整个文本: f.read()
2.读取一定的字符数: f.read(n)
如:f=open(r’ f:\zzj.text’)
f.read(7)
3.按行读取且按行输出
f1=open(r'f:\zzj.txt')for line in f1:print linef1.close()
三. 函数split() 常用于**序列格式处理**
提问:为什么是函数split()常用?
将表格信息选中后复制粘贴到txt文件里后,原来表格里的不同列,复制后在txt文档里之间的间隔是制表符而非空格。split函数就是用来对制表符进行识别分割。
Python中有split()和os.path.split()两个函数,具体作用如下:
split():拆分字符串。通过指定分隔符对字符串进行切片,并返回分割后的字符串列表(list)
os.path.split():按照路径将文件名和路径分割
(一)函数说明
1、split()函数
语法:str.split(str=””,num=string.count(str))[n]
参数说明:
str:表示为分隔符,默认为空格,但是不能为空(‘’)。若字符串中没有分隔符,则把整个字符串作为列表的一个元素
num:表示分割次数。如果存在参数num,则仅分隔成 num+1 个子字符串,并且每一个子字符串可以赋给新的变量
[n]:表示选取第n个分片
注意:当使用空格作为分隔符时,对于中间为空的项会自动忽略
2、os.path.split()函数
语法:os.path.split(‘PATH’)
参数说明:
1.PATH指一个文件的全路径作为参数:
2.如果给出的是一个目录和文件名,则输出路径和文件名。
3.如果给出的是一个目录名,则输出路径和为空文件名
(二)分离字符串
string =”www.gziscas.com.cn”
1.以’.’为分隔符
print(string.split(‘.’))
[‘www’,’gziscas’, ‘com’, ‘cn’]
2.分割两次
print(string.split(‘.’,2))
[‘www’,’gziscas’, ‘com.cn’]
3.分割两次,并取序列为1的项
print(string.split(‘.’,2)[1])
gziscas
4.分割两次,并把分割后的三个部分保存到三个文件
u1, u2, u3=string.split(‘.’,2)
print(u1)—— www
print(u2)—— gziscas
print(u3)——com.cn
实例:
1.多序列比对或Blast前进行格式处理,很常用。(分割)
代码1:
f1=open(r'f:\space.txt') #space.txt文件内容是3列多行,3列分别为为序列名称、编号、序列。不同列之间在txt文件里是制表符分隔开来。f2=open(r'f:\33.txt',"w") #用于储存结果的文件。for line in f1:d=f.readline()u1,u2, u3=d.split('\t',2)f2.write( ">"+u1+u2+"\n"+u3)f1.close()f2.close()
代码2:
f=open(r'f:\space.txt')f2=open(r'f:\33.txt',"w")for line in f1:d=f.readline()f2.write(">"+(d.split('\t',2)[0])+(d.split('\t',2)[1])+"\n"+(d.split('\t',2)[2]))f1.close()f2.close()
效果截图如下: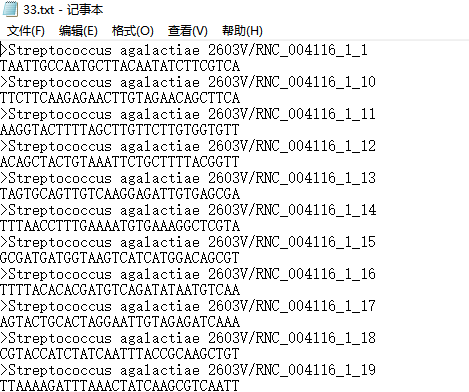
注意点:使用前看清有几个制表符,split里面的参数要做相应调整。
FAQ: 什么是**标准的fasta格式**序列?
标准的fasta格式序列,即对于每条序列而言,序列名称占一行,序列也只占一行,比如上图。
四.python如何去除重复行并分别统计重复的行数?
比如下面的重复,存在内容完全相同的行。怎么把内容重复的行只保留一行,并且统计这样内容重复的行它有多少条?<br /><br /> <br /> **代码如下:**
a=[] #初始化要用到的列表a,用于记录原始行信息b=[] #初始化要用到的列表b,用于记录结果数据。f1=open(r'f:\1.txt') #打开1.txt文件for line in f1:a.append(line) #将1.txt文件每一行作为一个元素,存入列表af1.closefor n in a: #遍历a中每一项(记为n),即1.txt中每一行flag=1for i in range(0,len(b)):if n == b[i][0]: #n与列表b中的每一项对比,如果有相等的:b[i][1]=b[i][1]+1 #那么对应的出现计数加1flag=0breakif flag==1: #如果前面的比对没有一个相等的,即该行是第一次出现:b.append([n,1]) #那么在列表b中添加改行为新的一项f2=open(r'f:\2.txt', "w") #打开2.txt文件,用于输出结果for n in b: #输出格式为:行信息(tab)出现次数 (回车)f2.write(str(n[0][0:-1]) +"\t")f2.write(str(n[1]) +"\n")f2.closeprint "Finished" #分析完成标记
如果不用统计个数,只需要剔除重复的行,则可以使用下面的代码:
lines_seen = set()outfile = open(r"f:\2.txt","w")for line in open(r'f:\1.txt'):if line not in lines_seen:outfile.write(line)lines_seen.add(line)outfile.close()print "Finished" #分析完成标记
五.如何将空字符换成制表符?
你没看错,说的就是空字符(空字符≠空格),空字符就是什么都没有。<br /> 生物信息学中,txt数据处理后有时要导入表格(EXCEL),只有字符与字符之间的制表符才可以EXCEL中分栏(分列)显示。<br /> 本人遇到过一例。将数据从表格导入txt进行批量处理时,写的代码忘记把制表符加上了,导致后来再将txt中的内容导入表格结果不能像以前一样分栏。<br />上面的序列,标黄色(菌株名)的和标蓝色(序列号)的之间为空字符,而序列号与后面的序列之间其实不是空格,而是为制表符。<br /> 将他们复制粘贴到EXCEL表格中,黄色部分会和蓝色部分在同一列,也就是在同一个格子里,但是我们需要在EXCEL表格中把他们放在不同列显示出来,怎么办?<br /> 这种情况我们可以先对txt文件里的信息用下面代码进行补救。
outfile =open(r"f:\out.txt", "w") #用作输出文件for line inopen(r'f:\1.txt'):line= line.replace('NC_','\t'+"NC_")line= line.replace('NZ_','\t'+"NZ_")outfile.write(line)outfile.close()print "Finished" #分析完成标记
上面代码其实是见招拆招。只是用到了replace函数。
这样一来,再将txt里的信息复制粘贴到EXCEL表格中时,黄色部分会和蓝色部分就在不同列了。
警告:所以将数据从表格导入txt后用代码进行某种批量处理时,一定要考虑制表符丢没丢?加不加会不会有影响?这样后期就不会有这么多麻烦事。
六.如何从字符串中提取特定字符串并输出?
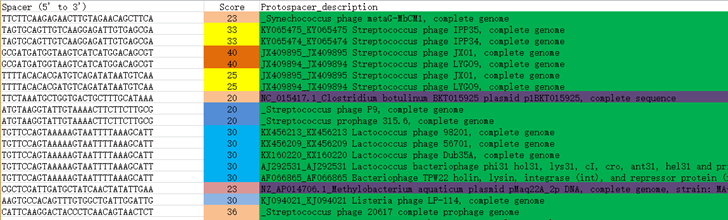
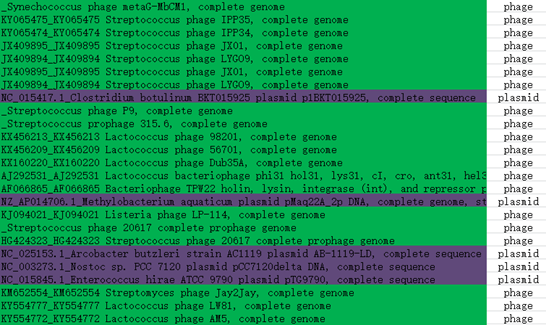
如上图,如果想要直观地知道protospacer_description里的每一行属于噬菌体,质粒,病毒的哪一种,则可以用下面代码。
outfile = open(r"f:\out.txt", "w")a="phage"b="virus"c="plasmid"for line in open(r'f:\1.txt'):if line.find(a) != -1:outfile.write(a+"\n")elif line.find(b) != -1:outfile.write(b+"\n")elif line .find(c) != -1:outfile.write(c+"\n")outfile.close()print "Finished" #分析完成标记
七. 统计两个文本有没有相同内容的行,并将相同的行输出
举个例子,有两个txt文件,1.txt和2.txt,1.txt文件里每一行是一株猪链球菌的CRISPR的DR序列,2.txt文件里每一行是一株肺炎链球菌的CRISPR的DR序列,现在我想知道猪链球菌的CRISPR和肺炎链球菌的CRISPR之间有没有相同的DR,相同的又是哪些DR,下面代码可以实现:
c1=open(r'h:\1.txt')c2=open(r'h:\2.txt')a=[]b=[]c=""k=0for line in c1:a.append(line)c1.closefor line in c2:b.append(line)c2.closefor n in range(0,len(a)):for i in range(0,len(b)):if b[i]==a[n]:c+=b[i]k=k+1breakprint c + str(k) #输出结果并统计个数
八. 分段数据相似度比较,联合输出结果
如在一个txt文本里,有多行序列信息,而每行又由两个部分组成,DNA序列+蛋白质序列(用制表符将两种序列隔开)
现在,我想比较下每行之间的DAN序列相似度和蛋白质序列相似度,并将比对的两个结果在同一行输出,可用以下代码:
import difflib #函数difflib为一种计算序列相似度的方法c1=open(r'e:\jj.txt') #jj.txt是储存序列信息的文件,每一行用制表符分成两部分(DNA序列+蛋白质序列)a=[]b=[]for line in c1:a.append(line)b.append(line)c1.closek=0for n in range(0,len(a)):for i in range(n+1,len(b)):u1, u2=a[n].split('\t',1)u3, u4=b[i].split('\t',1)C=""D=""C=difflib.SequenceMatcher(None,u1,u3).ratio()D=difflib.SequenceMatcher(None,u2,u4).ratio()print str(C)+"\t"+str(D)
示例结果如下: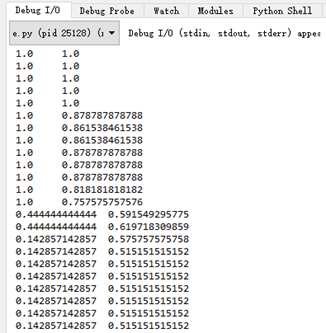
九. 关联信息并顺序输出
现在有两个文件,3.txt里有每一行是GenID号,4.txt每一行是含有GenID号的全名称。
如下图:(注:两个截图均只展示了实际内容的一部分)
图1,文件内容部分截图
图2,文件内容部分截图
现在我想要做一件事,一件什么事呢?我想把4.txt里这样信息全面的名称通过索引关联给3.txt里的GenID,并按照3.txt里的顺序输出。<br /> 代码如下:
f1=open(r"h:\3.txt") #短的名称a=[]b=[]for line1 in f1:line1=line1.strip()a.append(line1)f1.close()f2=open(r"h:\4.txt") #长的名称for line2 in f2:line2=line2.strip(" ")b.append(line2)for i in range(len(a)):flag=1s=a[i].strip()for n in range(len(b)):chang=b[n].strip()if chang[0:8]==s: #示例数据中GenID号为8个字符print b[n].strip()flag=0breakif flag==1:print a[i].strip()f1.close()f2.close()
效果如下:(部分截图)
图3 输出结果部分截图
可以发现输出结果与3.txt里的顺序是一样的。
这个到底有什么用?用处非常大,最简单用途是,比如你某个文件里的数据用的名称是GenID号,现在你想把名称给换换,但是信息比较全的名称在另外一个文件里,而且两个文件的含有相同GenID号的行的顺序不相同,你是要不断通过ctrl+F来一一查找替换还是通过几行代码来解决?
有没有想过,以上面的这样的对应索引来输出结果的代码为基础,进行改变,变成依据索引来进行批量替换?
十.去除名称重复的序列
当使用大数据批量blast的时候,我们可能匹配到一些名称(注意这个词)重复的序列,这个时候序列做去重处理,下面代码可以完成这个工作:
代码如下:
seq=open(r"h:\seq.txt") #输入文件路径,该文件格式要求:每条序列,序列名称占一行,序列也只占一行的fasta格式序列。out=open(r"h:\seq-out.txt","w") #输出文件路径a=[] #建立一个空的列表afor line in seq:line=line.strip() #去掉首尾制表符、空格、换行符a.append(line) #把输入文件里的每一行都存入到列表a中,一行即一个元素n=len(a)s=""b=[] #重建用来去重的列表for i in range((n/2)): #len表示长度,对于列表a而言,len(a)表示列表a里元素的数目s=a[2*i]+"\t"+a[2*i+1]b.append(s)#以下步骤为去重lines_seen = set()c=[]for line in b:u1,u2=line.split("\t")if u1 not in lines_seen:c.append(line)lines_seen.add(u1)for line in c:u1,u2=line.split("\t")out.write(u1+"\n"+u2+"\n")seq.close()out.close()print "Finished" #分析完成标记
FAQ:如果两条序列名称是不同的,但是序列是相同的(即重复的),这种情况下怎么剔除重复序列?
答:要达到这种目的,方法请参照本文第四部分。
十一.多行序列转成一行
我们下载的fasta格式的序列,每条毒(菌株)株序列可能会占多行。通过将序列文件导入诸如Mega等程序,可以将输出为序列名称占一行且序列只占一行的fasta格式的序列。
但是,当序列条数非常多且很长时,比如20000条,如果电脑运行内存较小(比如4G),Mega读取会很缓慢甚至程序卡死。
下面提供代码,将占多行的每条序列变成:一条序列只占一行。
fr=open(r'h:\seq.txt', 'r') #输入文件fw=open(r'h:\out.txt', 'w') #输出文件s=""for line in fr:if line.startswith('>'): #判断字符串是否以‘>开始’line="\n"+lineelse:line=line.replace('\n', '')s=s+linefw.write(s)fr.close()print "Finished" #分析完成标记
十二.输出特定位点为某氨基酸的序列
将氨基酸序列进行多序列比对对齐后,如果想筛选对齐后在第i位点为某氨基(比如M氨基酸)的序列,可以通过下面代码完成。
seq=open(r"h:\seq.fas") #输入文件路径,该文件格式要求为序列名称占一行,序列只占一行的fasta格式序列。out=open(r"h:\seq-out.txt","w") #输出文件路径a=[] #建立一个空的列表afor line in seq:line=line.strip()a.append(line) #把输入文件里的每一行都存入到列表a中,一行即一个元素s=""for n in range(len(a)): #len表示长度,对于列表a而言,len(a)表示列表a里元素的数目s=a[n] #把每一个元素(相当于输入文件里的每一行)的值赋给sif n % 2 == 1: #判断是否是第偶数行if s[i-1:i]=="M": #判断对齐后的第i个位点,具体i值依据实际情况来定out.write(a[n-1]+"\n"+s+"\n")seq.close()out.close()
定义子函数法:
#第一步,定义子函数panbieweidiandef panbieweidian (seqline,outline,i,aa):seq=open(seqline)out=open(outline,"w")a=[]for line in seq:line=line.strip()a.append(line)s=""for n in range(len(a)):s=a[n]if n % 2 == 1:if s[i-1:i]==aa:out.write(a[n-1]+"\n"+s+"\n")seq.close()out.close()#第二步,调用子函数panbieweidianpanbieweidian (r"h:\seq.fas",r"h:\seq-out.fasta",10,"M") #与定义的子函数里的参数一一对应即可#上面为判断第10位是不是氨基酸M,以后只需在调用函数这里修改相应位点和氨基酸类型即可,而第一步的子函数部分不需要改动。#注意,seq.fas该输入文件格式要求为序列名称占一行,序列只占一行。#采用定义子函数的方法写代码后,对相关位点和氨基酸的判别进行修改比较方便,代码重复利用率高。
十三.利用查询功能关联信息
前面在第“九”部分提到了利用索引关联信息,很多小伙伴反映一个问题:这种方法仅仅适用于索引信息规规矩矩地出现在被关联的文本**每行的前几个字符**,而且**字符数还是固定的**。<br />那么,对于**字符数不固定**,而且**位置也不固定**的索引,怎么通过它给两个文本关联信息呢?<br />比如下面的:<br /><br />**(图1,地区经纬度信息表,部分)**
(图2,名称表,部分)
我想把给图2的名称表添加上来自图1的地区经纬度信息,采用下面代码find()函数可以完成任务:
mingchen=open(r"h:\mingchen.txt") #名称列表文件jingwei=open(r"h:\jingwei.txt") #经纬度文件out=open(r"h:\xingxi-out.txt","w") #输出结果文件a=[]for line in jingwei:line=line.strip()a.append(line)for line in mingchen:flage=1line=line.strip()for n in range(len(a)):u1,u2=a[n].split("\t",1)if line.find(u1)!=-1:flage=0s=u2if flage==0:out.write(line+"\t"+s+"\n")elif flage==1:out.write(line+"\n")mingchen.close()jingwei.close()out.close()#经纬度表里的地区名字和名称表里的要相同。
将结果复制到Excle表里,效果如下:
也可以采用定义子函数的方法,如下:
def jingweichaxun (inputmingchen,inputjingwei,outline):mingchen=open(inputmingchen)jingwei=open(inputjingwei)out=open(outline,"w")a=[]for line in jingwei:line=line.strip()a.append(line)for line in mingchen:flage=1line=line.strip()for n in range(len(a)):u1,u2=a[n].split("\t",1)if line.find(u1)!=-1:flage=0s=u2if flage==0:out.write(line+"\t"+s+"\n")elif flage==1:out.write(line+"\n")mingchen.close()jingwei.close()out.close()jingweichaxun (r"h:\mingchen.txt",r"h:\jingwei.txt",r"h:\xingxi-out.txt")#很明显,这个例子定义子函数并没有什么优势。
十四.依据起始位点表截取基因片段
欲截取核苷酸序列的某一片段,手上只有每条序列需要截取的起始位点表,怎么用python脚本解决?
准备材料:
文件1:待截取的基因序列,要求为标准的fasta格式,对于每条序列:序列名称占一行,序列也只占一行。
文件2:需要截取的片段起始位点表(示例如下图),先在EXCLE里做好这个表,再将信息复制到txt文档里。
注意:文件1里的序列名称和文件2里的要一致。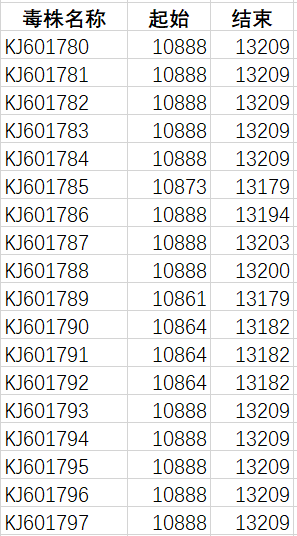
脚本:
import stringf1=open(r"h:\seq.fas") #上述文件1f2=open(r"h:\biaozhun.txt") #上述文件2out=open(r"h:\jiequ-out.fasta","w") #输出文件,事先不需要建立#将f1内容存入列表a,如下:a=[]for line in f1:line=line.strip() #去掉首尾制表符、空格、换行符a.append(line) #把输入文件里的每一行都存入到列表a中,一个元素表示原文件一行#重新改变序列样式,为“序列名称+制表符+序列”占一行,存入列表b,如下:n=len(a)s=""b=[]for i in range(int(n/2)):s=a[2*i]+"\t"+a[2*i+1]b.append(s)#将f2内容存入列表c,如下:c=[]for line in f2:line=line.strip()c.append(line)#依据f2里的长度限定,截取序列长度,如下:for n in range(len(b)):u1,u2=b[n].split("\t",1)u1=u1.strip()u1=u1.replace(">","")u2=u2.strip()for m in range(len(c)):mingchen,qishi,jieshu=c[m].split("\t",2)mingchen=mingchen.strip()x1 = string.atoi(qishi.strip()) #将字符转化为整数x2 = string.atoi(jieshu.strip()) ##将字符转化为整数jiequ=""if u1==mingchen:jiequ=u2[x1 - 1 : x2] #jiequ[x1 - 1 : x2]表示输出字符串jiequ的第x1个字符到第x2个字符(包括第x1和第x2)out.write(">"+u1+"\n"+jiequ+"\n")f1.close()f2.close()out.close()
用途:以冠状病毒为例,欲获取冠状病毒RdRp(RNA依赖的RNA聚合酶基因)的核苷酸序列,由于NCBI上只有氨基酸序列。
但是,RdRp基因位于冠状病毒ORF1ab内,故先将冠状病毒的ORF1ab核苷酸序列在Pfam在线工具上进行预测,得到RdRp基因的起始位点,再依据该起始位点对冠状病毒ORF1ab核苷酸序列进行截取,如上代码,即可得到RdRp的核苷酸序列。
如文中有错误之处,欢迎大家及时指正,一起交流学习。