测序/覆盖深度计算
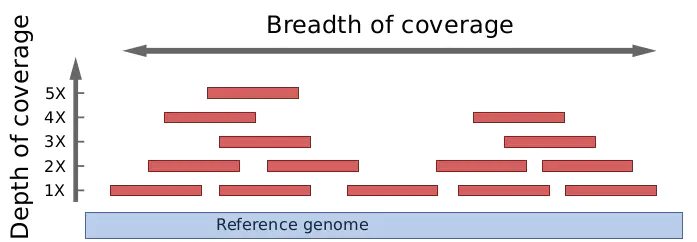
图:测序深度(sequencing depth)或者覆盖深度(depth of coverage);链接:https://www.jianshu.com/p/c43893c938d0
- 测序深度的含义
基因组被测序片段(短读 short reads)“覆盖”的强度有多大?每一碱基的覆盖率是基因组碱基被测序的平均次数。 基因组的覆盖深度是通过与基因组匹配的所有短读的碱基数目除以该基因组的长度来计算的。 它通常表示为1X、2X、3X、…(1、2或3倍覆盖)。此处通常被称为测序深度(sequencing depth)或者覆盖深度(depth of coverage)。
- Depth 计算公式
原始测序数据的测序深度:Depth = (# sequenced bases) / (# bases of reference)
比对上的数据的测序深度:Depth= (# bases of all mapped reads) / (# bases of reference).
计算指定区域的测序深度
描述:read depth per BED region with BAM file
命令:samtools bedcov
研究目的:输入按坐标排序过的bam比对结果文件和bed区间信息文件,统计bed文件中,目标区间内比对结果的深度信息。
输入文件有两个,一个是进过坐标排序的 bam 格式的测序数据比对结果文件,另一个 是 bed 区间信息文件。BED 文件示例:chr1 2000 4000chr2 13500 15000chr3 23000 25000输出文件:chr1 2000 4000 287685 143.84chr2 13500 15000 219340 146.23chr3 23000 25000 294815 147.41新增了两列,意思分别为:追加的第四列为,bed区间内比对上的碱基总数。追加的第五列为,bed区间内碱基的平均深度,计算方式为:第四列的值除以bed区间长度。命令:samtools bedcov -hsamtools bedcov genome.bed sorted.bam | awk '{G+=$2; total+=$3} END {print total"\t"G"\t"total/G}'
参考:https://www.jianshu.com/p/29e28e4f5a9e
测序深度计算的知识点一:bedtools coverage 和 samtools bedcov 的区别
head test.bed1 3073253 3074322 ENSMUSG00000102693.1 1070 + 4933401J01Rik TEC 21 3102016 3102125 ENSMUSG00000064842.1 110 + Gm26206 snRNA 31 3205901 3671498 ENSMUSG00000051951.5 465598 - Xkr4 protein_coding 21 3252757 3253236 ENSMUSG00000102851.1 480 + Gm18956 processed_pseudogene 11 3365731 3368549 ENSMUSG00000103377.1 2819 - Gm37180 TEC 21 3375556 3377788 ENSMUSG00000104017.1 2233 - Gm37363 TEC 21 3464977 3467285 ENSMUSG00000103025.1 2309 - Gm37686 TEC 21 3466587 3513553 ENSMUSG00000089699.1 46967 + Gm1992 antisense 21 3512451 3514507 ENSMUSG00000103201.1 2057 - Gm37329 TEC 21 3531795 3532720 ENSMUSG00000103147.1 926 + Gm7341 processed_pseudogene 1# 命令一:samtools bedcovsamtools bedcov test.bed T1N1_26_R1_bismark_bt2_pe.sorted.bam1 3073253 3074322 ENSMUSG00000102693.1 1070 + 4933401J01Rik TEC 2 01 3102016 3102125 ENSMUSG00000064842.1 110 + Gm26206 snRNA 3 01 3205901 3671498 ENSMUSG00000051951.5 465598 - Xkr4 protein_coding 2 793521 3252757 3253236 ENSMUSG00000102851.1 480 + Gm18956 processed_pseudogene 1 01 3365731 3368549 ENSMUSG00000103377.1 2819 - Gm37180 TEC 2 01 3375556 3377788 ENSMUSG00000104017.1 2233 - Gm37363 TEC 2 01 3464977 3467285 ENSMUSG00000103025.1 2309 - Gm37686 TEC 2 01 3466587 3513553 ENSMUSG00000089699.1 46967 + Gm1992 antisense 2 01 3512451 3514507 ENSMUSG00000103201.1 2057 - Gm37329 TEC 2 01 3531795 3532720 ENSMUSG00000103147.1 926 + Gm7341 processed_pseudogene 1 17# 命令二:bedtools coveragebedtools coverage -a test.bed -b T1N1_26_R1_bismark_bt2_pe.sorted.bamDefault Output:After each entry in A, reports:1) The number of features in B that overlapped the A interval.2) The number of bases in A that had non-zero coverage.3) The length of the entry in A.4) The fraction of bases in A that had non-zero coverage.1 3073253 3074322 ENSMUSG00000102693.1 1070 + 4933401J01Rik TEC 2 0 0 1069 0.00000001 3102016 3102125 ENSMUSG00000064842.1 110 + Gm26206 snRNA 3 0 0 109 0.00000001 3205901 3671498 ENSMUSG00000051951.5 465598 - Xkr4 protein_coding 2 886 1830 465597 0.00393041 3252757 3253236 ENSMUSG00000102851.1 480 + Gm18956 processed_pseudogene 1 0 0479 0.00000001 3365731 3368549 ENSMUSG00000103377.1 2819 - Gm37180 TEC 2 0 0 2818 0.00000001 3375556 3377788 ENSMUSG00000104017.1 2233 - Gm37363 TEC 2 0 0 2232 0.00000001 3464977 3467285 ENSMUSG00000103025.1 2309 - Gm37686 TEC 2 0 0 2308 0.00000001 3466587 3513553 ENSMUSG00000089699.1 46967 + Gm1992 antisense 2 0 0 46966 0.00000001 3512451 3514507 ENSMUSG00000103201.1 2057 - Gm37329 TEC 2 0 0 2056 0.00000001 3531795 3532720 ENSMUSG00000103147.1 926 + Gm7341 processed_pseudogene 1 1 17 925 0.0183784
覆盖度/覆盖范围

图:覆盖度(genome covarage)或者覆盖范围(breadth of coverage)
- 覆盖度/覆盖范围的含义
短读“覆盖”了基因组的多少区域?是否有些区域没有任何的短读覆盖?覆盖范围是参考基因组被一定深度覆盖的碱基的百分比。例如,一个基因组的90%区域在1X深度覆盖,另外仍然有70%的区域被覆盖了5X深度。此处通常称为覆盖度(genome covarage)或者覆盖范围(breadth of coverage)。
- Coverage 计算公式
Coverage = (# area covered by mapped reads) / (# area of reference).
延伸阅读
- samtools的说明文档
- [SAMtools] 常用指令总结,博客园上一篇写得很详细的samtools教程。

