1.数据下载
(1)下载aspera并配置:
wget -c https://download.asperasoft.com/download/sw/connect/3.8.1/ibm-aspera-connect-3.8.1.161274-linux-g2.12-64.tar.gztar zxvf ibm-aspera-connect-3.8.1.161274-linux-g2.12-64.tar.gzbash ibm-aspera-connect-3.8.1.161274-linux-g2.12-64.sh#手动安装需要自行添加环境变量echo 'export PATH=~/.aspera/connect/bin:$PATH' >> ~/.bashrcacsp --help
https://www.jianshu.com/p/112412b8883c
(2)示例数据下载
https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE52778
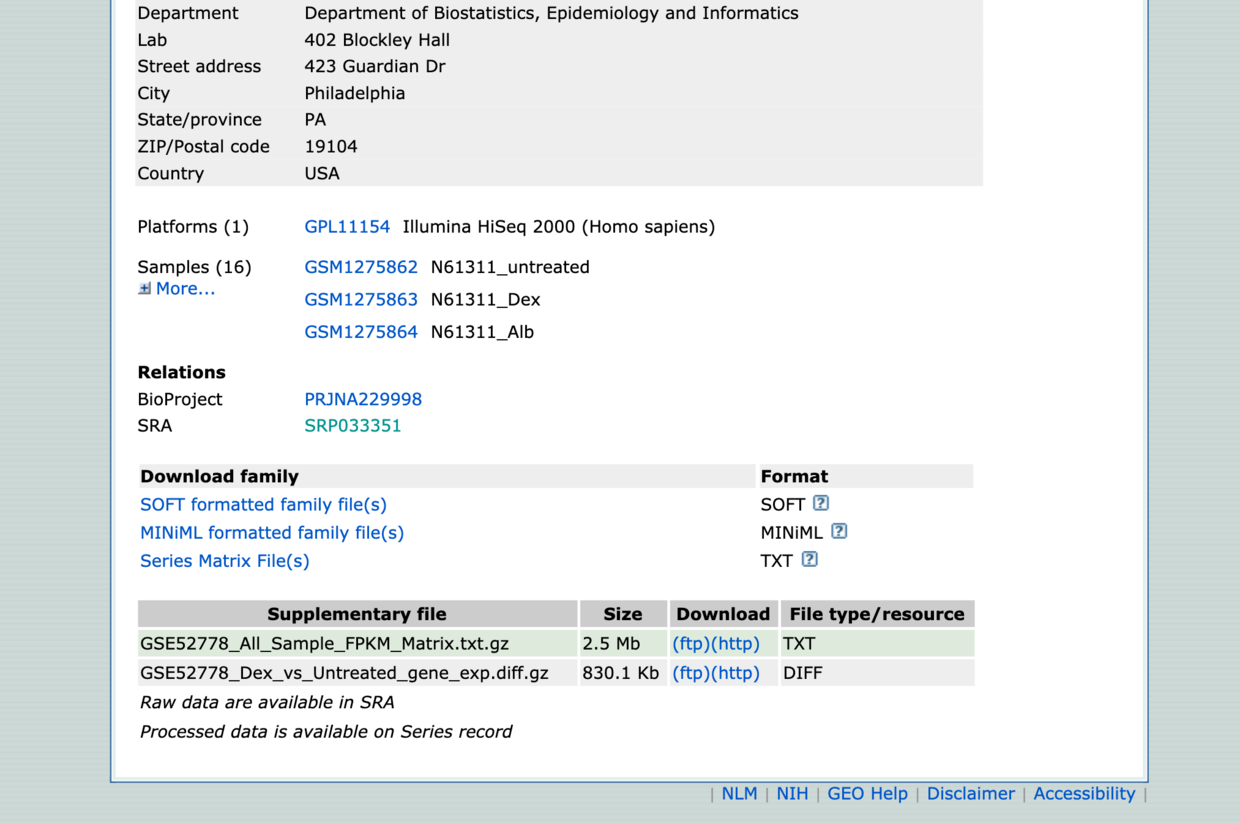
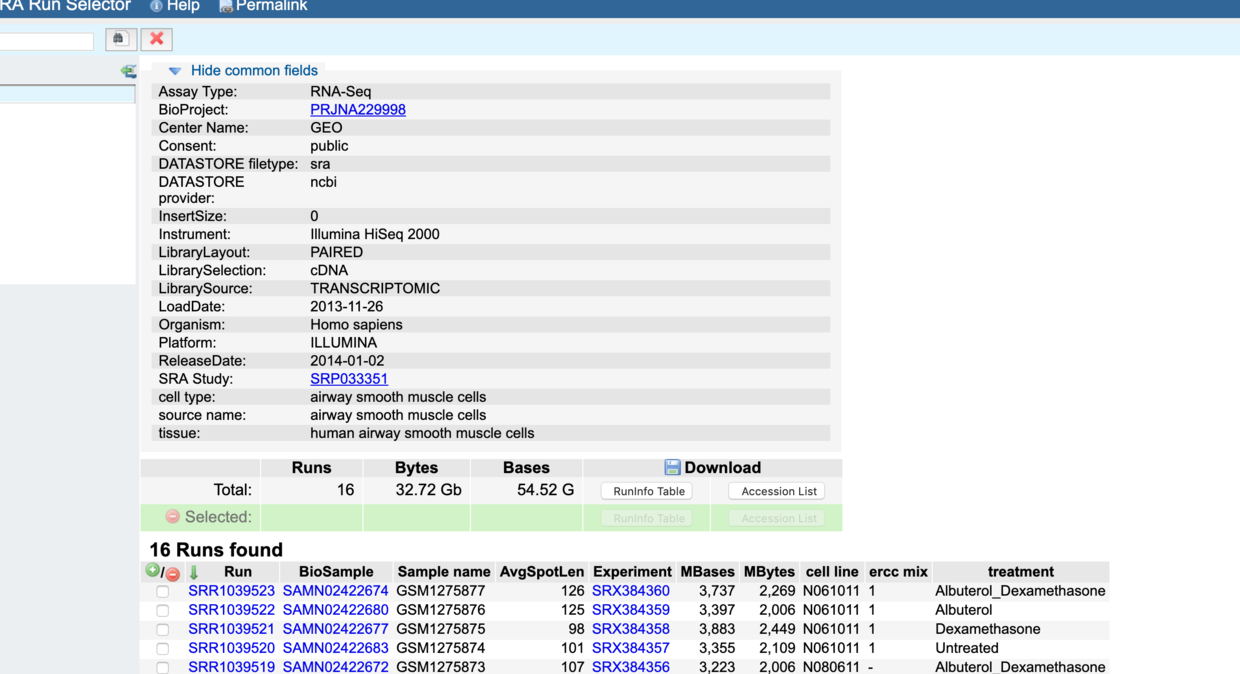
这是它的原始数据列表,文件内容如下:
SRR1039509SRR1039508SRR1039511SRR1039510SRR1039512SRR1039513SRR1039515SRR1039514SRR1039516SRR1039517SRR1039519SRR1039518SRR1039520SRR1039521SRR1039523SRR1039522
可以将该文件下载下来,通过ftp上传到服务器。
cd ~使用重定向将文件存到服务器cat >list.txt#粘贴上面的Assession list内容,换行,按ctrl+c
⚠️注意:必须换行后再ctrl+c,否则最后一行会作废
这里面就是要下载的文件的id,要对每个文件做的事情是:
下载,并将它转换为fastq格式。
先拿一个数据试一下是否可以:
prefetch SRR1039510 -O ~fastq-dump --gzip --split-3 -O ~ SRR1039510.sra
有这么多需要逐一操作太过复杂,所以使用while循环下载sra并转换为fastq。
cat SRR_Acc_List.txt | while read id; do (prefetch ${id} -O ~);donecat SRR_Acc_List.txt | while read id; do (fastq-dump --gzip --split-3 -O ~/ ${id}.sra);done
2.fastqc
示例数据在 /teach/project/1.rna/3.rawfq 25000reads/
fastqc -t 2 -o ~/ /teach/project/1.rna/3.raw_fq_ 25000reads/*.rawfq.gzmultiqc ./*zip
输出到了主目录下。

