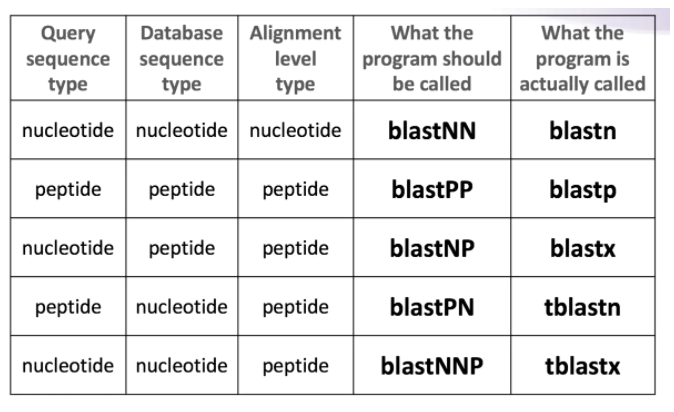
blast软件包括:blastn, blastp, blastx, tblastn, tblastx等。
blast在线使用网址:https://blast.ncbi.nlm.nih.gov/Blast.cgi?PROGRAM=blastn&PAGE_TYPE=BlastSearch&LINK_LOC=blasthome
结果解释:https://ftp.ncbi.nlm.nih.gov/pub/factsheets/HowTo_NewBLAST.pdf
配置blast软件
- 从ftp://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/LATEST/上下载然后安装
- 使用conda安装
conda install -c bioconda blast
blast工具介绍
| Program | Task Name | Description | | —- | —- | —- | | blastp | blastp | protein-protein BLAST
在蛋白质数据库(protein database)中查询蛋白序列(protein query) | | | blastp-short | 适用于短的蛋白序列查询(小于30) | | blastn | blastn | Traditional BLASTN requiring an exact match of 11 | | | blastn-short | 适用于短核酸序列搜索(小于50 bases) | | | megablast | Traditional megablast used to find very similar (e.g., intraspecies or closely related species) sequences
寻找相似序列 | | | dc-megablast | Discontiguous megablast used to find more distant (e.g., interspecies) sequences
寻找远端序列 | | blastx | blastx | search protein databases using a translated nucleotide query | | tblastx | tblastx | Search translated nucleotide databases using a translated nucleotide query | | tblastn | tblastn | search translated nucleotide databases using a protein query |
makeblastdb 构建自己的参考数据库
如果没有参考数据库,可以使用 makeblastdb 自己构建。
-in:输入fa文件
-input_type:指定输入的文件类型
-dbtype:数据库类型;nucl为核酸,prot为蛋白
-title:数据库起个标题,不能用于后面blastn 时 -db
-parse_seqids:推荐加上
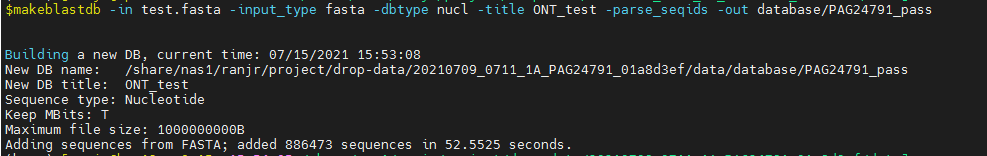
-out :数据库名,如下命令,我的数据库在当前路径下的database下,数据库名关键字是PAG24791_pass
最后构建的完整的数据库包括:.nhr,.nin,.nog,.nsd,.nsi,.nsq 六个文件。
$ makeblastdb -in test.fasta -input_type fasta -dbtype nucl -title ONT_test -parse_seqids -out database/PAG24791_pass$ ll database/total 749064-rw-r--r-- 1 ranjr bmk 255391008 Jul 15 15:54 PAG24791_pass.nhr-rw-r--r-- 1 ranjr bmk 10637752 Jul 15 15:54 PAG24791_pass.nin-rw-r--r-- 1 ranjr bmk 3545924 Jul 15 15:54 PAG24791_pass.nog-rw-r--r-- 1 ranjr bmk 81333296 Jul 15 15:54 PAG24791_pass.nsd-rw-r--r-- 1 ranjr bmk 1492526 Jul 15 15:54 PAG24791_pass.nsi-rw-r--r-- 1 ranjr bmk 220876989 Jul 15 15:54 PAG24791_pass.nsq
blastn 比对

参数介绍
-query:输入文件(例如 query.fa)
-db:数据库路径(本地完整路径,或者NCBI路径)
-query_loc : 指定检索的位置
-out:输出的文件名
-outfmt:输出格式指定,默认是0。
-evalue 科学计数法,比如说1e3,定义期望值阈值,表示随机匹配的可能性。blast会过滤掉期望值大于这个数的比对结果(即这个值越小比对结果就越好)。E值表明在随机的情况下,其它序列与目标序列相似度要大于这条显示的序列的可能性。 与S值有关,S值表示两序列的同源性,分值越高表明它们之间相似的程度越大
E值总结: 1.E值适合于有一定长度,而且复杂度不能太低的序列。2. 当E值小于10-5时,表明两序列有较高的同源性,而不是因为计算错误。3. 当E值小于10-6时,表时两序列的同源性非常高,几乎没有必要再做确认。
-a: 运行BLAST程序所使用的处理器的数目,缺省值1
短序列比对
在运行查询短序列时可以采用 blastn 加上 -task blastn-short -word_size 7 -evalue 1,可以对短序列进行快速比对。
- evalue越小,在查找短序列时返回的结果越可靠,与平时一般序列比对时设置的完全不一样,平时是1e-5
- 当设置为blastn-short时,就默认了word_size为7
blastn -query query.fasta -out seq.blast -db /share/nas1/ranjr/project/drop-data/20210709_0711_1A_PAG24791_01a8d3ef/data/database/PAG24791_pass -outfmt 6 -evalue 1e-5 -num_descriptions 10 -num_threads 8
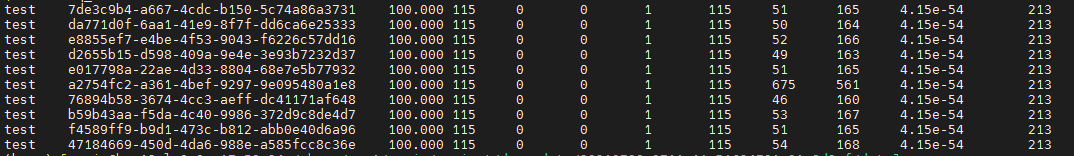
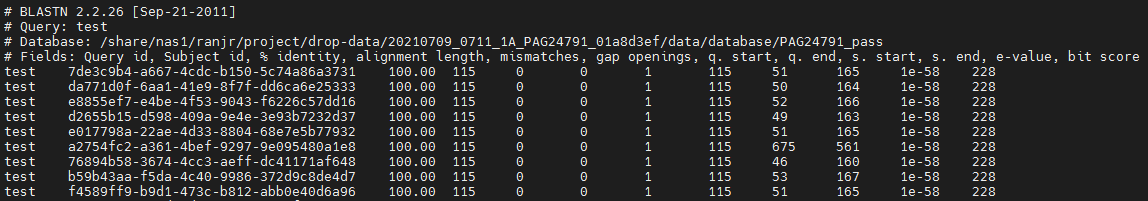
结果文件解释
结果文件一共包含12列。
第1列:查询序列的ID,query.fasta中序列的标识符
第2列:Subject id,比对上的目标序列ID
第3列:% identity,序列比对的一致性百分比
第4列:alignment length,符合比对的比对区域的长度
第5列:mismatches,比对区域的错配数
第6列:gap openings,比对区域的gap数目
第7列:q. start,比对区域在查询序列(Query id)上的起始位点
第8列:q. end,比对区域在查询序列(Query id)上的终止位点
第9列:s. start,比对区域在目标序列(Subject id)上的起始位点
第10列:s. end,比对区域在目标序列(Subject id)上的终止位点
第11列:e-value,比对结果的期望值,将比对序列随机打乱重新组合,和数据库进行比对,如果功能越保守,则该值越低;该E值越高说明比对的高得分值是由GC区域,重复序列导致的。对于判断同源性是非常有意义的几个参数。
第12列:bit score,比对结果的bit score值
参考资料
http://blog.shenwei.me/local-blast-installation/
https://www.jianshu.com/p/de28be1a3bea
blastall
blastall使用说明及常用参数解释
-p: 选择程序, blastn,blastx,blastp,tblastn,tblastx
-i:要查询的文件,query.fa
-d:待搜索的数据库
-o:输出的文件名
-e:期望值,默认10,控制搜索的灵敏度
-m:设置输出文件的格式,有 0-11 种选项,默认是0
0 = pairwise,1 = query-anchored showing identities,
2 = query-anchored no identities,
3 = flat query-anchored, show identities,
4 = flat query-anchored, no identities,
5 = query-anchored no identities and blunt ends,
6 = flat query-anchored, no identities and blunt ends,
7 = XML Blast output,
8 = tabular,
9 tabular with comment lines
10 ASN, text
11 ASN, binary [Integer]
-G:起始空位罚分,默认0
-q:blastn比对时,设定核酸错配的罚分,默认-3
-v:设定输出文件中,匹配列表最多输出多少个subject,默认500
-b:设定输出文件中,最多显示多少个query-subject两两比对的文本描述,默认250
-a:设定搜索过程中cpu数目,默认1
/share/nas2/genome/bin/blastall -p blastn -i db.fa -d /share/nas1/ranjr/project/drop-data/20210709_0711_1A_PAG24791_01a8d3ef/data/database/PAG24791_pass -o seq_blast.result6 -e 1 -q -1 -m 9 -b 886473